14.02.2006
Basisinfo
Gezielte Veränderungen im Pflanzengenom
In situ-Modifikation: Der direkte Zugriff
Warum nicht direkt in die Pflanzen-DNA eingreifen? Kann man nicht die einzelnen DNA-Bausteine neu arrangieren? Eine elegante Strategie - doch es ist leichter gesagt als getan.
Bei einer in situ-Modifikation sollen Gene dort verändert werden, wo sie sich natürlicherweise befinden: im Genom einer Pflanze. Indem DNA-Bausteine (Nukleotide) in ein Gen eingefügt, daraus entfernt oder modifiziert werden, kann die Aktivität des Gens oder des von dem Gen kodierten Genproduktes (Protein) beeinflusst werden. Qualitative oder quantitative Änderungen von Proteinen (Enzymen), die an einem bestimmten Stoffwechselweg beteiligt sind, resultieren dann in geänderten Stoffwechselprodukten, wie z.B. pflanzlichen Kohlenhydraten oder Ölen.
Bei diesem Verfahren bleiben die Veränderungen auf die zu modifizierenden Nukleotide vorhandener Erbanlagen beschränkt. Es werden keine neuen Gene eingefügt und keine Markergene verwendet.
Es gibt verschiedene Ansätze, wie man Gene in situ modifizieren kann. Dazu werden bestimmte DNA-Fragmente (Oligonukleotide) etwa mit einer Mikroinjektion oder durch andere Verfahren in die Pflanzenzelle gebracht. Je nach Typ und Funktionsweise bewirken sie bestimmte Modifikationen am Genom.
Ein SiFo-Projekt beschäftigte sich mit zwei Arten von Oligonukleotiden zur in situ-Modifizierung von pflanzlichen Genen. Aufgrund der spezifischen Bedingungen der pflanzlichen Zelle konnte das bei Tieren funktionierende System jedoch nicht auf die Pflanze übertragen werden.
Triple-Helix-bildende Oligonukleotide
Der Einsatz von Triple-Helix-bildenden Oligonukleotiden (= triple-helix-forming oligonucleotides; TFO’s) wurde bei der Arbeit mit Säugerzellen entwickelt. Mit dieser Methode können die betroffenen Gene ausgeschaltet werden. Dies kann über bestimmte, an das TFO gekoppelte Substanzen geschehen, die den Austausch oder den Verlust einzelner Buchstaben bedingen.
Unter bestimmten Voraussetzungen lagern sich spezielle Oligonukleotide sequenzspezifisch außen in die große Grube der Doppelhelix der DNA ein und bilden so eine Triple Helix – also eine dreifache Helix – aus. Allerdings ist das nur möglich, wenn der zu modifizierende Bereich der Erbanlage nur aus den DNA-Bausteinen A und G besteht (Homopurin-Bereich).
TFOs, die aus den Bausteinen C und T aufgebaut sind, lagern sich parallel, solche aus den Bausteinen A und G lagern sich anti-parallel dem Homopurinstrang der Doppelhelix an.
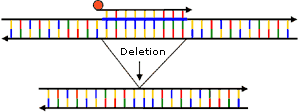
Triple-Helix-bildende Oligonukleotide. Die Oligonukleotide können sich nur da an die DNA-Helix heften, wo diese aus den Buchstaben A (Adenin, blau) und G (Guanin grün) besteht. Ein TFO-Oligonukleotid das aus den Bausteinen C und T aufgebaut ist, lagert sich diesem Bereich sequenzspezifisch und parallel an.
TFO zur Inaktivierung der Genaktivität.Substanzen, die sich in die DNA-Doppelhelix einlagern, können den Verlust von Nukleotiden hervorrufen. Wird das Leseraster der Erbanlagen so unterbrochen, kommt es zu einer Inaktivierung der Gene. Solche Substanzen werden terminal mit den TFOs verbunden. Durch Verwendung von TFOs„ die aus Purinnukleotiden (A, G) oder aus Pyrimidinnukleotiden (C, T) aufgebaut sind, kann gesteuert werden, ob sich das Oligonukleotid parallel oder anti-parallel zum Homopurinbereich
des Gens anlagert und somit auf welcher Seite des Zielbereiches im Gen die
Veränderungen eingefügt werden sollen.
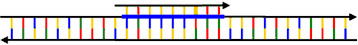
Oligonukleotid mit Basenanalogon. (grün)Diese sind den DNA-Buchstaben ähnlich. Sie binden mit einem anderen Buchstaben als normalerweise. Dadurch heftet sich das Oligonukleotid unter bestimmten Umständen auch an den Buchstaben T (Thymin, gelb) und C (Cytosin, rot) an.
Bi-funktionelle Oligonukleotide
Diese erstmals 1999 in Säugerzellen beschriebenen Oligonukleotide enthalten sowohl einen Triple-Helix-bildenden Bereich als auch eine Sequenz mit der gewünschten Änderung. (Daher der Name bi-funktionell.) Während der Triple-Helix-bildende Bereich durch die Bindung außen an die Doppelhelix als „Anker“ dient, enthält der zweite Bereich des bi-funktionellen Oligonukleotids die Nukleinsäuresequenz mit der gewünschten Veränderung. Dieser Bereich kann bei Öffnung der DNA-Doppelhelix an einen der beiden Stränge binden und so z.B. bei Reparaturprozessen die gewünschte Änderung bewirken. Mit diesem Verfahren können grundsätzlich alle DNA-Abfolgen modifiziert werden, nicht nur solche mit A- und G-Buchstaben.
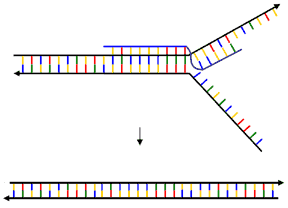
Bi-funktionelles Oligonukleotid.
Thematische Verknüpfungen
Streitfall Markergene

