14.05.2012
Forschung Projekte
Gezielter Einbau von Genen an definierten Positionen im Rapsgenom
(2008 – 2011) Technische Universität Carolo-Wilhelmina zu Braunschweig, Institut für Genetik
Thema
Bei der Übertragung neuer Gene in Pflanzen ist nicht vorhersehbar, an welcher Stelle sie in das Genom eingebaut werden. Dadurch können andere Gene in ihrer Funktion beeinflusst werden. Ein gezielter Einbau an bekannten und gut definierten Stellen im Genom könnte diese Unsicherheit ausschließen.
Ziel dieses Projektes war es, Pflanzen zu entwickeln, die an einem definierten Ort im Genom optimal für die Integration eines Gens vorbereitet sind. Durch den punktgenauen Einbau sollen mögliche Effekte durch eine ungünstige Positionierung der neu eingebrachten Gene auf den Chromosomen ausgeschlossen werden. Außerdem sollen Transgene gezielt in Regionen mit hoher Gen-Expression integriert werden.
Die Forschungsarbeiten wurden an der Kulturpflanze Raps (Brassica napus) durchgeführt. Neben dem Anbau als Öl liefernde Pflanze für die Ernährung, ist Raps ein nachwachsender Rohstoff mit zunehmender Bedeutung für die chemische Industrie und für die Erzeugung von Biodiesel.
Informationen zum Verfahren:
Zusammenfassung
Mit dem untersuchten System sollten transgene Pflanzenlinien hergestellt werden, bei denen der Integrationsort der neuen Gene bekannt ist und der eine hohe Genexpression ermöglicht. Alle dazu erforderlichen Komponenten wurden positiv auf ihre Funktionalität hin untersucht.
In Tabak konnten mit Hilfe des Systems entsprechende transgene Linien erzeugt werden. Für Raps konnten erste Untersuchungen mit dem System begonnen werden, jedoch ist die Transformationsfrequenz hier sehr gering.
Das System ist also prinzipiell unabhängig von der Kulturart und könnte in jeder Pflanzenart eingesetzt werden.
Versuchsbeschreibung
In der Natur gibt es verschiedene Mechanismen zur Übertragung und Neukombination von DNA. Zwei von ihnen wurden in diesem Projekt angewendet: ein Transposonsystem und ein Rekombinationssystem. Transposasen und Rekombinasen sind Enzyme, die an bestimmten Erkennungssequenzen DNA-Abschnitte ausschneiden und an anderer Stelle wieder integrieren können. Im Unterschied zu Transposasen benötigen Rekombinasen auch für den Einbau eines DNA-Abschnittes Erkennungssequenzen. Transposasen können das ausgeschnittene DNA-Stück an beliebiger Stelle wieder einbauen, in der Regel integrieren sie es aber in Regionen mit hoher Genexpression.
Beide Systeme wurden bereits einzeln erfolgreich bei Pflanzen eingesetzt, um Markergene zu entfernen. In diesem Projekt sollten sie erstmals kombiniert und für den gezielten Einbau von Genen genutzt werden. Mit Hilfe von Agrobakterien wurde ein gentechnisch verändertes Transposon in Rapspflanzen eingeschleust, das die Erkennungssequenzen für eine Rekombinase enthält. Darüberhinaus enthielt es ein Reportergen, mit dessen Hilfe sich messen lässt, ob das Transposon in einer Region mit hoher Genexpression eingebaut wurde. Mit molekularbiologischen Methoden wurde untersucht, ob sich durch den Einbau keine unerwünschten Veränderungen im Rapsgenom wie z.B. die Unterbrechung eines Gens ereignet haben. Rapslinien, auf die beides zutraff, wurden als Empfängerlinien ausgewählt.
In das Genom der Empfängerlinien kann mit Hilfe einer Rekombinase jedes gewünschte Transgen zielgerichtet eingebracht werden.
Entfernung des Transposase-Gens mit Hilfe eines negativen Selektionsmarkers
Damit das gentechnisch veränderte Transposon in eine stark exprimierende Position des Rapsgenoms eingebaut werden kann, muss auch ein Gen für eine Transposase in die Pflanzenzelle eingeschleust werden. Das Transposase-Gen soll aber nach Möglichkeit nicht in das Pflanzengenom eingebaut werden, denn dann könnte das Transposon jederzeit unkontrollierbar innerhalb des Genoms „springen“. Deshalb sollte das Transposase-Gen in diesem Projekt mit einem so genannten negativen Markergen gekoppelt werden. Das Nährmedium, auf dem aus transformiertem Blattgewebe Kalluskulturen angezogen wurden, enthält eine Chemikalie, die von den Pflanzen mit dem negativen Markergen zu einer toxischen Substanz umgewandelt wird. So überlebten nur Pflanzen, bei denen das Transposase-Gen nicht in das Genom eingebaut wurde.
Gleichzeitig wurden mit Hilfe eines herkömmlichen, „positiven“ Markers diejenigen Pflanzen selektiert, in deren Genom das Transposon eingebaut wurde.
Ergebnisse
Messung der Effizienz der Transposase
Zunächst sollte in Arabidopsis und in Tabak die Effizienz der verwendeten Transposase untersucht werden.
Es wurden vier verschiedene Gen-Konstrukte erstellt, die eine Transposase aus Mais mit verschiedenen Promotoren in unterschiedlichen Kombinationen enthalten. Diese Konstrukte wurden in T-DNA-Vektoren eingebracht und in sechs Arabidopsis-Linien transformiert. Die Pflanzen wurden anschließend mit einer Arabidopsis-Linie gekreuzt, die die Erkennungssequenzen für die Transposase enthielten (eine so genannte Ds-Linie). Durch das Herausschneiden der Erkennungssequenzen wird ein Markergen aktiviert, das eine Resistenz gegenüber Phosphinothricin vermittelt. Bei den Nachkommen traten jedoch keine Phosphinothricin-resistenten Pflanzen auf. Die Kreuzungen sollen daher mit anderen Ds-Linien wiederholt werden.
Parallel wurden die gleichen Konstrukte auch in Tabak transformiert. Auch hier wurden jeweils sechs Linien etabliert und mit einer Ds-Linie gekreuzt. Die Nachkommen wurden analog zum Arabidopsis-Experiment auf die Effizienz der Transposase hin analysiert. Alle vier untersuchten Transposase-Konstrukte zeigten in Tabakpflanzen eine ausreichend hohe Effizienz.
Überprüfung des negativen Selektionsmarkers
An Tabakpflanzen sollte überprüft werden, ob der negative Selektionsmarker funktioniert. Das Markergen (codA) wurde mittels PCR aus DNA von E. coli vermehrt. Anschließend wurde es in einen T-DNA-Vektor eingebracht und mit Hilfe von Agrobakterien in Tabak transformiert.

Abb. A: Entwicklung von Kallusgewebe einer Tabakpflanze mit codA-Gen auf Medium mit und ohne Selektion (5-FC) im Vergleich zu Kallusgewebe von Wildtyp. Nach 14 Tagen findet auf 5-FC-Medium fast kein Kalluswachstum statt und die Zellen werden braun und sterben ab.
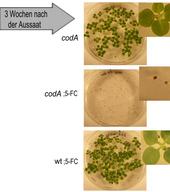
Abb. B: Das codA-Gen zeigt nach Rückkreuzung mit dem Wildtyp auch in den Nachkommen seine Wirkung. Nach 3 Wochen auf Medium mit 5-FC wachsen keine Keimlinge heran.
Es wurden sechs transgene Tabakpflanzen regeneriert, die mittels PCR auf die Anwesenheit des codA-Gens überprüft wurden. Keine der sechs Linien konnte auf dem Selektionsmedium wachsen, während Kallusgewebe von Wildtyp-Tabakpflanzen überlebten. Um die Funktionalität des codA-Gens in den Nachkommen zu überprüfen, wurden die Linien mit dem Wildtyp gekreuzt. Abbildung B zeigt die Entwicklung von Keimlingen nach 3 Wochen auf Medium mit und ohne 5-FC. Prinzipiell funktioniert das System also.
Transformation von Rapspflanzen
Parallel zur Erstellung der Konstrukte wurde eine Agrobakterien-vermittelte Transformationsmethode für Raps etabliert werden. Nach Regeneration und Selektion konnten drei transformierte Rapslinien ins Gewächshaus überführt werden. Zwei Pflanzen wuchsen auch in Erdkultur weiter, bildeten fertile Blüten aus und es konnten Samen geerntet werden.
Demnach funktioniert die Rapstransformation prinzipiell, jedoch ist die Transformationsfrequenz sehr gering.
Transformation von Tabakpflanzen
Aufgrund der geringen Transformationsfrequenz bei Rapspflanzen wurden auch die oben beschriebenen Tabakpflanzen weiter untersucht. So sollten auch bei anderen Kulturarten prinzipielle Aussagen über die Funktionalität des Systems möglich sein.
Dazu wurden für jedes der vier Gen-Konstrukte mehrere Tabakpflanzen auf Selektionsmedium regeneriert. Mit Hilfe von PCR konnte bei mehr als der Hälfte der Pflanzen die Anwesenheit von Ds-Elementen und die Abwesenheit der T-DNA gezeigt werden.
Thematische Verknüpfungen
Themen
Förderung
Bundesministerium für Bildung und Forschung
Förderkennzeichen
0315210F
Projekt
Originaltitel
Transgenintegration in stark exprimierende Positionen beim Raps
Kontakt
Prof. Dr. Reinhard Hehl
Technische Universität Carolo-Wilhelmina zu Braunschweig
Institut für Genetik
38092 Braunschweig
Forschungsprojekte
Verbundprojekt: Optimierung der biologischen Sicherheit gentechnisch veränderter Pflanzen 2008-2011
- Wie zuverlässig ist der Anbau von männlich sterilem Mais zur Begrenzung der Ausbreitung? JKI Quedlinburg
- Verhinderung der Ausbreitung von gentechnisch verändertem Mais durch transgen-freien Pollen, Universität Hamburg
- Plastidentransformation zur Verhinderung der Ausbreitung gentechnisch veränderter Pflanzen, MPI Potsdam
- Entwicklung einer Plastidentransformations- technologie für Mais, MPI Potsdam
- Transgene Zitterpappeln: Überprüfung der Zuverlässigkeit männlicher Sterilitätssysteme, vTI Großhansdorf
- Verhinderung der Ausbreitung von gentechnisch veränderten Pappeln durch transgen-freien Pollen, vTI Großhansdorf
- Gezielter Einbau von Genen an definierten Positionen im Rapsgenom, TU Braunschweig
- Entwicklung einer Technik zur zielgerichteten Integration von Genen bei Pflanzen, Universität Karlsruhe
