25.09.2005
Forschung Projekte
Optimierung von Vektoren für die Erzeugung Markergen-freier Pflanzen
(2001 – 2004) Bundesanstalt für Züchtungsforschung an Kulturpflanzen (BAZ) (seit 2008 Julius Kühn-Institut (JKI)), Institut für Rebenzüchtung Geilweilerhof, Siebeldingen
Thema
Bei der Agrobakterien-vermittelten Transformation von Pflanzen werden so genannte binäre Vektoren verwendet. Diese sollten dahingehend optimiert werden, dass nur gewünschte DNA-Sequenzen auf die Pflanze übertragen werden.
Die dabei verfolgten Ziele waren:
- Es sollten geeignete binäre Vektoren entwickelt werden, die für die Cotransformation zweier unabhängiger Gene verwendet werden können
- Es sollte nur die gewünschte T-DNA integriert werden. (Erhöhung der präziseren Integration - Terminationsgenauigkeit - der T-DNA an der linken Border.) Damit könnte das Problem der „undichten“ linken Border überwunden werden. Seit längerem ist bekannt, dass der Transfer und die Integration der T-DNA bei der RB (rechte Border) beginnt und bei der LB (linke Border) endet. Allerdings scheint die LB nicht „dicht“ zu sein, da es häufig vorkommt, dass auch Sequenzen außerhalb der T-DNA mit übertragen werden.
- Es sollten binäre Vektoren verkleinert werden. Dies ist notwendig, um auch große T-DNA-Abschnitte - evtl. mit mehreren Genen - übertragen zu können.
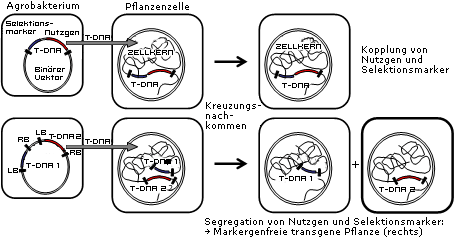
Prinzip der Herstellung markergenfreier Pflanzen durch Cotransformation. Liegen Nutzgen (rot) und Selektionsmarker (blau) auf einer T-DNA, so sind diese beiden Gene auch in den Nachkommen gekoppelt (obere Reihe). Werden Nutzgen und Selektionsmarker aber separat durch jeweils eine eigene T-DNA übertragen und integrieren sie unabhängig voneinander im Pflanzengenom, können sie in den Kreuzungsnachkommen segregieren, so dass markergenfreie Pflanzen entstehen (untere Reihe).
Informationen zu den Verfahren:
Zusammenfassung
Es hat sich gezeigt, dass die Cotransformation mit einem Vektor und zwei unabhängigen T-DNAs den meisten Erfolg verspricht. Ob in den Folgegenerationen Pflanzen, die nur noch das Nutzgen tragen, auftreten, konnte in der Projektlaufzeit nicht mehr geklärt werden.
Es wurden Vektoren entwickelt, mit denen eine präzisere Integration der T-DNA ermöglicht werden soll.
Es konnte ein deutlich kleinerer Vektor konstruiert werden (kleiner 5 kb). An dem passenden Agrobakterienstamm wird noch gearbeitet.
Versuchsbeschreibung
Zunächst wurden neue binäre Vektoren entwickelt, die geeignet erschienen, die Zielvorgaben zu erfüllen.
- Diese Vektoren wurden durch Agrobakterien-vermittelte Transformation an der Modellpflanze Arabidopsis getestet.
- Anschließend wurde die Funktionsweise der Vektoren überprüft. Dazu wurden die transformierten Pflanzen molekularbiologisch untersucht (PCR, Southern-Hybridisierung). Außerdem wurden mit ihnen verschiedene Kreuzungen und Selbstbefruchtungen durchgeführt (Segregationsanalyse).
In Zusammenarbeit mit einem anderen Teilprojekt des Forschungsverbundes wurden Vektoren entwickelt, die auch bei einkeimblättrigen Pflanzen (Monokotyle) wie der Gerste anwendbar sind.
Ergebnisse
Die erste Generation von Vektoren, die für eine Cotransformation zweier Gene geeignet sind, wird derzeit getestet.
- Es hat sich gezeigt, dass die Cotransformation mit einem Vektor und zwei unabhängigen T-DNAs den meisten Erfolg verspricht. Die Vektoren enthalten zwei unabhängige T-DNAs, die jeweils mit einer eigenen Start- und Stopp- Sequenz (right border und left border) versehen sind. Dazwischen liegt ein DNA-Stück als „Abstandhalter“ (spacer), der dafür sorgen soll, dass die T-DNAs nicht an der gleichen Stelle, sondern unabhängig voneinander im Genom integriert werden.
- Es wurden 33 transformierte Arabidopsis Pflanzen mittels PCR-Analyse auf das Vorhandensein der beiden durch den Spacer getrennten T-DNAs untersucht. Bei allen untersuchten Pflanzen ließ sich die T-DNA mit dem Selektionsmarker und bei elf Pflanzen auch die zweite T-DNA mit dem „Nutzgen“ nachweisen. Es wurde kein Spacer gefunden, was bedeutet, dass die T-DNAs unabhängig von einander integriert wurden. Ob sich bei der nun folgenden Vermehrung Pflanzen selektionieren lassen, die nur noch das Nutzgen tragen und nicht mehr das Selektionsgen (Segregationsanalyse), konnte innerhalb des Projektes nicht abschließend untersucht werden.
Es wurden Vektoren entwickelt, mit denen eine präzisere Integration der T-DNA ermöglicht werden soll. Hierbei wurde die „linke Grenze“ (left border), die Terminationssequenz, doppelt oder gar vierfach eingesetzt. Es wurden bisher 30 transformierte Arabidopsis Pflanzen untersucht. Nur bei einer der Pflanzen wurden DNA-Sequenzen in die transgene Pflanze übertragen, die außerhalb der linken Border lagen. Segregationsanalysen zur statistischen Absicherung konnten bis zum Projektende nicht mehr durchgeführt werden.
Im Versuchsteil zur Verkleinerung der Vektorgröße konnte der Vektor in etwa halbiert werden (auf unter 5 kb: 4,8 kb mit Selektionsmarker, 3,3 kb ohne). Es konnten Sequenzen, die nicht unbedingt auf dem Plasmid lokalisiert sein müssen, entfernt werden. Die Information der so genannten Partitionsgene muss dann statt auf dem Plasmid im Genom des Agrobakteriums vorhanden sein. Die Integration dieser Gene in das Genom des Agrobakteriums erwies sich jedoch als relativ schwierig und konnte daher nicht in der Projektlaufzeit abgeschlossen werden.
Thematische Verknüpfungen
Themen
Förderung
Bundesministerium für Bildung und Forschung
Förderkennzeichen
0312627 B
Projekt
Originaltitel
Optimierte binärer Vektoren für die Herstellung transgener Pflanzen ohne unerwünschte Sequenzen
Kontakt
Dr. Ludger Hausmann
Bundesanstalt für Züchtungsforschung an Kulturpflanzen (BAZ)
(seit 2008 Julius Kühn-Institut (JKI))
Institut für Rebenzüchtung
Geilweilerhof, 76833 Siebeldingen
Tel: 06345 41-117
Fax: 06345 919050
Veröffentlichungen
Forschungsprojekte
Neue Methoden der Gen-Übertragung 2001-2004
- Gezielter Einbau von Genen, Universität Karlsruhe
- Entwicklung alternativer Marker, SunGene
- Ausschneiden unerwünschter Gene bei der Zuckerrübe, Planta
- Ausschneiden unerwünschter Gene: Cre/lox-System, BBA Braunschweig
- Ausschneiden unerwünschter Gene mit Hilfe eines neuen Rekombinationssystems, Bayr. LA Landwirtschaft, Freising
- Negative Marker, Uni Rostock
- Mikroinjektion bei Pflanzen, Uni Giessen
- Gene für die Mikroinjektion, Bioplant
- Geeignete Pflanzenzellen für die Mikroinjektion, Bioplant
- Mikroinjektion, FI Schmallenberg
- Direkte Veränderungen von Pflanzengenen, BBA Braunschweig
- Sicherheitssystem zur Proteinproduktion mittels pflanzlicher Viren, BBA Braunschweig
- Verbesserte binäre Vektoren, BfZ Siebeldingen
- Markergen-freie Pflanzen durch Auskreuzen, MPIZ Köln
- Transformation bei Plastiden, SunGene
