CRISPR/Cas: Baukasten zur Editierung von Genomen wird erweitert
Basen-Editierung ermöglicht jetzt Korrektur von Punktmutationen in DNA und RNA

Die Bausteine der Erbinformationen sind Nukleotide. Ein Nukleotid besteht aus drei Bestandteilen: einer Phosphorsäure, einem Einfachzucker und einer von fünf Nukleinbasen (A, G, C, T oder U). (Bildquelle: © MIKI Yoshihito/flickr/CC BY 2.0)
Mit einer Weiterentwicklung der CRISPR/Cas-Technik können einzelne Basen in DNA oder RNA ausgetauscht werden. Dadurch lassen sich Punktmutationen im Genom präzise korrigieren. In der Petrischale haben die Forscher damit bereits krankheitsauslösende Mutationen beim Menschen geheilt. Bis zur klinischen Anwendung wird es noch eine Weile dauern, doch für die Grundlagenforschung sind diese Werkzeuge bereits jetzt Gold wert. Und auch für die Pflanzenzüchtung ergeben sich daraus große Potenziale.
Vor fünf Jahren tauchte CRISPR zum ersten Mal auf der Bildfläche auf. Damals wurde die Technologie, mit der sich doppelsträngige DNA gezielt schneiden lässt, als präzises Werkzeug gefeiert. Doch wirkt das alte CRISPR/Cas-System jetzt geradezu plump und grobschlächtig im Vergleich mit zwei neuen Methoden, die vor kurzem in den Journals Nature und Science vorgestellt wurden.
Die Rede ist von Basen-Editierung in sowohl DNA als auch RNA. Das Prinzip dahinter: Anstatt die Erbgut-Stränge durchzuschneiden, kommen spezielle Enzyme zum Einsatz, die die Basen chemisch verändern.
Nicht besser, sondern anders
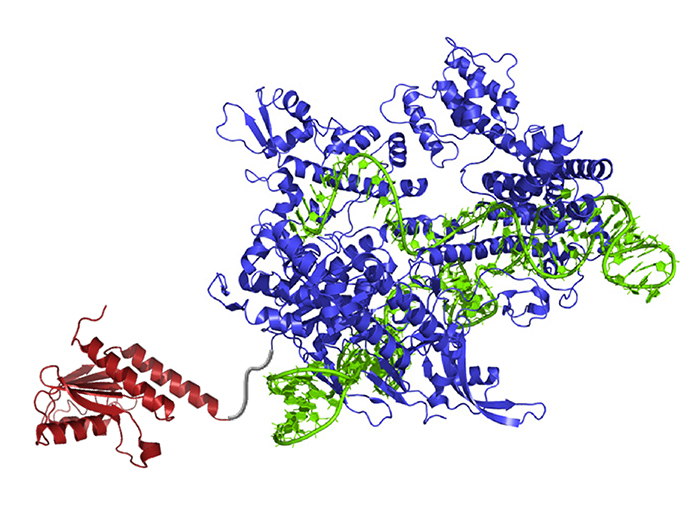
Der neue DNA-Basen-Editor enthält ein Enzym, dass Moleküle umbauen kann und somit Adenin in Inosin (von der Zelle als Guanin erkannt) verwandelt (rot). Die guideRNA (grün) führt das Molekül an den richtigen Ort, Cas9 (blau) schneidet den gegenüberliegenden DNA-Strang und veranlasst die Zelle dazu, die gegenüberliegende Base auszutauschen.
Bildquelle: © Gaudelli et al./ Nature 2017
„Man sollte diese Basen-Editoren nicht als dem alten CRISPR/Cas-System überlegen betrachten, sie sind einfach anders“, sagte der Chemiker David Liu von der Harvard-Universität, der maßgeblich an der DNA-Studie beteiligt war, gegenüber dem Science Magazin. Anders heißt in diesem Fall, dass einzelne Basen gezielter verändert werden können. Das eröffnet die Möglichkeit für die Humanmedizin, Punktmutationen und damit verbundene Krankheitsbilder besser zu korrigieren.
Unsere DNA besteht aus vier Basen, von denen immer zwei ein Paar bilden: Adenin und Thymin (A×T) sowie Guanin und Cytosin (G×C). Bereits 2016 hatte David Liu gezeigt, dass sich mit einer modifizierten Form der CRISPR-Technik das Basenpaar G×C zu A×T verändern lässt. Jetzt legen die Forscher nach und präsentieren das Werkzeug für die Veränderung in entgegengesetzte Richtung: von A×T zu G×C.
Mit gerichtete Evolution zum benötigten Enzym
Ihre größte Herausforderung dabei war, dass sie ein Enzym brauchten, was in der Natur nicht existiert oder zumindest bisher nicht entdeckt wurde. Die Wissenschaftler versuchten sich daher an einer seit Jahren etablierten Methode zur Proteinveränderung, der „gerichteten Evolution“.
Ihr Ausgangspunkt war das Enzym TadA aus dem Bakterium E. coli. TadA kann Adenin in Inosin (wird wie Guanin gelesen) verwandeln, allerdings nur in DNA. Indem die Wissenschaftler Bakterien starkem Selektionsdruck aussetzten, gelang es ihnen, eine modifizierte Form von TadA zu generieren. Dieses Enzym namens ABE7.10 kann auch in RNA ein A in ein I verwandeln.
Die weiteren Zutaten für die Basen-Editierung sind eine guideRNA (gRNA) gekoppelt an ein defektes Cas9 (dCas9), welches die doppelsträngige DNA zwar noch aufspalten, aber nicht mehr komplett durchschneiden kann. Die gRNA bindet an die richtige Stelle der DNA, das Cas9 schneidet in einen der beiden DNA-Stränge hinein und regt die Zelle somit zur Reparatur dieses Strangs an.
Weil aber ABE7.10 im gegenüberliegenden Strang ein A in ein I (G) verwandelt hat, baut die Zelle beim Zusammenflicken des Strangs anstelle des T ein C ein. Das Inosin selbst wird durch zelleigene Reparaturmechanismen oder während der Verdoppelung der DNA in Guanin umgewandelt. Die Änderung von A×T zu G×C ist geglückt.
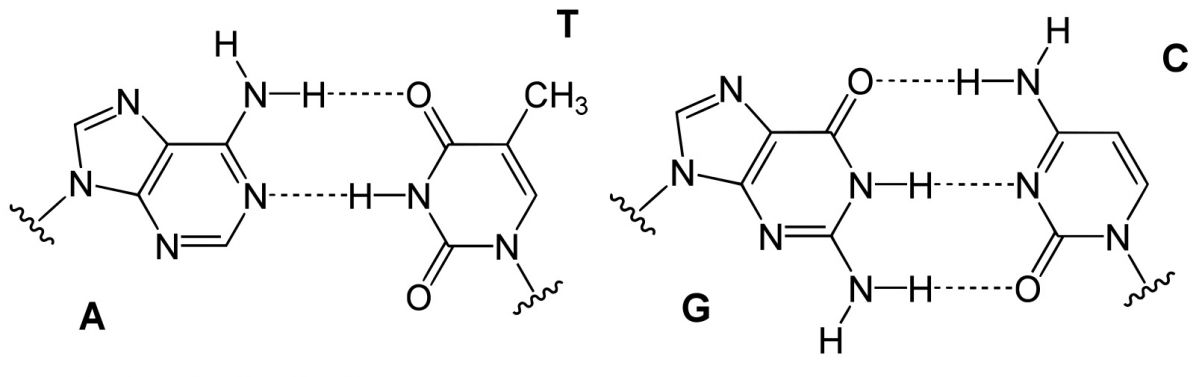
Basenpaare der DNA:
A-T (Adenin und Thymin) und G-C (Guanin und Cytosin) - die Nukleobasen werden durch Wasserstoffbrücken zusammengehalten (hier als gepunktete Linien dargestellt).
Bildquelle: © Yikrazuul/ wikimedia.org/ gemeinfrei
„Das außergewöhnliche hieran ist, dass es den Forschern gelungen ist, das TadA-Enzym dazu zu bringen, etwas ziemlich Unnatürliches zu tun“, kommentierte CRISPR-Forscher George Church von der Harvard-Universität die Methode gegenüber Science.
Veränderungen an der RNA sind nicht von Dauer
In einer zweiten Studie gingen Wissenschaftler einen anderen Weg. Wie bereits auf Pflanzenforschung.de beschrieben („C2c2 ergänzt Cas9 auf RNA-Ebene“), gibt es auch Werkzeuge für die RNA-Ebene. Damit wird es möglich, einzelne Basen der RNA - das Zwischenglied von der DNA-Information zu den Proteinen - umzuschreiben. Das dazu notwendige Werkzeug heißt REPAIR. REPAIR ist ein Akronym für RNA Editing for Programmable A-to-I Replacement. Wissenschaftler um Feng Zhang vom Broad Institute sind bereits vergangenes Jahr auf das Enzym Cas13 (früher C2c2) gestoßen, dass RNA statt DNA schneidet.
Sie koppelten eine katalytisch inaktive Form namens C13b mit einem Enzym, dass die RNA-Base A in I umwandeln kann. Das I wird auch hier als G gelesen, der Basenaustausch ist perfekt. Den Forschern gelang es bereits, mit REPAIR schädliche Mutationen auf der RNA-Ebene zu korrigieren. Dies haben die Wissenschaftler bei menschlichen Zellen in einer Kulturschale zeigen können. Die Zellen stammten von Patienten, die an der Fanconi-Anämie leiden. Diese Erbkrankheit führt zu Missbildungen und Blutarmut.
Wichtiges Werkzeug für die Grundlagenforschung
Diese RNA-Editing-Methode hat eine Reihe von Vorteilen und Anwendungsbereichen. RNA wird bekanntlich von den Zellen ständig neu synthetisiert und relativ schnell wieder abgebaut. Falls bei der Reparatur ein Fehler passiert ist, ist das nicht weiter tragisch: der Fehler verschwindet. In der Medizin könnte mit dem RNA-Editing auch auf die ethisch problematischen Keimbahntherapien verzichtet werden, die eine dauerhafte Veränderung des Individuums und seiner Nachkommen bedeuten. Gleiches gilt für andere Organismen wie Pflanzen.
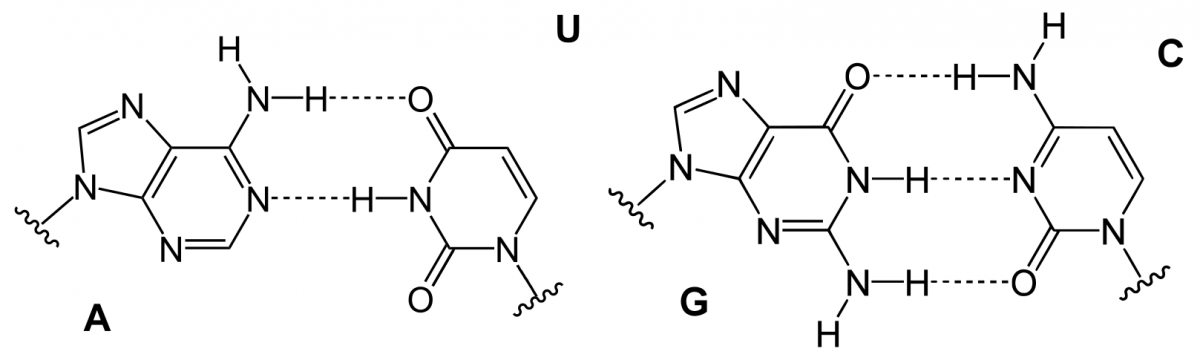
Basenpaare der RNA:
A-U (Adenin und Uracil) und G-C (Guanin und Cytosin)
Bildquelle: © Yikrazuul/ wikimedia.org/ gemeinfrei
RNA-Editing mit REPAIR wäre damit auch hervorragend für zeitlich begrenzte Eingriffe geeignet, etwa die Abschaltung der natürlichen Abstoßungsreaktion des Körpers kurz nach einer Organtransplantation. Bei Pflanzen könnte über diesen Mechanismus die Aktivierung eines Abwehrstoffes in dem Moment eingeleitet werden, wenn ein Schädling in eine Pflanzenzelle eindringt.
Dies sind natürlich noch alles Spekulationen und es ist noch viel Arbeit nötig, um die REPAIR-Methode ausreichend präzise zu machen. Im Moment ist das Enzym Cas13b noch zu ungenau. Jedoch ist durch die Publikation eine Tür aufgestoßen worden und Forscher weltweit werden sich daran machen, die Methode weiter zu optimieren. In jedem Fall wird REPAIR die Grundlagenforschung beflügeln. Durch das gezielte Einfügen von Punktmutationen lassen sich post-transkriptionelle Veränderungen wie Spleißmechanismen, RNA-Editierung oder auch die Translation besser untersuchen.
Quellen:
- Gaudelli, N. M. et al. (2017): Programmable base editing of A×T to G×C in genomic DNA without DNA cleavage. In: Nature, (25. Oktober 2017), doi: 10.1038/nature24644.
- Cox, D.B.T., et al. (2017): RNA editing with CRISPR-Cas13. In: Science, (25. Oktober 2017), doi: 10.1126/science.aaq0180.
- Shimatani, Z. et al. (2017): Targeted base editing in rice and tomato using a CRISPR-Cas9 cytidine deaminase fusion. In: Nature Biotechnology, (21. Februar 2017), doi: 10.1038/nbt.3833.
Zum Weiterlesen:
- Nach getaner Arbeit abschalten – Ein Inhibitor erhöht die Spezifität der Genschere CRISPR/Cas9
- Effiziente neue Genschere – Die Genschere CRISPR/Cpf1 arbeitet präziser als das bisherige Standardwerkzeug CRISPR/Cas9
- Genomeditierung noch präziser – CRISPR ohne Schnitt
Titelbild: Die Bausteine der Erbinformationen sind Nukleotide. Ein Nukleotid besteht aus drei Bestandteilen: einer Phosphorsäure, einem Einfachzucker und einer von fünf Nukleinbasen (A, G, C, T oder U). (Bildquelle: © MIKI Yoshihito/flickr/CC BY 2.0)
