Klassische Sequenzierungsmethoden im Überblick
DNA-Proben für die Analyse (Quelle: © iStockphoto.com/ nikesidoroff)
Die DNA-Sequenzierung hat die biologischen Wissenschaften revolutioniert und die Ära der Genomik eingeleitet. Heute gibt es viele Verfahren zum Ablesen der Sequeninformationen. Der folgende Überblick stellt die bekanntestens klassichen Sequenzierungsmethoden vor.
Kettenabbruchsynthese nach Sanger
Die sogenannte Kettenabbruchsynthese, die 1975 vom britischen Biochemiker Frederick Sanger erfunden wurde, bildet auch heute noch häufig die Grundlage moderner Sequenzierungstechniken. Zusammen mit Gilbert erhielt Sanger dafür im Jahr 1980 den Nobelpreis für Chemie.
Seine Methode beruht hauptsächlich auf einer enzymatischen Reaktion. Das zu sequenzierende, doppelsträngige DNA-Molekül (Doppelhelix) wird zunächst durch Hitze in die zwei Einzelstränge gespalten (denaturiert). Ausgehend von einem kurzen Abschnitt bekannter Sequenz, dem so genannten Primer, der sich an einen Einzelstrang anlagert und so ein kurzes Stück Doppelstrang bildet, verlängert das Enzym DNA-Polymerase den fehlenden der beiden komplementären DNA-Stränge.
In vier sonst gleichen Ansätzen (alle beinhalten die vier Nukleotide) wird je eine der vier Basen zum Teil als Didesoxynukleosidtriphosphat (ddNTP) zugegeben. Diese Kettenabbruch-ddNTPs besitzen keine 3’-Hydroxylgruppe, die für die Verknüpfung mit dem nächsten Nukleotid unbedingt notwendig ist. Es kommt an dieser Stelle im neusynthetisierten Strang zu einem Kettenabbruch. In der Folge entstehen DNA-Fragmente unterschiedlicher Länge, die in jedem separaten Ansatz stets mit dem gleichen ddNTP enden. Entweder der Primer oder die ddNTPs sind radioaktiv markiert.
Nach der Sequenzierung werden alle Fragmente mittels Gel-Elektrophorese der Länge nach aufgetrennt. Durch Vergleich der vier Ansätze kann die Sequenz nach der Entwicklung des radioaktiven Gels auf einem fotografischen Film abgelesen werden.
Seit Anfang der neunziger Jahre werden die radioaktiven Substanzen durch Fluoreszenz-Farbstoffe ersetzt. Jedes der vier ddNTPs wird dabei mit einem unterschiedlichen Farbstoff gekoppelt. Diese Modifikation erlaubt es, alle vier ddNTPs in einem Reaktionsgefäß zuzugeben. Mittels Kapillarelektrophorese werden die entstandenen Kettenabbruchprodukte aufgetrennt und durch einen Laser zur Fluoreszenz angeregt. Die Fluoreszenfarbstoff-gekoppelten ddNTPs am Ende jedes DNA-Fragmentes zeigen dadurch Fluoreszenz unterschiedlicher Farbe und können so von einem Detektor erkannt werden. Das Chromatogramm (die Abfolge der Farbsignale, die am Detektor erscheinen) gibt direkt die Sequenz der Basen des sequenzierten DNA-Stranges wieder.
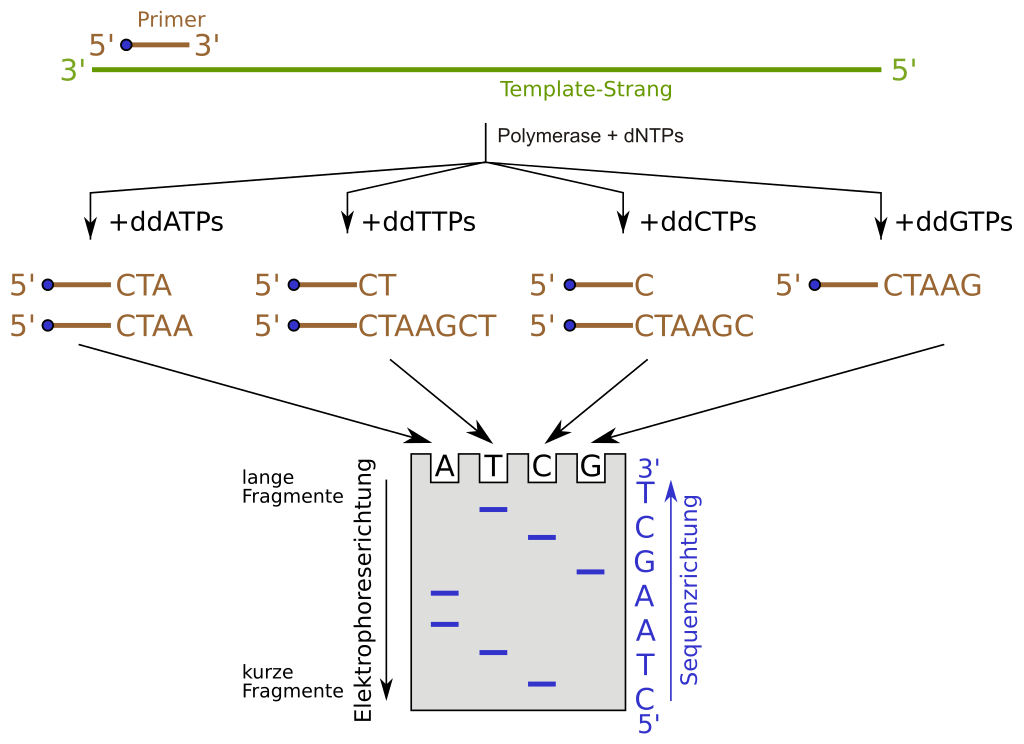
Prinzip der DNA-Sequenzierung nach der Didesoxy-Methode.
Bildquelle: Urheber: © Christoph Goemans, Wikimedia.org; CC BY-SA 3.0
Sequenzierung nach Maxam und Gilbert
Im Jahr 1977 revolutionierten die beiden Chemiker Allan Maxam und Walter Gilbert die Molekularbiologie, indem sie durch spezifische chemische Spaltung die Basenabfolge (Sequenz) eines DNA Moleküls entschlüsselten („sequenzierten“). Die gespaltene DNA wurde am 5’ oder 3’-Ende mit radioaktivem Phosphat markiert. In vier getrennten Ansätzen entfernten die beiden Wissenschaftler jeweils einen bestimmten Typ der vier vertretenen Basen vom Zucker-Phophat-Rückgrat der DNA. Danach spalteten sie den dann basenlosen DNA-Strang an der basenlosen Stelle.
In jedem der vier Ansätze waren nun Fragmente unterschiedlicher Länge entstanden, deren 3'-Ende stets an einer bestimmten Base gespalten worden war. Mit einer hochauflösenden Gel-Elektrophorese trennten sie die Fragmente mit jeweils einer Base Längenunterschied auf. Durch den Vergleich der vier Ansätze auf dem Elektrophoresegel konnten sie die DNA-Sequenz ablesen.
Aufgrund der geringen Eignung zur Automatisierbarkeit und den gesundheitsschädlichen (radioaktiven) Reagenzien kommt diese Sequenzierungsmethode heute kaum noch zum Einsatz.
Die Pyrosequenzierung
1996 entwickelten Pal Nyren und Mostafa Ronaghi aus Schweden die Pyrosequenzierung, ein Hochdurchsatzverfahren, mit dem sich die DNA viel schneller und billiger sequenzieren lässt als mit der Methode nach Sanger. Beim Pyrosequenzing wird die DNA-Polymerase durch ein ausgeklügeltes Enzymsystem unter Beteiligung einer Luziferase dabei beobachtet, wie sie ein Nukleotid nach dem anderen an den neusynthetisierten DNA-Strang anhängt.
Die zu sequenzierende DNA dient als Matrize und liegt auch hier einzelsträngig vor. Analog zur Sanger-Sequenzierung verlängert die DNA-Polymerase von einem Primer ausgehend den komplementären DNA-Strang Nukleotid um Nukleotid. Der Einbau eines NTPs wird durch die Luziferase in einen Lichtblitz umgesetzt, der von einem Detektor erkannt und aufgezeichnet wird. Es wird nun nacheinander immer eine der vier Nukleotidsorten zum Ansatz hinzu gegeben. Bei Zugabe des passenden (komplementären) Nukleotids erhält man ein Signal; wurde ein an dieser Stelle ein nicht passendes NTP zugegeben, bleibt der Lichtblitz aus. Danach werden die vorhandenen NTPs zerstört und eine andere Art wird zugesetzt, dieser Vorgang wird fortgesetzt, bis sich wieder eine Reaktion zeigt.
Die enzymatische Reaktion
Baut die DNA-Polymerase ein komplementäres Nukleodtid erfolgreich ein, wird Pyrophosphat (PPi) freigesetzt. Das Enzym ATP-Sulfurylase wandelt dieses zu Adenosintriphosphat (ATP) um. ATP wiederum treibt die Luziferase-Reaktion an, wodurch Luziferin in Oxyluziferin verwandelt wird. Dies resultiert wiederum in einem detektierbaren Lichtsignal, dessen Stärke proportional zum verbrauchten ATP ist. Eine Anschauliche Präsentation des Pyrosequenzing finden Sie hier.
Vorteil und Einsatz der Pyrosequenzierung
Ein großer Vorteil der Pyrosequenzierung ist ihre gute Automatisierbarkeit, die sich zur hochparallelen Analyse von DNA-Proben eignet. Auch zur Bestimmung von Einzel-Nukleotid-Unterschieden (Single Nucleotide Polymorphisms = SNPs), die Hinweise z.B. auf Erbkrankheiten geben können, wird die Pyrosequenzierung aufgrund ihrer Lesegenauigkeit häufig eingesetzt.
Zum Weiterlesen:
- DNA-Sequenzierungsmethoden - Motoren der Genomforschung
- Ultra-Hochdurchsatz-Sequenziermethoden
- Direkte RNA-Sequenziermethoden
- Ein Interview mit Dr. Kerstin Stangier, Director Business Development, der GATC Biotech AG über maßgeschneiderte DNA-Sequenzierung finden Sie hier: Maßgeschneiderte DNA-Sequenzierung Interview mit Dr. Kerstin Stangier
