Nach getaner Arbeit abschalten
Ein Inhibitor erhöht die Spezifität der Genschere CRISPR/Cas9
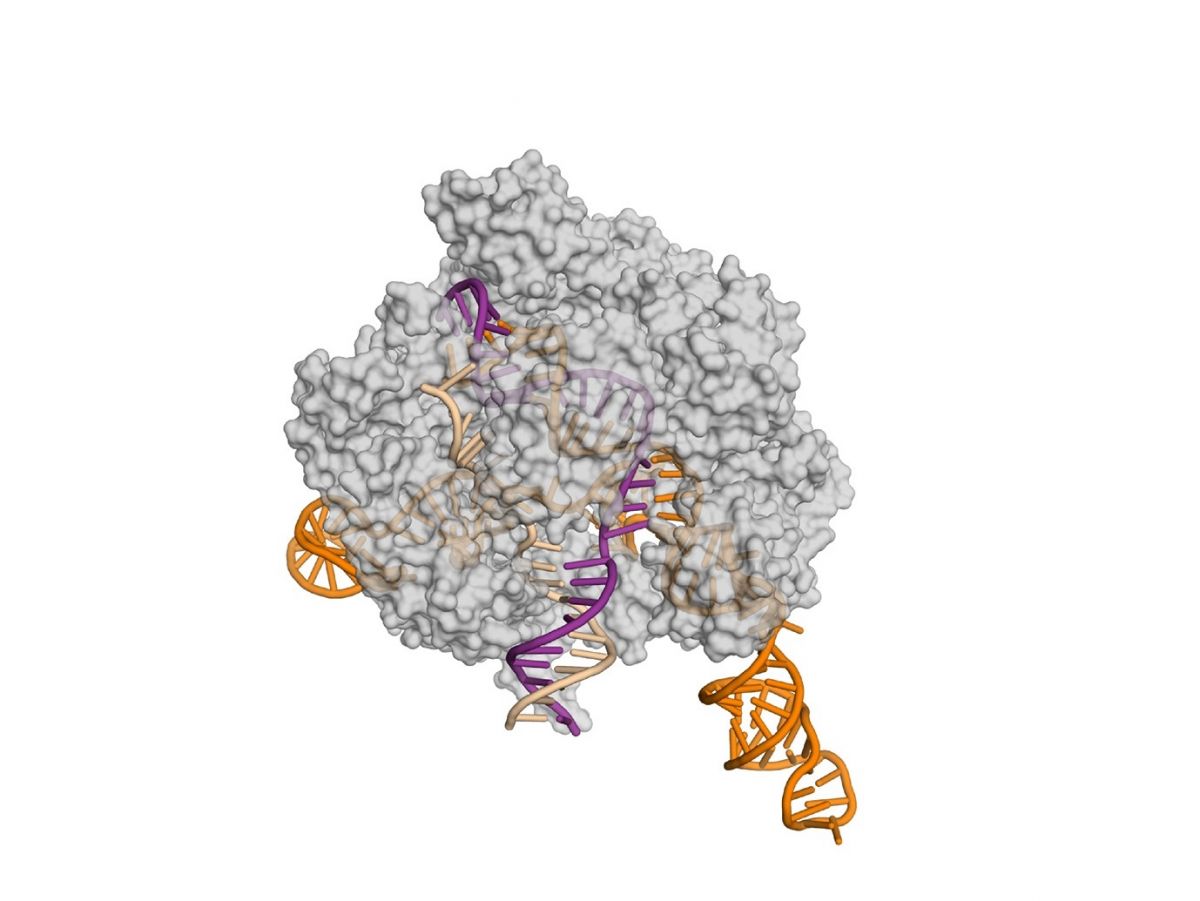
Cas9 (grau) und hat ein spezifisches DNA-Stück (lila) erreicht, an das es andockt, bevor es zur Tat schreitet. Zu diesem DNA-Stück wird es durch die Guide-RNA (orange) geleitet. (Bildquelle: © Fuguo Jiang, UC Berkeley)
Die Genschere CRISPR/Cas9 ermöglicht präzise Veränderungen am Erbgut. Allerdings erlaubt sich die Genschere auch Fehler und schneidet an unerwünschten Stellen. Jetzt haben Wissenschaftler gezeigt, dass ein virales Protein die Genauigkeit der Genschere steigert, indem es off-target-Effekte verhindert.
Keine andere Entdeckung hat die Molekularbiologie in den letzten Jahren so fundamental verändert wie die Genschere CRISPR/Cas9. Das Werkzeug, mit dem sich Bakterien gegen Viren verteidigen, wird inzwischen in Labors auf der ganzen Welt dazu genutzt, pflanzliches oder tierisches Erbgut gezielt zu verändern.
Problematisch ist bisher jedoch die Tatsache, dass CRISPR/Cas9 nicht ganz so präzise arbeitet, wie die Wissenschaftler es sich wünschen. Sogenannte off-target-Effekte, also unerwünschte Punktmutationen im Erbgut, schmälern die Erfolge. Die Forscher vermuteten, dass diese off-target-Effekte erst mit zeitlicher Verzögerung auftreten. Falls es gelingen sollte, CRISPR/Cas9 nach getaner Arbeit vollkommen auszuschalten, könnten sich Fehler vermeiden lassen.
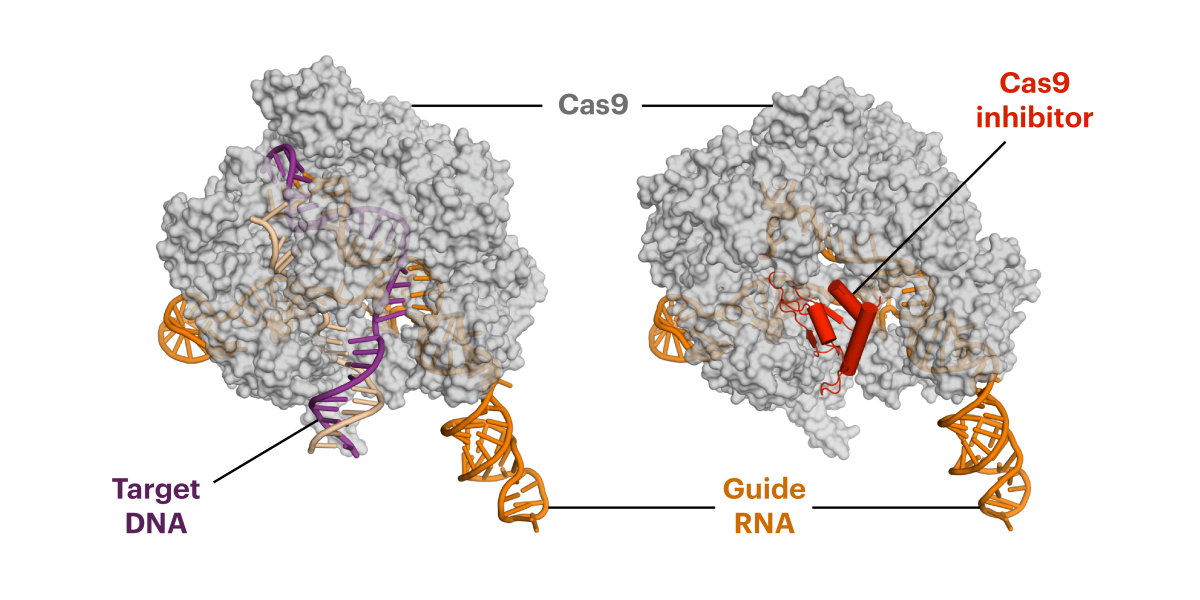
Das Anti-CRISPR-Protein (rot auf der rechten Seite) imitiert die DNA, wo das schneidende Enzym Cas9 typischerweise andockt ("Ziel-DNA"; links) bevor es schneidet. Aber das Anti-CRISPR-Protein lässt nicht los, was Cas9 davon abhält weiter aktiv zu sein.
Bildquelle: © Fuguo Jiang, UC Berkeley
Inhibitor-Proteine verfeinern Wirkung von CRISPR/Cas9
Jetzt haben Wissenschaftler einen neuen Ansatz entdeckt, mit dem sich solche Fehler wohl tatsächlich effektiv verhindert lassen. Im Mittelpunkt stehen dabei Inhibitoren, auch anti-CRISPR-Proteine genannt. Sie wurden erst Ende 2016 entdeckt. Sie stammen aus Bakteriophagen und dienen als Verteidigung gegen die bakterielle CRISPR-Maschine.
Aufnahmen im Kryo-Elektronenmikroskop zeigen: Diese Inhibitoren interagieren mit dem Cas9-sgRNA-Komplex und besetzen genau die Bindestelle, die eigentlich für die DNA reserviert ist.
Dadurch kann Cas9 DNA weder binden, noch schneiden. Durch das richtige Timing gelingt es, dass die Genschere die gewünschten Veränderungen vornimmt, während off-target-Effekte unterbunden werden.
Richtiges Timing ist wichtig
In ihrem Experiment untersuchte das Forscherteam die Wirkung des Inhibitor-Proteins AcrIIA4 am Beispiel menschlicher Blutzellen. Es stammt aus Viren, die Bakterien der Gattung Listerien infizieren, sogenannten Listerien-Bakteriophagen. Zwei Genscheren kamen zum Einsatz. Eine war auf das Schneiden des Gens HBB programmiert, das bei Sichelzellanämie eine Rolle spielt, die andere auf das Zerstückeln von VEGFA, einem endothelialen Wachstumsfaktor.
Gaben sie zuerst AcrIIA4 zu den Zellen und dann CRISPR/Cas9, so wurde die Wirkung der Genschere komplett blockiert. Wurden beide Komponenten gleichzeitig in die Zellen injiziert, war die Genschere auch nicht mehr aktiv. Doch als sie zuerst CRISPR/Cas9 und sechs Stunden später AcrIIA4 hinzugaben, führte CRISPR die erwünschten Veränderungen an der DNA aus. Die Nebenwirkungen jedoch wurden unterbunden.
Off-target-Effekte langsamer?
Ein möglicher Erklärungsansatz ist, dass CRISPR/Cas9 am schnellsten die Schnitte ausführt, auf die es programmiert wurde. Erst später „schnippelt“ es auch an anderen Stellen im Genom mit etwas abweichender Basensequenz herum. Es könnte jedoch auch sein, dass die off-target-Effekte einfach langsamer ablaufen.

Kürzlich ist es Pflanzenforschern aus Kiel gelungen, Gene des tetraploiden Raps mit Hilfe von CRISPR/Cas9 gezielt zu verbessern. Hier zu sehen ist die Regeneration von Rapssprossen in der Petrischale nach Crispr/Cas Mutagenese.
Bildquelle: © Janina Braatz
Doch ganz gleich, welcher Erklärungsansatz stimmt. Mit Inhibitor-Proteinen lässt sich das Problem der off-target-Effekte beheben.
Die Experimente fanden unter Federführung von Jennifer Doudna statt, einer der CRISPR-Entdeckerinnen. Doudna beschäftigt sich vor allem mit der Anwendung der Methode in tierischen Zellen. Doch auch Pflanzenforscher und -züchter sind an effizienteren Genscheren interessiert. Schließlich eröffnet CRISPR auch ihnen bisher ungeahnte Möglichkeiten.
CRISPR kann Ernteverluste bei Raps verringern
Erst kürzlich ist es Pflanzenforschern aus Kiel gelungen, Gene des tetraploiden Raps (Brassica napus) mit Hilfe von CRISPR/Cas9 gezielt zu verbessern. Zurzeit müssen Landwirte Ernteverluste von bis zu 25 Prozent hinnehmen, weil die Rapsschoten platzen und ihre Samen vor der Ernte freilassen. Ziel der Kieler Wissenschaftler waren daher Pflanzen mit platzfesten Schoten. Dazu führten sie mit CRISPR/Cas9 Mutationen in zwei nahezu identische Gene ein.
Als nächstes wollen die Forscher die sekundären Pflanzenstoffe ins Visier nehmen. CRISPR/Cas soll dabei helfen, ernährungsphysiologisch ungünstige Inhaltsstoffe wie Glucosinolate oder Phytinsäure aus den Samen zu eliminieren und damit die Qualität der Rapsprodukte zu verbessern.
Quellen:
- Shin, J. et al. (2017): Disabling Cas9 by an anti-CRISPR DNA mimic. In: Science Advances Vol. 3, (12. Juli 2017), doi: 10.1126/sciadv.1701620.
- Braatz, J. et al. (2017): CRISPR-Cas9 targeted mutagenesis leads to simultaneous modification of different homoeologous gene copies in polyploid oilseed rape (Brassica napus L.). In: Plant Physiology, (18. April 2017), doi: 10.1104/pp.17.00426.
Zum Weiterlesen:
- Effiziente neue Genschere – Die Genschere CRISPR/Cpf1 arbeitet präziser als das bisherige Standardwerkzeug CRISPR/Cas9
- Mit CRISPR/Cas gegen Zitruskrebs – Forscher setzen erfolgreich auf Promotor-Editing
- RNA Live-Tracking mit CRISPR/Cas – Forscher nutzen CRISPR/Cas-Komplex als “GPS-Sender” für RNA
Titelbild: Cas9 (grau) und hat ein spezifisches DNA-Stück (lila) erreicht, an das es andockt, bevor es zur Tat schreitet. Zu diesem DNA-Stück wird es durch die Guide-RNA (orange) geleitet. (Bildquelle: © Fuguo Jiang, UC Berkeley)
