No more Lollipops
Neue miRNA-Software setzt auf maschinelles Lernen und künstliche Intelligenz

Eine von vier Modellpflanzen, bei der die neue Software auf die Suche nach MicroRNA (miRNA) ging: Arabidopsis thaliana (Bildquelle: © Carl Davies / CSIRO/ CC BY 3.0)
„Klein aber Oho!“ „In der Kürze liegt die Würze!“ Passende Aphorismen zur Umschreibung von MicroRNA sind leicht gefunden. Ganz anders verhält es sich mit den kurzen RNA-Sequenzen selbst. Forscher haben nun eine neue Software (Naive Bayesian Classifier) entwickelt, die die Suche und Identifizierung von MicroRNA erleichtern soll.
MicroRNA-Sequenzen (miRNA) sind kurze, einzelsträngige, nichtkondierende RNA-Abschnitte, die an der Transkriptionsregulation von Mensch, Tier und Pflanze beteiligt sind. Bekannt sind sie dafür, Gene nach der Transkription (posttranscriptional gene silencing) hochspezifisch stillzulegen (RNA silencing). Sie tun dies, indem sie an der Boten-RNA (mRNA) andocken, ihre Expression bzw. Translation blockieren oder gleich zerstören. Auf genetischer Ebene sind sie damit zwar kleine, aber sehr einflussreiche Mitspieler. Aufgrund ihrer Größe und ihrer großen Ähnlichkeit zu anderen RNA-Fragmenten sind sie jedoch unbequeme Forschungsobjekte.
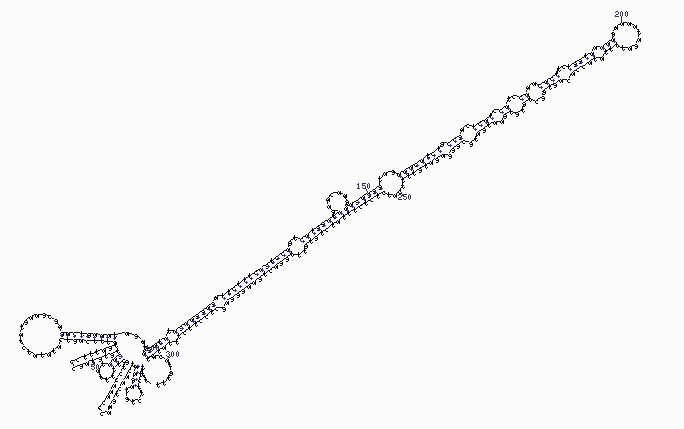
Schematische Darstellung der MicroRNA. Deutlich erkennbar sind die Nadelöhrform und die Lollipop-Struktur am oberen Ende des Moleküls.
Bildquelle: © Opabinia regalis/ wikimedia.org/ CC BY-SA 3.0
Pflanzenforscher im Nachteil
Hinzu kommt, dass sich zwar die Entstehung von miRNAs in Pflanzen und tierischen Organismen ähnelt, darüber hinaus jedoch gravierende Unterschiede existieren. Diese sind so groß, dass Pflanzenforscher bisher nicht von den Verfahren profitieren konnten, die für die miRNA-Analyse in tierischen Systemen entwickelt wurden. Speziell für sie hat nun ein Team aus Bioinformatikern eine Software entwickelt, die den Umgang mit Pflanzen-miRNA erleichtern soll. Kennzeichen ist, dass sie zu maschinellem Lernen (Deep Learning) fähig ist und damit eine Abkehr vom starren filter- und strukturbasierten Ansatz darstellt. Ein entscheidender Fortschritt im Vergleich zu den bisher gebräuchlichen Methoden.
Die Suche nach der Haarnadel im Heufhaufen
An ihnen bemängeln die Wissenschaftler vor allem das starre und grobe Raster, durch das viele miRNA-Sequenzen fallen. Zum Beispiel, wenn sie nicht aus den standardmäßigen 21 bis 23 Nukleotiden bestehen oder die Suche nach der charakteristischen Nadelöhr- oder Lollipop-Struktur ergebnislos verläuft.
Im Test an vier sehr unterschiedlichen Modellpflanzen – Arabidopsis thaliana, Soja (Glycine max), Reis (Oryza sativa) und Pfirsich (Prunus persica) – bewiesen die Forscher, dass ihre Software tatsächlich mehr und bessere Resultate liefert als andere Verfahren. Vor allem, dass sie miRNA-Fragmente aufspürt und identifiziert, die außerhalb der gängigen Norm liegen oder anderen RNA-Fragmenten zum Verwechseln ähneln, z.B. small interfering RNA-Sequenzen (siRNA), die meist aus 24 oder 25 Nukleotiden bestehen.
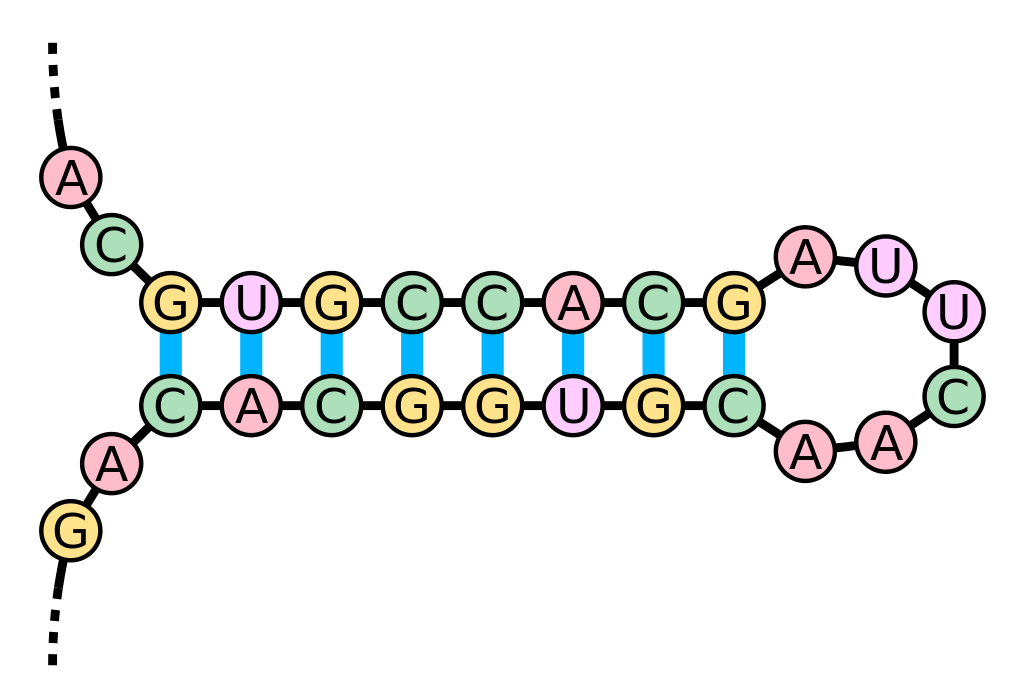
Schematische Vergrößerung der Lollipop-Struktur.
Bildquelle: © Sakurambo/ wikimedia.org/ CC BY-SA 3.0
NBC - Naive Bayes Classifier
Doch wie gelingt es der Software namens „Naive Bayes Classifier“ (NBC), die Spreu vom Weizen zu trennen, miRNA-Sequenzen von anderen RNA-Schnipseln zu unterscheiden? Ausgangspunkt bilden jeweils drei Datensätze pro Modellpflanze: Einer mit den Sequenzinformationen aller kurzen RNA-Moleküle (sRNA), u. a. miRNA, siRNA, snRNA etc. Ein weiterer Datensatz mit bereits bekannten miRNA-Molekülen und letztlich das Referenzgenom der Versuchspflanze selbst.Mit Hilfe von fünf kombinierten Parametern pickt die Software nun unter allen sRNA-Molekülen die gesuchten miRNA-Sequenzen heraus. Doch um welche Parameter handelt es sich dabei?
5 Parameter machen den Unterschied
Da ist zum einen die Sequenzlänge, mit der die Suche auf RNA-Sequenzen bestehend aus Nukleotidbausteinen im unteren zweistelligen Bereich eingegrenzt wird, wodurch längere sRNA Sequenzen ausgesiebt werden. Zweitens die Häufigkeit, wie oft ein RNA-Molekül auftaucht, da miRNA allgemein seltener vorkomme als andere sRNA, so die Entwickler. Für die NBC-Software ergibt sich daraus eine höhere Wahrscheinlichkeit, eine miRNA-Sequenz gefunden zu haben, je seltener sie im Datensatz auftaucht.
Als dritter Parameter kommt die Verteilung ins Spiel. Während viele andere sRNA-Moleküle dazu neigen, Cluster zu bilden, sind miRNA-Moleküle typischerweise großflächig verteilt, sprich im gesamten Datensatz auffindbar und nicht in einigen wenigen Regionen gehäuft. Typisch ist auch, dass sie im Vergleich zu anderen sRNA-Molekülen häufig gleich mehreren genetischen Ursprungsorten im Referenzgenom zuordenbar sind (Multi Mapping Reads). Hier sprechen die Forscher von Multiplizität, dem vierten Parameter. Je höher sie ist, desto höher auch die Wahrscheinlichkeit, es mit einer miRNA-Sequenz zu tun zu haben.
Ein Blick in die Entstehungsgeschichte der miRNA
Zuletzt erfolgt die Suche nach miRNA-Komplementärsträngen. Die Gelegenheit für einen Kurzabriss zur Entstehungsgeschichte : Sie beginnt mit einer doppelsträngigen RNA aus mehreren Hundert Nukleotiden, die primary-miRNA (pri-miRNA), welche von einem RNA spaltenden Enzym (RNAse III Dicer-like1 – kurz DLC1) mehrmals zerteilt wird.

Den Forschern gelang es, mit Hilfe der NBC-Software miRNA-Sequenzen bei Soja zu detektieren. Und das ohne Vorwissen bzw. vorhandene Daten zu möglichen miRNA-Kandidaten.
Bildquelle: © US Department of Agriculture / wikimedia.org/ CC0
So entstehen mehrere kürzere (i.d.R. aus 70 Nukleotiden bestehende) precursor-miRNA-Sequenzen (pre-miRNA), die erneut dem Enzym DLC1 zum Opfer fallen. Auf diese Weise entsteht der direkte miRNA-Vorläufer: Ein miRNA-Doppelstrang (miRNA:miRNA*) mit der charakteristischen Nadelöhr- oder Lollipop-Struktur, dessen Komplementärstrang (miRNA*) abgebaut wird, bis nur noch ein Strang übrig bleibt. Wonach die Software im Grunde sucht, sind noch intakte miRNA*-Sequenzen. Aus der Kombination aller fünf Parameter berechnet die Forscher die Wahrscheinlichkeit, mit der es sich bei einer kurzen RNA-Sequenz um miRNA handelt oder nicht.
Software mit Verstand
Der entscheidende Vorteil der neuen Software besteht darin, zu lernen, automatisiert und autonom zu arbeiten. Dank des Abgleichs innerhalb der drei Datensätze werden keine strikten Filter zur Kategorisierung oder Klassifizierung mehr benötigt. An ihre Stelle treten stringente Filter, die von der Software unter Berücksichtigung aller zur Verfügung stehenden Sequenzinformationen pflanzenspezifisch gesetzt werden.
Neue Impulse für die miRNA-Forschung
Zwar räumen die Forscher ein, dass ihre Entwicklung noch in den Kinderschuhen stecke, die Software vorerst nur für die Hypothesenbildung geeignet sei und noch nicht zur Beweisführung tauge, dennoch sind sie vom Nutzen überzeugt. Am Beispiel der Sojapflanze konnten sie zeigen, dass die Software bereits in der Lage ist, miRNA eigenhändig und ohne Vorwissen zu detektieren. Die Forscher erhoffen sich dadurch neue Impulse für die Forschung zum Thema Pflanzen-miRNA. Zumal vieles nach wie vor im Verborgenen liegt, z.B. die Wechselwirkung mit bekannten Zielmolekülen im Speziellen oder die Bandbreite der Zielmoleküle im Allgemeinen.
Fakt ist, dass miRNA-Moleküle trotz ihrer geringen Größe nicht unterschätzt werden dürfen, da sie an wichtigen Prozessen entscheidend beteiligt sind. Dazu zählen unter anderem das Abpassen des optimalen Blühzeitpunkts oder die Erhöhung der Widerstandsfähigkeit gegenüber biotischem (Krankheiten) und abiotischem Stress (Kälte). Allesamt Eigenschaften, die sie zu praktischen Werkzeugen für die Pflanzenzucht machen.
Quelle:
Douglass, S. et al. (2016): A Naive Bayesian Classifier for Identifying Plant miRNAs. In: the plant journal Vol. 86 (117-207), (7. April 2016), DOI:10.1111/tpj.13180
Zum Weiterlesen auf Pflanzenforschung.de:
- Neue Methode zur Strukturaufklärung von RNA
- Mikro-RNAs in Pflanzen: Neuer Regulator entdeckt
- RNA-Viren-Abwehr
Titelbild: Eine von vier Modellpflanzen, bei der die neue Software auf die Suche nach MicroRNA (miRNA) ging: Arabidopsis thaliana (Bildquelle: © Carl Davies / CSIRO/ CC By 3.0)
