24.02.2010
Basisinfo
Neue Verfahren
Gene Targeting – Gezielter Einbau von Genen ins Pflanzengenom
In der Theorie leicht, in der Praxis schwierig: Durch Gene Targeting (target, engl.= Ziel) könnten Gene gezielt ausgeschaltet, verändert oder ersetzt werden. Durch den gezielten Einbau von Genen ins Pflanzengenom könnten unerwünschte Nebeneffekte weiter minimiert werden. Seit langem wird das Gene Targeting in Bakterien, Hefen und bei der Maus erfolgreich angewendet. Der Einsatz in der Pflanze gestaltet sich jedoch als sehr schwierig.
Die Basis für das Gene Targeting ist die homologe
Rekombination, ein natürlich vorkommender
Mechanismus, bei dem homologe, d.h. identische oder ähnliche DNA-Abschnitte im
Erbgut gegeneinander ausgetauscht werden. Dies kann man dazu nutzen, ein neues
Gen gezielt an einer bestimmten Stelle im Erbgut zu integrieren. Dazu
muss das Gen an beiden Seiten von DNA-Abschnitten flankiert sein, die homolog zu
dem geplanten Integrationsort sind (siehe Schaubild).

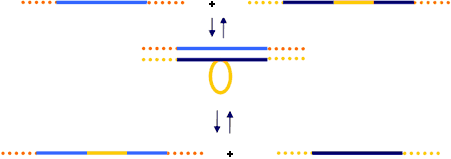
Homologe Rekombination
Austausch homologer Sequenzen. (links) Zwei DNA-Stränge mit homologen DNA-Sequenzen (hellblau, dunkelblau) werden ausgetauscht.
Austausch eines nicht-homologen DNA-Abschnitts. (unten) Ist ein Gen (orange) von Sequenzen flankiert, die zu einer anderen Stelle im Erbgut homolog sind, kann es aus dem ursprünglichen Ort ausgeschnitten und am anderen eingefügt werden.
Soweit die Theorie. In der Praxis jedoch konnte für Pflanzen bislang noch keine effiziente Methode für Gene Targeting etabliert werden. Dies liegt unter anderem daran, dass die homologe Rekombination in der Pflanze nicht oft und effizient genug stattfindet. Ein Ansatz ist deshalb, die homologe Rekombination in der Pflanzenzelle mit Hilfe von Transposons und bakteriellen Rekombinationssystemen zu verstärken. Eine andere Methode stimuliert die natürlich in der Pflanzenzelle vorkommenden Rekombinationsvorgänge, damit sie häufiger und effizienter stattfinden.
Gene Targeting mit Transposons und bakteriellen Rekombinationssystemen
Transposons und Rekombinationssysteme bestehen aus bestimmten Erkennungssequenzen sowie einem Enzym (Transposase bzw. Rekombinase), das an diesen Erkennungssequenzen DNA-Abschnitte aus dem Genom ausschneiden und an anderer Stelle wieder integrieren kann. Im Unterschied zu Transposasen benötigen Rekombinasen auch für den Einbau eines DNA-Abschnittes Erkennungssequenzen. Transposasen können hingegen das ausgeschnittene DNA-Stück an beliebiger Stelle im Erbgut wieder einbauen, in der Regel integrieren sie es aber bevorzugt in Regionen mit hoher Gen-Expression.
In einem abgeschlossenen SiFo-Projekt wurden mit Hilfe einer Rekombinase gleichzeitig ein Markergen entfernt und ein anderes Gen an dessen Stelle gezielt eingebaut. Dafür mussten beide Gene von Sites eingeschlossen sein.
In einem aktuellen SiFo-Projekt wird in das Genom von Rapspflanzen ein gentechnisch verändertes Transposon eingeschleust, das zugleich die Erkennungssequenzen für eine Rekombinase enthält. Darüberhinaus enthält es ein Reportergen, mit dessen Hilfe sich messen lässt, ob das Transposon in einer Region mit hoher Genexpression eingebaut wurde. Denn die Integrationsstelle des Transposons ist gleichzeitig der Ort, an dem später ein Gen gezielt eingebaut werden soll. Dieser Genort sollte daher möglichst „aktiv“ sein.
Mit molekularbiologischen Methoden wird untersucht, ob sich durch den Einbau keine unerwünschten Veränderungen im Genom wie z.B. die Unterbrechung eines Pflanzengens ereignet haben. Pflanzen, auf die beides zutrifft, werden als geeignete Empfängerlinien für den späteren gezielten Geneinbau ausgewählt.
In das Erbgut der Empfängerlinien kann anschließend mit Hilfe einer Rekombinase jedes gewünschte Transgen zielgerichtet an der Integrationsstelle des Transposons eingebracht werden.
Gene Targeting durch Stimulation zelleigener Rekombinationsvorgänge
Bei dieser Methode wird durch ein spezielles DNA-schneidendes Enzym (Restriktionsenzym „I-SceI“) an zwei Stellen im Pflanzengenom ein Bruch des DNA-Stranges erzeugt. An dieser Stelle können anschließend Rekombinationsvorgänge und somit eine Austausch von homologen Sequenzabschnitten gezielt ablaufen.
Schritt 1: Herstellung von zwei verschiedenen transgenen Pflanzenlinien
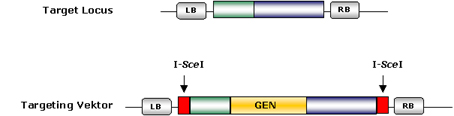
Es werden zunächst zwei unterschiedliche transgene Pflanzenlinien hergestellt, die jeweils einen Target Locus (den Zielort für die Integration eines neuen Gens) und einen Targeting Vector enthalten. Mit Hilfe von DNA-Analysen wird überprüft, ob der Target Locus an einer gewünschten Stelle des Pflanzengenoms sitzt.
Der Targeting Vector enthält eine zum Target
Locus homologe DNA-Sequenz (grün und blau in der Abbildung), in die das zu
übertragene neue Gen eingebettet ist. Außerdem wird der Targeting Vector
flankiert von zwei Schnittstellen für das Restriktionsenzym „I-SceI“.
Schritt 2: Kreuzung der Pflanzenlinien
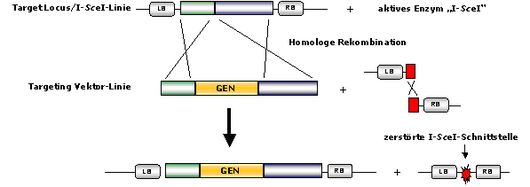
Beide Pflanzenlinien, die Target Locus-Linie und die Targeting Vector/I-SceI-Linie, werden anschließend miteinander gekreuzt.
Durch das Restriktionsenzym „I-SceI“ wird der Targeting Vector an den I-SceI-Schnittstellen geschnitten. Dadurch wird eine homologe Rekombination zwischen dem Targeting Vector mit dem Target Locus ausgelöst.
Die Schnittstelle, aus welcher der Targeting Vector herausgeschnitten wurde, wird
durch homologe oder illegitime Rekombination wieder geschlossen. Die
Schnittstellen werden dabei zerstört, der Vorgang ist somit irreversibel und der
Targeting Vector kann nicht wieder in seinen Ursprungsort integriert werden.
Schritt 3: Analyse der Pflanzen
Mittels DNA-Analysen müssen dann die Pflanzen herausgesucht werden, bei denen das Gene Targeting tatsächlich funktioniert hat.
Um das System des Gene Targeting in der Pflanze zu etablieren, wird zunächst mit Markergenen gearbeitet. Dies erleichtert es, das System so effizient wie möglich zu machen. Sobald dies gelungen ist, kann das Markergen durch ein „Gen von Interesse“ ausgetauscht werden. Ein langfristiges Ziel ist es zudem, jede natürlich vorkommende Sequenz im Pflanzengenom durch Gene Targeting als Zielort für eine Genintegration verfügbar zu machen.
Mehr bei bioSicherheit
Thematische Verknüpfungen
Forschungsprojekte
2008-2011
- Gezielter Einbau von Genen an definierten Positionen im Rapsgenom, TU Braunschweig
- Entwicklung einer Technik zur zielgerichteten Integration von Genen bei Pflanzen, Universität Karlsruhe
2005-2008
- Gezielter Einbau von Genen mittels Gene Targeting; Universität Karlsruhe
- Gezielter Einbau von Genen bei gleichzeitiger Entfernung des Markergens durch ortsspezifische Rekombinationssysteme; Universität Hamburg
