Effiziente Genomeditierung mit weniger Nebeneffekten
Vielversprechende Weiterentwicklungen der CRISPR/Cas9-Methode

DNA-Doppelstrang: Mit dem „Prime Editing“ können auch ohne Doppelstrangbruch ganze Sequenzen ins Genom eingefügt werden. (Bildquelle: © Arek Socha/Pixabay/CC0)
Biochemikern ist es gelungen, eine Methode zur Genomeditierung zu entwickeln, die mit hoher Effizienz und wenig Nebenwirkungen neue DNA-Sequenzen präzise einfügen kann. Vor drei Jahren wurden nach dem Prinzip erstmals Punktmutationen nebenwirkungsarm korrigiert.
Die erst wenige Jahre alte Methode der Genomeditierung CRISPR/Cas9 hat bereits eine Revolution in der Genforschung ausgelöst. Die Heilung von Erbkrankheiten erschien als Möglichkeit am Horizont, ebenso wie ein schnellerer Weg, Pflanzen mit neuen und verbesserten Eigenschaften zu züchten. Doch obwohl die Methode kontinuierlich verbessert wird, hat sie noch immer eine Schattenseite: Bei aller Präzision, mit der auf diese Weise Mutationen in einen Genomabschnitt eingefügt werden können, entstehen auch ungewollte Veränderungen an der Editierungsstelle.
Was in der Pflanzenzüchtung den Züchtungsfortschritt konterkarieren kann, ist in der Humanmedizin ethisch gar nicht zu vertreten. Genau dieses Problem haben US-amerikanische Biochemiker 2016 mit einer Weiterentwicklung der Methode deutlich verringert: Anstatt eine DNA-Sequenz einzufügen, verändern sie eine einzelne Base des Genoms und zwingen den gegenüberliegend Strang zur Homologie.
Dieser Vorgang wird als Basen-Editierung bezeichnet. Denn häufig genügt die Editierung einer einzelnen Base, um ein defektes Gen zu reparieren. Jetzt setzen die Forscher noch eins drauf und präsentieren eine Methode, die genauso nebenwirkungsarm nicht nur gewünschte Punktmutationen ins Genom einfügt, sondern auch längere Sequenzabschnitte verändern kann.
Standardmethode mit Effizienzproblemen und Nebenwirkungen
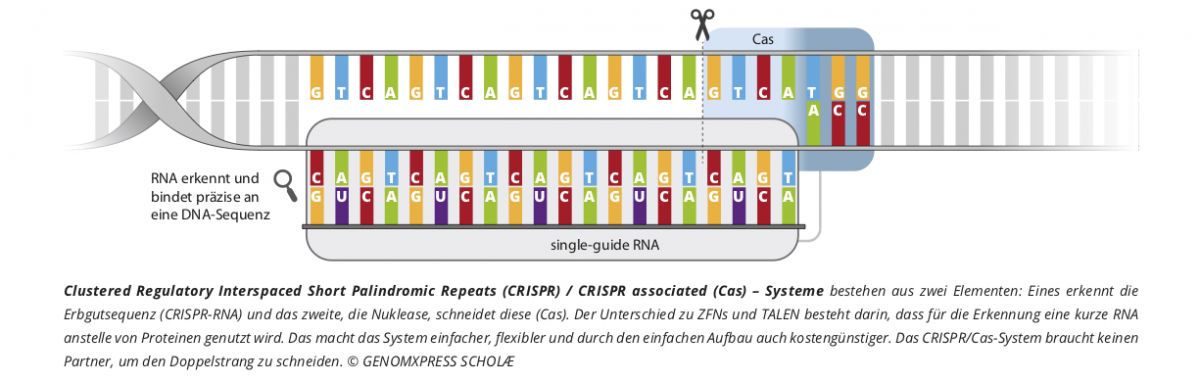
CRISPR-Cas9 funktioniert, indem eine daran gebundene RNA-Zielsequenz diesen Komplex an eine definierte Stelle des DNA-Doppelstrangs leitet. Dort erzeugt Cas9 einen Doppelstrangbruch. Natürliche Reparaturmechanismen der Zelle führen dazu, dass bei der Wiedervereinigung der Strangenden zufällige Insertionen oder Deletionen (Indels) entstehen. Liegt ein homologer DNA-Strang vor, kann dieser an der Schnittstelle eingefügt werden. Dieses Verfahren nutzen Forscher, um zusätzliche DNA-Sequenzen ins Erbgut einzufügen. Beide Prozesse konkurrieren, und die direkte Wiedervereinigung mitsamt ihrer Indels kommt weit häufiger vor als der Einbau der neuen DNA-Sequenz.
Base ändern statt Sequenz einfügen
In vielen Fällen ist es jedoch gar nicht notwendig, eine längere DNA-Sequenz einzubauen. Zahlreiche Erbkrankheiten des Menschen und auch der Verlust von Resistenzen oder bestimmten sekundären Pflanzenstoffen bei Kulturpflanzen gehen auf Punktmutationen zurück, die das betroffene Gen inaktivieren oder zu einer veränderten, dysfunktionalen Proteinstruktur des Genprodukts führen. Was wäre also, wenn man die DNA-spaltende Aktivität von CRISPR/Cas9 ausschaltet und den Komplex mit einer Basen-Editierungsfunktion ergänzt?
Genau das ist den US-Forschern 2016 gelungen. Zunächst hat das Team Cas9 so mutiert, dass das Enzym seine Nukleaseaktivität verloren hat (dCas9), also den DNA-Strang nicht mehr schneiden konnte. Dann haben sie eine Cytidindeaminase an den Komplex gebunden, die Cytosin in Uracil verwandelt. Uracil hat für die Basenpaarung die gleiche Funktion wie Thymin. Allerdings reagieren Cytidindeaminasen nur mit Einzelsträngen der DNA. Das passt jedoch, da eine Sequenz von etwa neun Basenpaaren im Bereich des dCas9-Ziel-DNA-Komplexes als geöffneter Doppelstrang und damit für die Deaminase zugänglich vorliegt.
Baseneditor der ersten Generation
Als besonders aktiv erwies sich dabei die aus der Ratte stammende Deaminase APOBEC1 in Kombination mit dem Linker XTEN. Den Komplex APOBEC1-XTEN-dCas9 bezeichnen die Forscher als Baseneditor der ersten Generation. In vitro testeten die Biochemiker den Baseneditor an sieben Fällen, in denen die Mutation von Thymin zu Cytosin einen krankheitsauslösenden Effekt hat. In sechs von sieben Fällen resultierten Genprodukte, die auf eine erfolgreiche Editierung hindeuteten. Die Effizienz lag dabei bei 44 Prozent.
Übersicht: Nukleobasen, auch Nukleinbasen
| Base: | Bestandteil von: |
| Adenin (A) | DNA und RNA |
| Cytosin (C) | DNA und RNA |
| Guanin (G) | DNA und RNA |
| Thymin (T) | nur DNA |
| Uracil (U) | nur RNA |
Die Überraschung folgte beim Test an menschlichen Zellen: Gegenüber den in vitro-Versuchen sank die Effizienz der Basen-Editierung bis um den Faktor 36. Zelleigene Reparaturmechanismen hatten auf die Uracil-Guanin-Paarung angesprochen, so die Vermutung der Wissenschaftler. Also erweiterten die Biochemiker ihren Editierungskomplex um einen Uracil-DNA-Glykosylase-Inhibitor, der die natürliche Reparatur verhindert. Der Baseneditor der zweiten Generation entstand – und bewährte sich: Die Konversionseffizienz in lebenden Zellen war dreimal höher als noch in der ersten Generation und erreichte bis zu 20 Prozent. Bemerkenswert: In weniger als 0,1 Prozent der Fälle entstanden Indels.
Baseneditor der dritten Generation
Um die Effizienz noch weiter zu steigern, erdachte das Team eine dritte Generation Baseneditoren. Diese hatten zum Ziel, auch das Guanin des nicht editierten Stranges zu korrigieren. In eukaryotischen Zellen gibt es einen natürlichen Mechanismus, der auf Einzelstrangbrüche in neu synthetisierter DNA reagiert, indem er die nicht homologe Stelle abbaut und neu synthetisiert. Die Forscher veränderten daher ihren Baseneditorkomplex so, dass er im nicht editierten Strang einen Bruch hervorruft. Das Resultat war eine weitere Effizienzsteigerung auf nun bis zu 37 Prozent. Allerdings stieg der Anteil der Indels auf durchschnittlich 1,1 Prozent an. Zum Vergleich: Mittels der konventionellen CRISPR/Cas9-Methode erzielten die Forscher lediglich eine Effizienz von 0,5 Prozent bei 4,3 Prozent Indels.
Inzwischen wurde die Basen-Editierung weiterentwickelt und kann für alle vier Transitionsmutationen (C→T, G→A, T→C, A→G) genutzt werden. Transversionsmutationen (C→A, C→G, G→C, G→T, A→C, A→T, T→A, T→G) sind mit der Technologie jedoch nicht möglich.
Prime Editing ergänzt Basen-Editierung
Jetzt hat das Team um David Liu einen noch größeren Durchbruch vermeldet: Basierend auf Ideen der Basen-Editierung haben die Forscher „Prime Editing“ entwickelt. Damit lassen sich ohne Doppelstrangbruch hoch effizient ganze Sequenzen ins Genom einfügen, bei deutlich geringeren Nebeneffekten als der klassischen CRISPR-Cas9-Methode. Außer Insertionen sind zudem Deletionen und alle zwölf Basenkonversionen möglich sowie Kombinationen daraus.
Das Prinzip setzt wieder bei der CRISPR-Methode an. Auch beim Prime Editing nutzt das Enzym Cas9 eine Guide-RNA, um an die Ziel-DNA zu binden. Allerdings enthält diese RNA zudem die genetische Information, wie die DNA editiert werden soll. Deshalb haben die Forscher sie „Prime Editing Guide RNA“, kurz: pegRNA, getauft. Außerdem erzeugt der Editierkomplex an der Bindestelle einen Einzelstrangbruch (einen sog. Nick). Dadurch entsteht eine frei 3‘-Hydroxylgruppe, die eine reverse Transkription auslöst, bei der die einzufügende Sequenz der pegRNA in DNA übersetzt wird.
Natürliche Mechanismen erübrigen Doppelstrangbruch
CRISPR-Cas9: Die erst wenige Jahre alte Methode der Genomeditierung hat bereits eine Revolution in der Genforschung ausgelöst.
Bildquelle: © molekuul.be - Fotolia.com
Jetzt liegt der DNA-Doppelstrang als ein verzweigtes Zwischenprodukt vor, das zwei offene Einzelstrangüberhängsel aufweist: ein 5‘-Ende mit der Originalsequenz sowie ein 3‘-Ende mit der editierten Sequenz, die von der pegRNA kopiert wurde. Weil strukturspezifische Endonukleasen bevorzugt offene 5‘-Enden abbauen, wird die editierte 3‘-Sequenz in den dann wieder geschlossenen Doppelstrang eingefügt.
An dieser Stelle greifen die Forscher auf einen Trick aus der Basen-Editierung zurück: Sie verpassen dem unveränderten DNA-Strang mittels Cas9-Nickase einen Einzelstrangbruch. Der schon bekannte natürliche Mechanismus baut in der Folge die nicht homologe Sequenz im offenen Strang ab und repariert den Strang homolog zur editierten Sequenz im unversehrten Strang. Die Editierung beider Stränge ist erfolgreich abgeschlossen.
Weniger Nebeneffekte, aber immer noch zu viele
Effizienz und Spezifität der Methode variieren zwar, anhängig vom Zelltyp, in dem sie genutzt werden. Doch die unerwünschten Nebeneffekte sind deutlich geringer als bei CRISPR-Cas9, während die Stärken und Schwächen im Wesentlichen der Basen-Editierung entsprechen. Basenkonversionen erfolgen beispielsweise mit einer Effizienz von durchschnittlich 33 Prozent und 7,5 Prozent Indels. Insertionen und Deletionen mit ein bis drei Basen erzielten 32 bis 39 Prozent bzw. 29 bis 32 Prozent Effizienz mit im Durchschnitt 6,8 Prozent Indels. Deletionen mit bis zu 80 Basen konnten die Forscher mit bis zu 78 Prozent Effizienz und 11 Prozent Indels durchführen. Abschließend testete das Team eine Kombination aus zwölf Editierungen. Deren Effizienz betrug 55 Prozent bei 6,4 Prozent Indels.
Die Ergebnisse zeigen, dass die neue Methode immer noch mit hohen Nebeneffekten zu kämpfen hat. Ähnlich wie bei CRISPS-Cas9 sind aber auch hier stetige Verbesserungen zu erwarten. Die Effizienz hingegen ist bereits auf einem praxistauglichen Niveau. Während somit ethische Aspekte einen baldigen Einsatz in der Humanmedizin unwahrscheinlich erscheinen lassen, dürften Anwendungen in der Pflanzenzüchtung nicht lange auf sich warten lassen.
Quellen:
- Komor, A. et al. (2016): Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. In: Nature, volume 533, pages 420–424, (19. Mai 2016), doi: 10.1038/nature17946.
- Anzalone, A. et al. (2019): Search-and-replace genome editing without double-strand breaks or donor DNA. In: Nature, (Accelerated Article Preview, 21. Oktober 2019), doi: 10.1038/s41586-019-1711-4.
Zum Weiterlesen auf Pflanzenforschung.de:
- Was CRISPR/Cas leisten kann - Der Editierung von Genomen scheinen technologisch kaum noch Grenzen gesetzt
- CRISPR/Cas: Baukasten zur Editierung von Genomen wird erweitert - Basen-Editierung ermöglicht jetzt Korrektur von Punktmutationen in DNA und RNA
- Wie CRISPR/Cas funktioniert - Eine einfache Technologie verbessert die Effizienz der Genom Editierung
Titelbild: DNA-Doppelstrang: Mit dem „Prime Editing“ können auch ohne Doppelstrangbruch ganze Sequenzen ins Genom eingefügt werden. (Bildquelle: © Arek Socha/Pixabay/CC0)