Ethanol aus pflanzlichen Zellwänden

Die Ackerschmalwand (Arabidopsis thaliana) diente den Forschern als Versuchsobjekt. (Quelle: © GABI Geschäftsstelle)
Bioethanol wird bislang meist aus Samen und Pflanzensäften gewonnen. Die Forscher im Projekt GABI-CELLWALL wollen die Struktur pflanzlicher Zellwände so verändern, dass auch diese als Zuckerquelle für die Ethanolherstellung geeignet sind.
Schon in der Frühzeit des Automobils, in den 1920er Jahren, war Bioethanol als Treibstoff im Gespräch. Heute, in Zeiten der Klimaerwärmung, finden erneuerbare Treibstoffe mehr Beachtung denn je. Doch Bioethanol der so genannten ersten Generation, hergestellt aus Weizen- und Maiskörnern, Zuckerrohr und Zuckerrüben, konkurriert mit Lebens- und Futtermitteln um die Rohstoffe. Pflanzenforscher wollen deshalb neue Quellen für Bioenergie erschließen: Entweder Pflanzenbestandteile, die Bei- oder Abfallprodukte aus der Lebens- und Futtermittelindustrie sind, oder gleich Pflanzenarten, die nicht der Ernährung dienen.
Keine Konkurrenz zu Nahrungsmitteln.
Das Projekt GABI-CELLWALL analysiert deshalb die pflanzliche Zellwand. Während Ethanol der ersten Generation aus Stärke entsteht, enthält die pflanzliche Zellwand vor allem Zellulose. Die besteht zwar ebenso wie Stärke aus Glukosebausteinen, doch sind diese in der Zellwand einer einzigen Zelle auf mehr als 50 verschiedene Weisen miteinander verknüpft. Dort endet die Komplexität aber noch nicht: Unterschiedliche Gewebe und Organe haben spezialisierte Zellwände, die anders aufgebaut sind. Will man die Zellulose der Zellwände nutzen, um Ethanol zu erzeugen, muss man diese komplexen Strukturen verstehen.
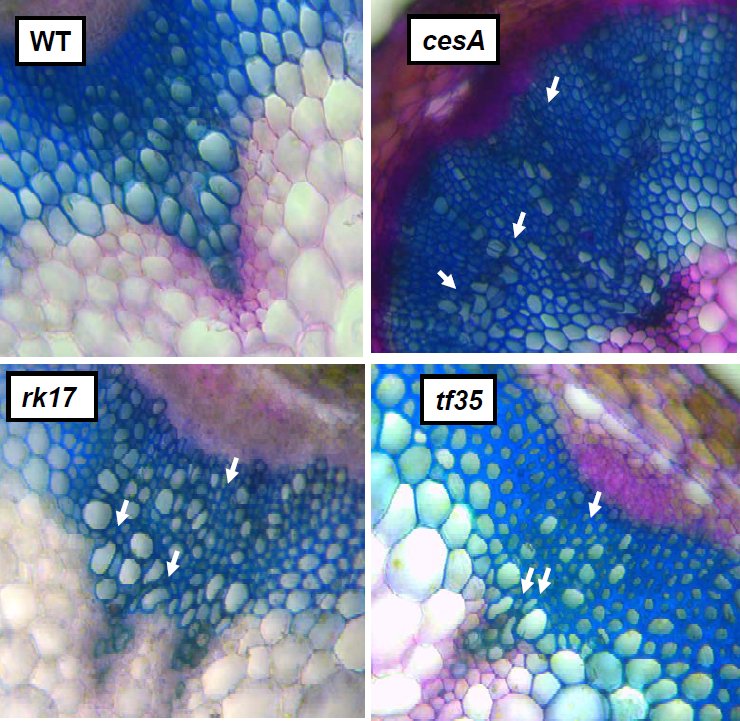
Querschnitte von gefärbten 4-6 Wochen alten Ackerschmalwand Stämmen.
Bildquelle: © Lutz Neumetzler, AG Persson, MPIMP, Golm
Darin besteht die erste Aufgabe der GABI-CELLWALL-Forscher. Anfänglich war lediglich bekannt, dass die Pflanze die Komponenten der Zellwandkomplexe gemeinsam exprimiert. Das Forschungsprojekt will nun die regulatorischen Netzwerke aufdecken, die den Stoffwechsel der pflanzlichen Zellwand steuern. Konkret bedeutet das, die Transkriptionsfaktoren und Rezeptorkinasen zu finden, die für jene Enzyme verantwortlich sind, die die Zellwände auf-und umbauen.
Zellwände verändern, ohne sie zu zerstören
Dann wird es erst richtig kniffelig, denn das langfristige Ziel besteht darin, die Ethanolgewinnung aus Zellwänden auch rentabel zu machen. Dazu müssen die Forscher die Zellwände so verändern, dass sie leicht verwertbare Zuckerbausteine in möglichst großer Menge beinhalten – ohne wichtige Eigenschaften der Pflanze dadurch zu beeinträchtigen. Würde die Zellwand aufgrund der neuen Struktur kollabieren, wäre schließlich nichts gewonnen. Und auch der Stofftransport in die Zelle hinein und hinaus sowie die Erkennung von Stoffen an der Zelloberfläche dürfen nicht gestört werden. Letzteres ist beispielsweise wichtig, um das pflanzliche Immunsystem zu aktivieren, wenn Pathogene eingedrungen sind. Da sich GABI-CELLWALL auf die sekundäre, also die innere Zellwand konzentriert, ist das Risiko jedoch gering, dass die Pflanze auf diese Weise Schädlingsresistenzen verliert.
Deshalb schalten die Forscher der Reihe nach an der Zellwandbildung beteiligte Gene aus und untersuchen, wie sich die sekundäre Zellwand dadurch verändert. Die gefundenen Zusammenhänge hat GABI-CELLWALL online zusammengefasst: Für die Ackerschmalwand und sieben weitere Arten gibt es dort ein Co-Expressions-Tool, das auch andere Forscher nutzen können, um sowohl die Co-Expression von für die Zellwand relevanten Genen als auch die transkriptionelle Koordination von anderen Stoffwechselprozessen zu studieren.
Die Forscher von GABI-CELLWALL müssen nun herausfinden, wie sie die regulatorischen Module an- und ausschalten oder gegen einander austauschen, um hochkomplexe Zuckerpolymere durch weniger komplexe zu ersetzen, ohne die Fitness und die Biomasse der Pflanze zu sehr zu beeinträchtigen. Diese ehrgeizige Idee soll innerhalb der nächsten Jahre ein solides Fundament erhalten.

Das Forscherteam: Lutz Neumetzler, Marek Mutwil, Staffan Persson, Thibaut Douché, Elisabeth Jamet, Hugo Alonso, Michael Harrington, Elene R. Valdivia, Javier Sampedro
Bildquelle: © GABI-CELLWALL
Pflanzen mit einem wie mit zwei Keimblättern berücksichtigt
Wichtig ist den Forschern, ihre Erkenntnisse nicht nur für die Modellpflanze Ackerschmalwand (Arabidopsis thaliana) zu gewinnen, sondern auch für das Süßgras Zwenke (Brachypodium distachyon), dessen Genom unlängst ebenfalls entschlüsselt worden ist. Dadurch sind Pflanzen mit einem wie mit zwei Keimblättern berücksichtigt. Sowohl Gras- und Getreidearten als auch Laubgehölze könnten somit später für die Bioethanolgewinnung in Frage kommen.
Einige Schritte auf dem Weg zu den hoch gesteckten Zielen haben die Projektpartner bereits zurückgelegt: Die auch für die Bioinformatik in diesem Projekt zuständige Arbeitsgruppe um Staffan Persson vom Max-Planck-Institut für molekulare Pflanzenphysiologie in Potsdam hat rund 80 Mutationslinien isoliert, von denen etwa 20 viel versprechende Kandidaten für die weitere Forschung sind. Letztlich sollen für die vielversprechendsten fünf Rezeptorkinasen und fünf Transkriptionsfaktoren die regulatorischen Funktionen genau bestimmt werden.
Der französische Projektpartner um Elisabeth Jamet (CNRS/UPS, Toulouse) entwickelt für Ackerschmalwand und Zwenke eine Proteindatenbank, die mit der Expressionsdatenbank am MPI in Golm verknüpft ist. Die Partner um Herman Höfte am INRA in Versailles und um Ignacio Zarra an der spanischen Universität in Santiago de Compostela haben ein Transformationsprotokoll für Brachypodium entwickelt und sind an der genetischen und biochemischen Analyse beider Modellpflanzen beteiligt.
Anregungen zum Weiterlesen:
- Ein Interview mit dem Projektleiter von GABI-CELLWALL Staffan Persson finden sie hier: Zellwandsynthese verstehen, um Bioethanol zu gewinnen
- Einen weiteren Artikel zum Projekt GABI-CELLWALL finden Sie in der neuen Ausgabe 1/2010 des GENOMXPRESS.
- Einen TV-Beitrag zur Celluloseproduktion von Pflanzen finden Sie im biotechnologie.tv: Folge 52
- Weiterführende Informationen zum Projekt finden Sie hier: CELLWALL
