Gen-Karte für C4-Photosynthese
Chromatin-Struktur liefert Hinweise zur Genaktivität

Der wohl bekannteste Vertreter der C4-Pflanzen ist Mais. (Bildquelle: © Juraj Berta/Pixabay/CC0)
Die C4-Photosynthese gilt als verbesserte Version der C3-Photosynthese und das macht sie für Pflanzenforscher und -züchter interessant. Die Suche nach den beteiligten Genen ist jedoch schwierig. Eine Lösung für das Problem bietet die DNase-Sequenzierung.
Schwer zu glauben, aber es gab eine Zeit in der Erdgeschichte, in der nicht der Anstieg der CO2-Konzentration in der Atmosphäre ein Problem war, sondern ihr Absinken. Das war natürlich lange, bevor der Mensch auf der Bildfläche erschien. Leidtragenden waren damals die Pflanzen. Denn um Photosynthese zu betreiben, sind sie auf ausreichend CO2 angewiesen. Im Zuge des knapper werdenden Angebots von CO2 stellten einige Pflanzenarten ihre Photosynthese um: Sie veränderten ihre Anatomie und passten die biochemischen Prozesse an. Es war die Geburtsstunde der C4-Photosynthese, die heute von Nutzpflanzen wie Mais, Zuckerrohr und Hirse betrieben wird.
Meister der Photosynthese
Diese sogenannten C4-Pflanzen gelten heute als Photosynthese-Spezialisten und effiziente CO2-Verwerter. Der Clou ist, dass sie anders als C3-Pflanzen, den Betreibern der „regulären“ Photosynthese, die CO2-Fixierung und photochemische Reaktion räumlich trennen.
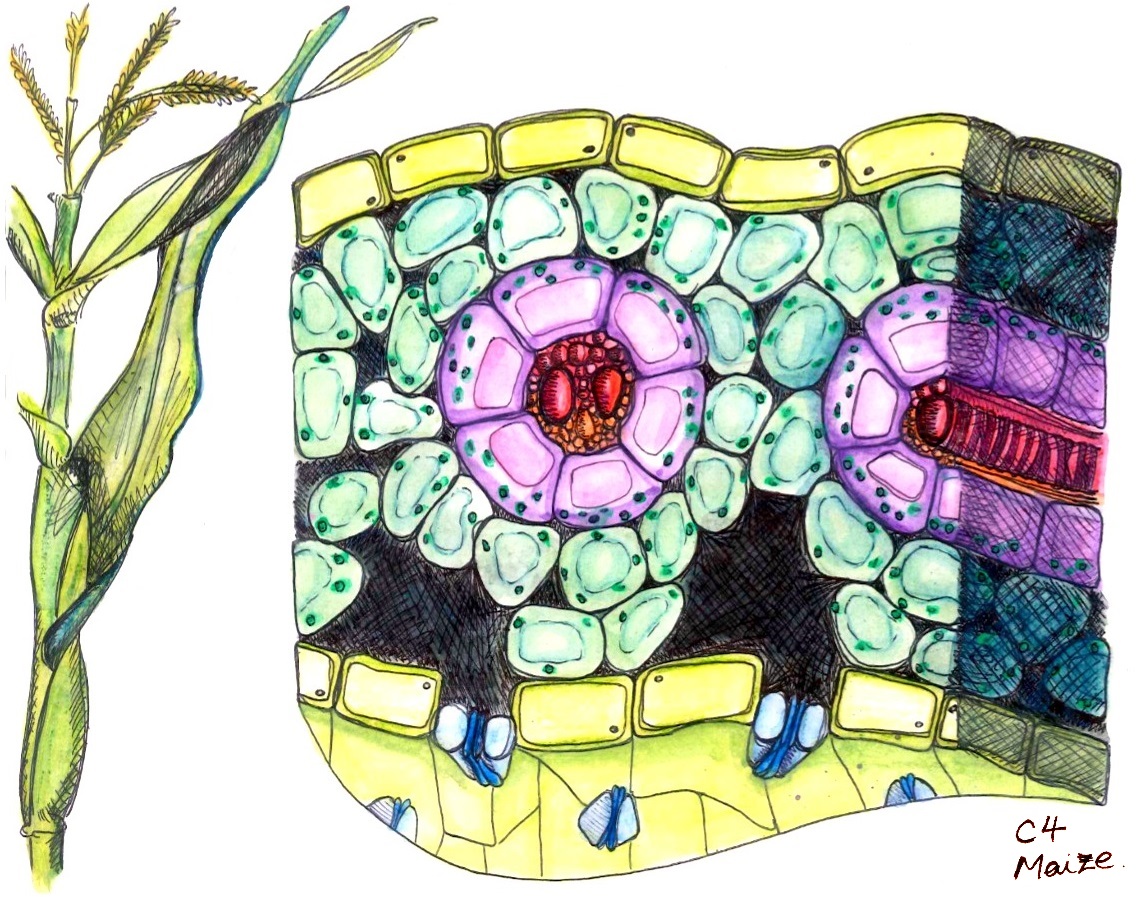
Künstlerische Zeichnung eines Blattquerschnittes aus der C4-Pflanze Mais. In rosa sind die Bündelscheidenzellen dargestellt, die von den Mesophyllzellen (türkis) umgeben sind. Auf der Blattunterseite befinden sich die Spaltöffnungen (blau). Die Leitbündel sind in Rot dargestellt.
Bildquelle: © Ninghui Shi/Wikimedia.org/CC BY-SA 3.0
Eine „Vorfixierung“ des Kohlendioxids erfolgt in den Mesophyllzellen. Durch Aufbringung von Energie wird hier das Kohlenstoffdioxid in der Luft in Form von Oxalacetat regelrecht angereichert, was dann zu einer deutlich höheren Photosyntheserate führt. Oxalactetat enthält vier Kohlenstoffatome, daher stammt der Name C4-Pflanze.
Die übliche Licht-Reaktion und der Calvin-Zyklus finden dann in den mit Chloroplasten vollgepackten Bündelscheidenzellen quasi nebenan statt. Da C4-Pflanzen durch die effektive Vorfixierung des Kohlendioxids ihre Spaltöffnungen für die CO2-Aufnahme nicht voll öffnen müssen, wachsen diese Pflanzen an trockenen, heißen und lichtintensiven Standorten besser als C3-Pflanzen. Daher arbeiten Pflanzenforscher und -züchter an der Vision, die C4-Photosynthese auch in C3-Kulturpflanzen wie Weizen oder Raps einzubauen. Doch dazu bedarf es detaillierter Kenntnisse über die Mechanismen und genetischen Hintergründe.
C4-Pflanzen unter der Lupe
Zwar wurden in den letzten Jahren einige Gene identifiziert, die in den Bündelscheiden- und Mesophyllzellen an der C4-Photosynthese beteiligt sind. Doch bislang fehlte es an einem Überblick, welche Gene insgesamt bei der Photosynthese aktiv und beteiligt sind. Ein internationales Forscherteam hat nun eine Methode entworfen, solche Gene zu identifizieren und sie gleich an drei C4-Pflanzen ausprobiert: Mais (Zea mays), Sorghum bicolor und Kolbenhirse (Setaria italica).
Endonuklease als Tippgeber
Dazu suchten die Forscher in Bündelscheiden- und Mesophyllzellen nach aktiven Genom-Abschnitten. Hierbei handelt es sich um Bereiche, in denen die ansonsten dicht gepackte Struktur des Chromatins geöffnet ist, damit Transkriptionsfaktoren und andere Elemente der Transkriptionsmaschinerie andocken können. Dadurch sind diese Abschnitte auch ungeschützt vor der DNA-spaltenden Nuklease DNase1, die die Forscher in ihren Experimenten einsetzten. In der Fachsprache heißen diese Abschnitte daher auch „DNase1-hypersensitive Stellen (DHS)“, die eingesetzte Methode DNase-Sequenzierung. Insgesamt spürten die Forscher so rund 160.000 solcher DHS-Stellen auf, die zwischen 150 und 15.000 Basenpaare lang waren. Die Zahl der Sequenzen, die als Gene identifiziert wurden und mindestens eine DHS-Stelle besaßen, lag zwischen 20.817 und 27.746.
Die Forscher fanden aber nicht nur heraus, welche genetischen Abschnitte während der Photosynthese aktiv sind, sondern auch wie ihre Genaktivität gesteuert wird. Ein Großteil der für die Transkriptionsfaktoren erreichbaren Abschnitte lag entgegen ihrer Erwartung nicht vor den eigentlich zu aktivierenden Gensequenzen, in der Nähe der Promotoren, sondern mitten in codierenden Abschnitten oder dazwischen.
Gen-Karte gibt Orientierung
Bei den drei untersuchten C4-Pflanzenarten konnten die Forscher eines deutlich erkennen: Die Genregulation hat sich im Laufe der Evolution trotz mancher Überscheidungen überwiegend artspezifisch entwickelt. So hat jede Pflanzenart einen eigenen Weg gefunden, auf C4-Photosynthese umzusteigen. Für die Wissenschaft ist die Arbeit der Forscher insofern wichtig, als sie nun mit der entstandenen Genkarte einen Überblick hat, wo für die C4-Photosynthese relevante Sequenzen mitsamt den genregulatorischen Anknüpfungspunkten liegen. Im nächsten Schritt wird es vor allem darum gehen, die jeweiligen Funktionen dieser Gene zu identifizieren.
Quelle:
Burgess, S.T. et al. (2019): Genome-wide Transcription Factor Binding in Leaves from C3 and C4 Grasses. In: The Plant Cell, (14. August 2019), doi: 10.1105/tpc.19.00078.
Zum Weiterlesen auf Pflanzenforschung.de:
- Vergleichende Photosyntheseforschung
- Die Evolution von C4-Pflanzen vorhersagen
- Wachstumsturbo C4-Photosynthese
Titelbild: Der wohl bekannteste Vertreter der C4-Pflanzen ist Mais. (Bildquelle: © Juraj Berta/Pixabay/CC0)
