Genbanken werden zu digitalen Ressourcenzentren
IPK Gatersleben präsentiert die weltweit erste molekulargenetisch charakterisierte Sammlung

Gerste mit sechszeilig begrannten Ähren. Es ist eine Akzession aus der Genbank in Gatersleben. (Bildquelle: © IPK Gatersleben)
In einer Genbank wird die genetische Vielfalt einer Art gesammelt und so für Forschung und Züchtung nutzbar gemacht. Diese ist vor allem wichtig, um Kulturpflanzen gezielt z. B. an sich verändernde Umweltbedingungen anzupassen. Eines der weltweit umfassendsten Kulturpflanzensortimente ist die bundeszentrale Ex-situ Genbank am Leibniz-Institut für Pflanzengenetik und Kulturpflanzenforschung (IPK Gatersleben). In einer aktuellen Studie beschreibt ein internationales Forscherteam, wie sich die dortige Gerstenkollektion durch systematische molekulargenetische Charakterisierung zu einem bio-digitalen Ressourcenzentrum weiterentwickelt. Erstmals wird so der Zugriff auf interessante Kandidatenpflanzen über eine Datenbankabfrage möglich.
Genbanken sind umfassende Sammlungen von Sorten, Landrassen und wilden Verwandten aller wichtigen Kulturpflanzen. Solche Sammlungen von Keimplasma gibt es für nahezu alle Nutzpflanzen rund um den Globus. Die Dimensionen solcher Genbanken können ein gigantisches Ausmaß annehmen: Die Bestände der großen nationalen Genbanken belaufen sich auf Zehntausende von Mustern pro Art.
Das Problem: Bisher waren die Pflanzen solcher Sammlungen nur ungenügend charakterisiert und historische Aufzeichnungen über die einzelnen Pflanzen auch teilweise verloren gegangen. Höchste Zeit für eine Inventur!
Vollständige Genotypisierung der Sammlung
Wissenschaftler unter Führung des Leibniz-Instituts für Pflanzengenetik und Kulturpflanzenforschung (IPK) in Gatersleben haben dazu fast das gesamte IPK-Weltsortiments der Gerste - insgesamt 22.626 Saatgutmuster - molekulargenetisch analysiert. Die Daten geben unter anderem darüber Aufschluss, wie sich die globale Population der domestizierten Gerste zusammensetzt. Dabei traten auch Redundanzen und Versorgungslücken in einer der größten Genbanken der Welt zu Tage. Das bedeutet, dass nicht alle bekannten genetischen Ressourcen in der bundeszentralen Ex-situ Genbank enthalten sind. Um dies zu testen, wurden auch Proben aus anderen Genbanken in die Studie einbezogen.
Die Forscher nutzten dafür die Methode des „Genotyping-by-Sequencing“ (GBS), ein Verfahren zum Nachweis von Single Nucleotid Polymorphismen (SNP). Beim GBS kommen Restriktionsenzyme zum Einsatz, die das Genom in kleinere Stücke zerschneiden und so seine Komplexität reduzieren. Im Anschluss wird eine PCR durchgeführt, um den Pool der Fragmente zu erhöhen. Die so entstandenen GBS-Bibliotheken werden dann mit Hilfe von Sequenzierungstechnologien der nächsten Generation analysiert.
„Mit unserer Arbeit gelingt es, ein großes Sortiment einer weltweiten Sammlung in einer Genbank molekulargenetisch komplett zu beschreiben – mit anderen Worten, die weltweite natürliche Diversität einer der wichtigsten Kulturarten mit einem Blick zu erfassen“, so Nils Stein vom IPK Gatersleben.
Kandidatenpflanzen lassen sich nun per Datenbanksuche schneller finden
Grundlage der Arbeit bildete zuvor die vollständige Sequenz der Gerstensorte „Morex“, die seit 2017 in hoher Qualität vorliegt und als Referenzgenom diente.
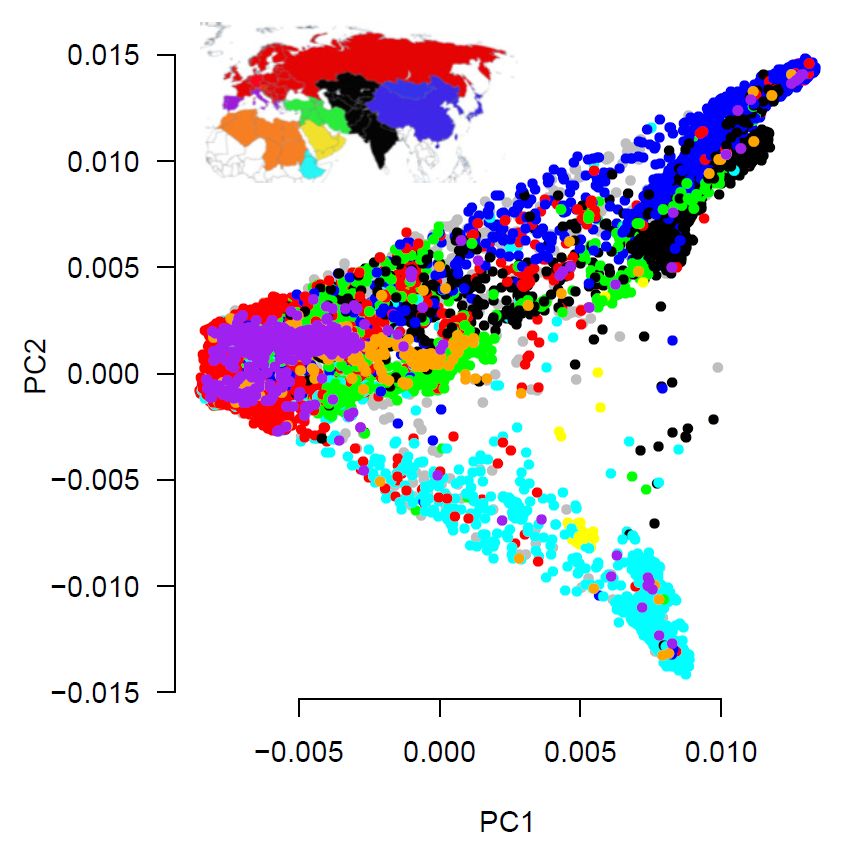
Die genetische Struktur der Sammlung spiegelt die geographische Herkunft wider. Jeder farbige Punkt stellt ein Genbank-Muster dar. Kultursorten und Landrassen, die aus denselben Weltregionen kommen, sind auch in einer Analyse der genetischen Struktur benachbart. Die genetischen Daten können traditionelle Passport-Daten ergänzen und so die Auswahl züchterisch wertvoller alter Landrassen erleichtern.
Bildquelle: © IPK Gatersleben
Mit den über 171.000 in den Gerste-Mustern und Wildformen gefundenen Einzelnukleotidpolymorphismen konnten die Forscher die Genome dieser Pflanzen ausreichend charakterisieren. „Diese Dichte eignet sich im Genom der Gerste mit insgesamt 5 Milliarden Basenpaaren, um kleinste Unterschiede, aber auch Dopplungen zielsicher aufzuspüren“, so Stein.
In ihrer Studie berücksichtigten die Forscher außerdem Proben von 297 Muster aus der Sammlung der Nationalen Nutzpflanzen-Genbank von China, 684 Gerstenmuster der Schweizer National Genebank of Agroscope und 240 GBS-Proben aus einer früheren Studie. „Valide Rückschlüsse auf Herkunft, Verbreitungsgebiet und Ähnlichkeiten der Pflanzen in der Genbank sind so möglich. Durch die Digitalisierung und öffentliche Verfügbarkeit lassen sich die Ergebnisse dank eines modernen Datenbanksystems gezielt abfragen und mit den Passport-Daten der Genbanken, aber auch mit eigenen Forschungs- und Züchtungsdaten kombinieren“, so Martin Mascher vom IPK.
Neuer Schub für die Pflanzenforschung und -züchtung
In Genbanken schlummern immer wertvolle Ressourcen, die jedoch erst durch die „Bio-Digitalisierung“ wie im Fall der IPK-Gerstensammlung vollständig zu erfassen sind.
„Da sich Forschung und Pflanzenzüchtung vermehrt auf wechselnde Umweltbedingungen, aber auch auf veränderten Ressourceneinsatz und Verbraucherinteressen einstellen müssen, stellt die genetische Variabilität eine der wichtigsten Ressourcen der Landwirtschaft dar. Mögliche von Genen aus Landrassen oder verwandten Wildarten beeinflusste Eigenschaften lassen sich jetzt gezielter in modernen Sorten einbringen“, so der an dieser Arbeit ebenfalls beteiligte Frank Ordon vom Julius Kühn-Institut in Quedlinburg.
Quelle:
Millner, S.G. et al. (2018): Genebank genomics highlights the diversity of a global barley collection. In: Nature Genetics, (12. November 2018), https://doi.org/10.1038/s41588-018-0266-x.
Zum Weiterlesen auf Pflanzenforschung.de:
- Genbank erweitert Portfolio Historische Daten liefern Phänotyp-Informationen
- Wilde Gerstenart aus Israel liefert passendes Gen - Trockentolerante Brau-Gerste für deutsche Äcker
- Exomsequenzierung von Gerste - Der extremen Anpassungsfähigkeit der Gerste auf der Spur
- Genetischer und Geografischer Ursprung der Gerste aufgeklärt - Kulturgerste ist zwei Mal unabhängig voneinander gezüchtet worden
- Ur-Gerstenpopulation entdeckt - Moderne Zuchtlinien haben gemeinsamen Vorfahren
Titelbild: Gerste mit sechszeilig begrannten Ähren. Es ist eine Akzession aus der Genbank in Gatersleben. (Bildquelle: © IPK Gatersleben)

