Immer im Wandel: Die 1001 Genome der Arabidopsis thaliana

Ackerschmalwand: Lieblingsmodell der Pflanzenzüchter (Quelle: © iStockphoto.com/pkujiahe).
Vor drei Jahren startete das 1001 Genom-Projekt, um einen Gesamtkatalog der genetischen Vielfalt der Ackerschmalwand zu erstellen. Wissenschaftler stellen jetzt die ersten Ergebnisse vor. Der Sequenzkatalog gibt bereits Hinweise darauf, welche genetischen Eigenschaften die Pflanze so anpassungsfähig machen.
Pflanzen verfügen über eine erstaunliche Anpassungsfähigkeit und können sich zu den unwirtlichsten Regionen der Erde ausbreiten. Die Ackerschmalwand Arabidopsis thaliana ist gewissermaßen ein Paradebeispiel biologischer Wandlungsfähigkeit. Von Nordafrika bis zum Polarkreis erstreckt sich das Verbreitungsgebiet der kleinen Pflanze, der- je nach Standort- kleine, große, schmale, breite, glatte oder gezackte Blätter wachsen.
Obwohl es sich dabei immer um die gleiche Pflanzenart handelt, wurden bereits hunderte unterschiedliche Arabidopsis thaliana-Stämme entdeckt, die sich nicht nur in ihrem Erscheinungsbild, sondern auch anderen Eigenschaften wie beispielsweise Blühverhalten oder Hitze- oder Kälteresistenzen unterscheiden.
Das 1001 Genom-Projekt
Diese Anpassungsfähigkeit spiegelt sich auch in dem Erbgut der Pflanzen wider. Es scheint so, als befinde sich das Arabidopsis-Genom in einem ständigen Umbruch, um mit den Veränderungen der Umwelt Schritt zu halten. Das zeigen die ersten veröffentlichten Ergebnisse des 1001 Genom-Projektes. Das Projekt startete 2008 mit dem Ziel hunderte Genome unterschiedlicher Arabidopsis-Stämme aus Europa, Asien und Afrika zu entziffern, um einen Katalog der genetischen Vielfalt der Ackerschmalwand zu erstellen. Auf diese Weise wollen Wissenschaftler hinter das Geheimnis der enormen genetischen Plastizität und Anpassungsfähigkeit der Pflanze kommen.

Verschiedene Mutanten von Arabidopsis thaliana. (Quelle: © Detlef Weigel / Max-Planck-Institut für Entwicklungsbiologie).
Warum 1001-mal Arabidopsis thaliana?
A. thaliana gehört seit Langem zum Lieblingsmodell der Pflanzenforscher und war die erste Pflanzenart, deren Erbgut im Jahre 2000 komplett und in hoher Qualität entziffert wurde. Sie ist zudem eine enge Verwandte von Kulturpflanzen wie Raps, Blumenkohl und Broccoli. „Mit einem derart umfassenden Datensatz des 1001 Genom-Projektes können wir Genklassen identifizieren, die auch für bestimmte Funktionen in der Pflanzenzüchtung von Interesse sind.“, beschreibt Karl Schmid, Leiter des Fachgebietes Nutzpflanzenbiodiversität und Züchtungsinformatik der Universität Hohenheim, einen Nutzen des Projektes. „Wir können beispielsweise herausfinden, welche Gene die Samengröße von Kulturpflanzen bestimmen.“
Während die Sequenzierung des ersten A. thaliana Genoms noch fast 100 Millionen US $ kostete, wurden im Projekt bereits die neuesten Ultra-Hochdurchsatz-Sequenziermethoden eingesetzt, mit denen komplette Genome für ein Zehntausendstel des Betrages durchsequenziert werden können. Ein großer Vorteil ist dabei das kleine Genom der Pflanze. Ein Winzling im Vergleich zu anderen Nutzpflanzen, wie beispielsweise Mais, Gerste oder Weizen, deren Genome teilweise um ein Zwanzig- bis Hundertfaches größer sind.
Ein Genom im Fluss
In Zusammenarbeit mit Detlef Weigel des Max Planck-Institutes für Entwicklungsbiologie in Tübingen und anderer internationaler Forschungsgruppen untersuchten Karl Schmid und sein Team, in welchem Umfang sich die genetische Grundausstattung der unterschiedlichen Arabidopsis-Stämmen tatsächlich unterscheidet. Sie verglichen die Genome von 80 Arabidopsis-Stämmen aus unterschiedlichen geografischen Regionen und stießen dabei auf eine erstaunliche Variabilität von mehreren Millionen Einzelnukleotid-Polymorphismen. Diese Sequenzabweichungen in jeweils einem Baustein der DNA sind gewissermaßen die Fußspuren der Evolution, denn sie geben einen Hinweis darauf, wie stark der Druck ist, den die Selektion auf manche Gene ausübt. Darüber hinaus enthielt das Erbgut einiger Stämme gleich mehrere Kopien mancher Gene, die in anderen Ackerschmalwand-Stämmen gänzlich fehlten. Auch neue Gene, die in dem ersten sequenzierten Genom von A. thaliana gar nicht vorhanden sind, entdeckten die Forscher.
Mutationen reichern sich in kleine Populationen und Genen für die Anpassungsfähigkeit an
Besonders variabel waren Genfamilien, die bei der Anpassung an Umweltbedingungen eine Rolle spielen, wie beispielsweise Gene der Immunabwehr und Schädlingsresistenz. In einigen Stämmen kleinerer Populationen fanden die Genetiker jedoch auch tausende Mutationen, die nachteilig für die Pflanzen sind, weil sie Gene stilllegten oder die Funktion wichtiger Proteine verändern. Die Forscher halten es jedoch für unwahrscheinlich, dass alle schädlichen Mutationen mit einer Anpassung an neue Lebensbedingungen zusammenhängen. „In kleinen Randpopulationen ist der genetische Drift stärker als in großen. Mit anderen Worten heißt das, die Mutationen mit geringem Fitnesseffekt werden von der Selektion nicht erkannt und entfernt. Ihr Schicksal wird stärker von zufälliger Evolution bestimmt. Dadurch können sich auch nachteilige Mutationen anhäufen, ohne dass dies mit Anpassung zu erklären wäre.“, erklärt Karl Schmid. Dies sei beispielsweise bei den sibirischen Arabidopsis-Populationen der Fall, die erst nach der Eiszeit „ausgewandert“ sind. Aber auch beim Menschen gäbe es dieses Phänomen. So besitzen wir Europäer beispielsweise einen höheren Anteil an nachteiligen Aminosäureabweichungen in unseren Proteinen, als die Schwarzafrikaner aus dem Ursprungsgebiet der Menschheit. Weil unsere Vorfahren als kleine Gründerpopulation auswanderten, konnten sich zufällige, nachteilige Mutationen im Genom der Europäer anreichern.
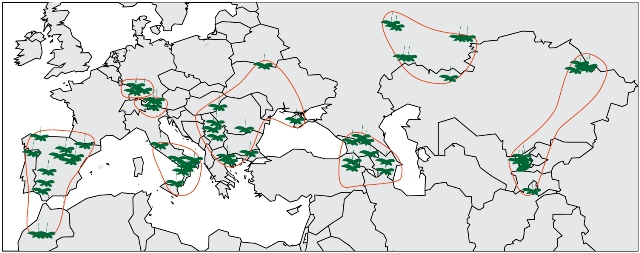
Verbreitungskarte der Arabidopsis thaliana. Die Orte, an denen für die Studien Arabidopsis-Pflanzen entnommen wurden, sind orange markiert (Quelle: © Jun Cao / Max-Planck-Institut für Entwicklungsbiologie).
Viele Mutationen werden in den Zellen kompensiert
Möglicherweise sind jedoch nicht alle Mutationen, die in den 80 Arabidopsis-Genomen entdeckt wurden so schädlich, wie es die Genom-Analysen zunächst vermuten lassen. Das zeigt eine zweite Studie um den Bioinformatiker Gunnar Rätsch des Friedrich-Miescher-Institutes. Um herauszufinden, wie die DNA der Ackerschmalwand im Einzelnen abgelesen wird, nutzten sie neue Analysemethoden für ihre Studie, um auch alternative Genstrukturen zu erfassen. Demzufolge werden viele Mutationen, die die Funktion eines Gens zerstören könnten, während des Ablesens und weiterer Schritte in der Proteinproduktion wieder kompensiert. Obwohl mutiert, entsteht aus dem Gen in solchen Fällen auch weiterhin das korrekte Protein. Untermauern konnten die Wissenschaftler ihre Daten, indem sie die Zwischenstufen der Proteinproduktion, die RNA-Moleküle, aus Knospen, Wurzeln und Keimlingen sequenzierten. Ihre Analysen führten die Wissenschaftler an mehreren hundert Pflanzen durch, die durch Kreuzung aus 19 genetisch besonders variablen Elternstämmen hervorgegangen waren. Die genetische Variabilität der Nachkommen war so stark ausgeprägt, dass bereits in den Keimlingen deutliche Unterschiede der Genaktivität gemessen wurden. Vor allem Gene, die der Pflanze helfen, sich an ihre Umgebung anzupassen, waren in den Nachkommen unterschiedlich reguliert.
1001 neue Forschungsideen
Karl Schmid sieht bereits viele Möglichkeiten, die neuen Daten des Variabilitäts-Kataloges von Arabidopsis zu nutzen. So beispielsweise im Bereich der Epigenetik. In zukünftigen Feldstudien wollen die Pflanzenforscher testen, ob sich Umweltbedingungen auch auf die Genaktivitätsmuster der Pflanzen auswirken, ohne dass deren DNA dabei verändert wird. Das Anheften von Methylierungsgruppen an bestimmte Gene bewirkt beispielsweise, dass diese stillgelegt werden. Kürzlich wurde gezeigt, dass dieser Mechanismus beim sogenannten Imprinting in Arabidopsis eine Rolle spielt, bei dem entweder die Genkopie des Vaters oder der Mutter stumm vererbt wird. Auch Schmids Team will untersuchen, ob durch die Umwelt veränderte Genaktivität in den Pflanzen erblich ist. „In vielen Kulturpflanzen ist das sehr aufwendig, da man an eine Generation pro Jahr gebunden ist. Dort sind auch die Fortschritte in der genetischen Kartierung viel langsamer als in Arabidopsis, bei der die Generationszeit nur 6 Wochen bis drei Monate beträgt.“
Mit den bisherigen Ergebnissen und dem Stand des Projektes sind die beteiligten Wissenschaftler sehr zufrieden. Fast 500 Genome wurden bereits im Rahmen des 1001 Genom-Projektes, an dem mehrere internationale Forschungsinstitute beteiligt sind, sequenziert. Schon im nächsten Jahr will das Projekt mit dem 1000 Genom Projekt des Menschen gleichziehen und die Zahl auf 1001 erhöhen. „Die von uns geschaffene Ressource leistet einen wichtigen Beitrag für die Studien der Evolution und Adaptation von Pflanzen. Das Material ist außerdem komplett öffentlich zugänglich. Jeder Wissenschaftler kann sich zukünftig die Samen der unterschiedlichen Stämme bestellen, um funktionelle Studien für die Pflanzenzüchtung durchzuführen.“
Quellen:
- J. Cao et al. (2011) Whole-genome sequencing of multiple Arabidopsis thaliana populations. Nature Genetics, doi: 10.1038/ng.911 (Link).
- X. Gan et al. (2011) Multiple reference genomes and transcriptomes for Arabidopsis thaliana. Nature, doi: 10.1038/nature10414. (Link).
- K. Schneeberger et al. (2011) Reference-guided assembly of four diverse Arabidopsis thaliana genomes. Proceedings of the National Academy of Sciences of USA 108, 10249-10254, doi: 10.1073/pnas.1107739108. (Link).
Zum Weiterlesen:
- Forschungsbericht der Max Planck-Gesellschaft von 2009
Zum Weiterlesen auf Pflanzenforschung.de:
