Integriertes Datennetzwerk für die Pflanzenforschung
Ein neues Netzwerk priorisiert Daten von Wachstumsregulatoren in Arabidopsis

Die Modellpflanze Arabidopsis thaliana: Im Laufe der Zeit wurden unzählige Daten der Pflanze gesammelt und in unterschiedlichen Datenbanken gespeichert. (Bildquelle: © sinitar - Fotolia.com)
Unglaublich viele molekularbiologische Daten über die Modellpflanze Arabidopsis thaliana sind in themenspezifischen Datenbanken gespeichert. Könnte man diese miteinander vernetzen und in einen analytischen Zusammenhang bringen, so würde sich der Wert der Daten für die Wissenschaft vervielfachen. Denn ein integriertes Netzwerk könnte Überschneidungen von Ergebnissen identifizieren und Hinweise auf wichtige, noch zu identifizierende Genfunktionen liefern. Kürzlich gelang es Wissenschaftlern, ein solches Meta-Netzwerk aus sieben bestehenden Datenquellen zu generieren. Ihr Test zeigte: Das neue Netzwerk ist eine valide Datenbasis für eine effektivere Forschung. Im beschriebenen Fallbeispiel suchten die Forscher nach neuen Wachstumsfaktoren.
Wie kontrolliert ein vielzelliger Organismus sein Wachstum? Generationen von Wissenschaftlern haben sich diese Frage gestellt und daran geforscht. Dank neuer Methoden, die seit einigen Jahren im Hochdurchsatz und immer öfter automatisiert arbeiten, wurde eine riesige Datenbasis geschaffen. Manuell auszuwerten ist diese schon lange nicht mehr. Wichtige Teilantworten liegen bereits vor. Um aus diesen ein Gesamtbild zu generieren, müssen diese Einzeldaten in einen sinnvollen Zusammenhang gesetzt und die Ergebnisse neu interpretiert werden. Gelingt dies, können neue und weiterführende Forschungsfragen gestellt und beantwortet werden.
Da Wissenschaftler nicht über unbegrenzte Ressourcen verfügen, unterstützt eine solche Zusammenführung einen effizienten Umgang mit den verfügbaren finanziellen und personellen Mitteln sowie der vorhandenen Infrastruktur. Können Synergien aus Daten unterschiedlicher Forschergruppen aus unterschiedlichen Projekten gewonnen und sinnvoll kombiniert werden? Das war die zentrale Frage, denen sich die Wissenschaftler in ihrer aktuellen Studie widmeten.
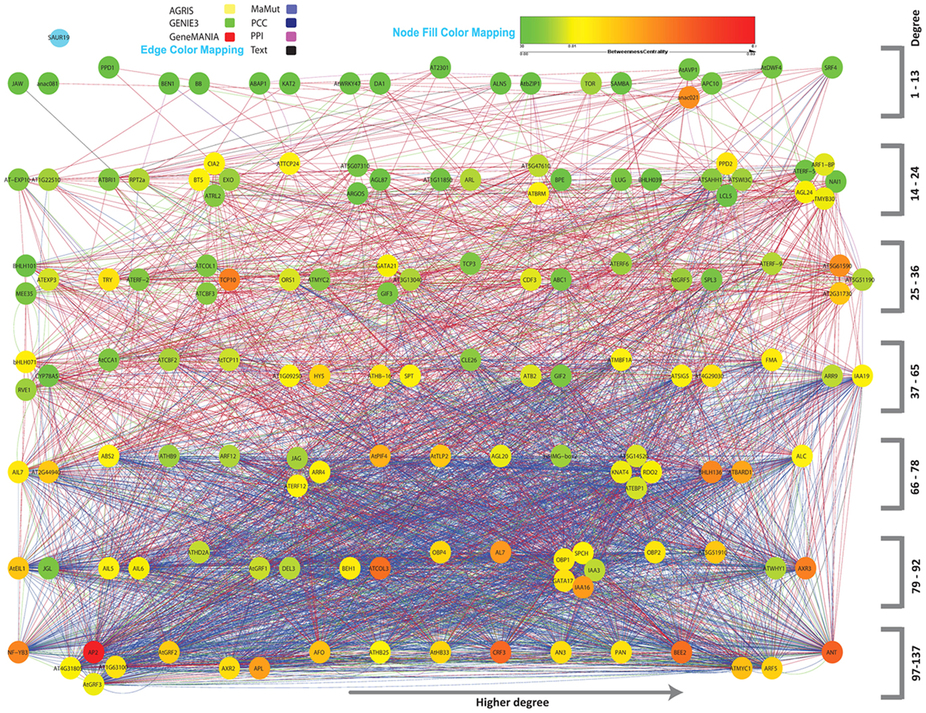
Visualisierung der Ergebnisse: Von den Autoren dieser Studie wurden insgesamt sieben Quellen verwendet, um daraus alle vorhandenen Daten zu wachstumsregulierenden Genen der Pflanze Arabidopsis thaliana zu extrahieren und diese Gene zu priorisieren. Daraus entstand ein Netzwerk.
Bildquelle: © Scientific Reports 12/2015; 5. DOI:10.1038/srep17617; CC BY 4.0
Ein Mammutprojekt
Herausforderungen gibt es viele: Die Daten sind vielseitig und können deshalb nicht ohne weiteres direkt miteinander verglichen werden. In der Pflanzenforschung sind molekulare Daten bisher in verschiedenen, thematisch eingegrenzten Datenbanken hinterlegt: Eine enthält beispielsweise Informationen über unterschiedlich ausgeprägte Gene, wenn Wild-Typen mit transgenen Pflanzen verglichen werden (MaMut), wieder eine andere besteht aus vorhergesagten und experimentell identifizierten Protein-Protein – Interaktionen (PPI).
Zu welchem Grad die Informationen in den Datenbanken bereits verknüpft sind, oder ob die Quellen um Instrumente ergänzt sind, die eine datenspezifische Analyse erlauben, ist ganz unterschiedlich. Die Datenbanken MaMut und PPI liegen auf einer Plattform, CORNET, die auch die Instrumente zur Verfügung stellt, mit denen ein Nutzer die Daten in einen sinnvollen Zusammenhang bringen und so jeweils eine Art Netzwerk aus dem gespeicherten Datenstamm entwickeln kann.
Andere, zum Beispiel die Plattform AGRIS, stellt drei Datenbanken zur Verfügung, die Informationen zur Wachstumsregulation von Arabidopsis thaliana auf unterschiedlichen Ebenen enthalten: Eine enthält bekannte Genregionen, die zweite führt klassifizierte Transkriptionsfaktoren auf, während die dritte bekannte transkriptionale regulatorische Verbindungen enthält.
Diese und weitere Plattformen, in denen Informationen über Pflanzengene, -proteine und ihre Interaktionen auf diverse Art hinterlegt sind, stehen der wissenschaftlichen Gemeinschaft zur Verfügung. Von den Autoren dieser Studie wurden insgesamt sieben Quellen verwendet, um daraus alle vorhandenen Daten zu wachstumsregulierenden Genen zu extrahieren und diese Gene zu priorisieren.
Die zielführende Frage
Ein solches Netzwerk, das all diese Informationen in einen sinnvollen Kontext bringt, gab es bisher in der Pflanzenforschung nicht. Forscher haben die bestehenden Instrumente zur Priorisierung der molekularen Daten weiterentwickelt. Diese sind dafür verantwortlich, genomische Daten aus den zusammengeführten Quellen nach Wichtigkeiten zu ordnen („Gene prioritization“). Sie testeten außerdem, ob sich aus den bekannten, priorisierten Genen auch neue, noch unbekannte Gene ableiten lassen.
Die bisherigen Ergebnisse sind vielversprechend: Trotz der Vielseitigkeit der Daten ist es möglich, aus ihnen ein Netzwerk aufzubauen, das auf eine zu Grunde liegende Fragestellung hin Informationen priorisiert. In diesem Fall wurden alle vorliegenden Daten daraufhin analysiert und eingeordnet, welche Rolle sie für die Regulation des Blattwachstums in Arabidopsis thaliana spielen.
Maschinenbasiertes Lernen schafft Ordnung
Dafür wurden Computer so programmiert, dass sie die Stärke der Zusammenhänge zwischen allen Daten berechnen und diese anschließend priorisieren, also ihnen eine Wichtigkeit für das System „Pflanze“ zuweisen. Damit die Ergebnisse nicht im Chaos münden und nur mit Mühe zu erkennen sind, integrierten die Wissenschaftler ein Visualisierungswerkzeug.

Die eher unscheinbare Pflanze Arabidopsis thaliana dient der Forschung als Modellpflanze. Mehr dazu unter: Ackerschmalwand.
Bildquelle: © Pflanzenforschung.de
Die Ergebnisse der Analysen werden hier bildhaft, sodass die Nutzer die Informationen als ein Netzwerk aus Genen unterschiedlicher Priorität sehen. Unterschiedlich große Punkte stellen Wichtigkeit bzw. eine geringere Bedeutung eines Gens im Netzwerk dar. Deren Zusammenhänge untereinander werden durch unterschiedlich starke Verbindungslinien sichtbar gemacht. Alle bisher in den Datenbanken vorliegenden aber auch zukünftige Daten wurden in die Berechnung einbezogen. Damit ausschließlich valide Daten in die Visualisierung einfließen, wird das Interaktions-Netzwerk in seiner Tiefe analysiert. Zum Beispiel mit Hilfe von Algorithmen, die berechnen, wie sehr ein einzelnes Gen das Netzwerk stabilisiert.
Systembiologie
Die als „gene prioritization“ bezeichnete Methode ist eine Basis der Systembiologie. Diese hat das Ziel, ein möglichst umfassendes Bild eines Organismus zu erarbeiten. Geklärt werden soll wie ein biologisches System funktioniert. Dabei werden alle Ebenen integriert - von der genetischen, über die molekulare, die zelluläre Ebene, bis hin zum Verhalten des Organismus.
Noch basiert biologische Forschung auf Empirie. Von der Aufklärung der molekularen Zusammenhänge erhofft man sich eine stärker rationale Basis legen zu können. Dafür entwickeln Systembiologen nicht nur mathematische Modelle, um Daten miteinander in einen sinnvollen Zusammenhang zu bringen. Vergleichende Forschungsansätze, wie der Versuch mehr und mehr Systemkomponenten wie Klima, Wetter, Geologie, Physik, Chemie oder biologische Interaktionen usw. einzubeziehen, ermöglichen ein mehr und mehr holistisches Bild.
Dabei kommt den Biologen eine Tatsache zu Gute: Alle Organismen sind aus genetisch verwandten Vorfahren entstanden. Beim Vergleich der Daten unterschiedlicher Organismen lassen sich Schnittstellen zwischen diesen finden. Erkenntnisse von Modellorganismen wie der Ackerschmalwand Arabidopsis thaliana lassen sich auf andere Organismen übertragen, die zum Beispiel für die Landwirtschaft wichtig sind.
In der biomedizinischen Forschung ist „gene prioritization“ ein lange erprobtes Instrument. In der Pflanzenforschung steckt sie noch in den Kinderschuhen und hat mit dieser Veröffentlichung einen neuen Anstoß erfahren.
Quelle:
Sabaghian, E., et al. (2015): An integrated network of Arabidopsis growth regulators and its use for gene prioritization. In: Scientific reports 5, 17617, (01. Dezember 2015), doi: 10.1038/srep17617.
Zum Weiterlesen:
- Pflanzeneigenschaften als Schlüssel zum Ökosystem - Drei Millionen gesammelte Daten über Pflanzeneigenschaften helfen bei der Modellierung von Ökosystemen
- Mit Rechenkraft voraus - Datenmengen mit Potential für die Naturwissenschaften
Titelbild: Die Modellpflanze Arabidopsis thaliana: Im Laufe der Zeit wurden unzählige Daten der Pflanze gesammelt und in unterschiedlichen Datenbanken gespeichert. (Bildquelle: © sinitar - Fotolia.com)
