Jenseits der Genetik
Das Methylom als „Hypothesen-Generator“
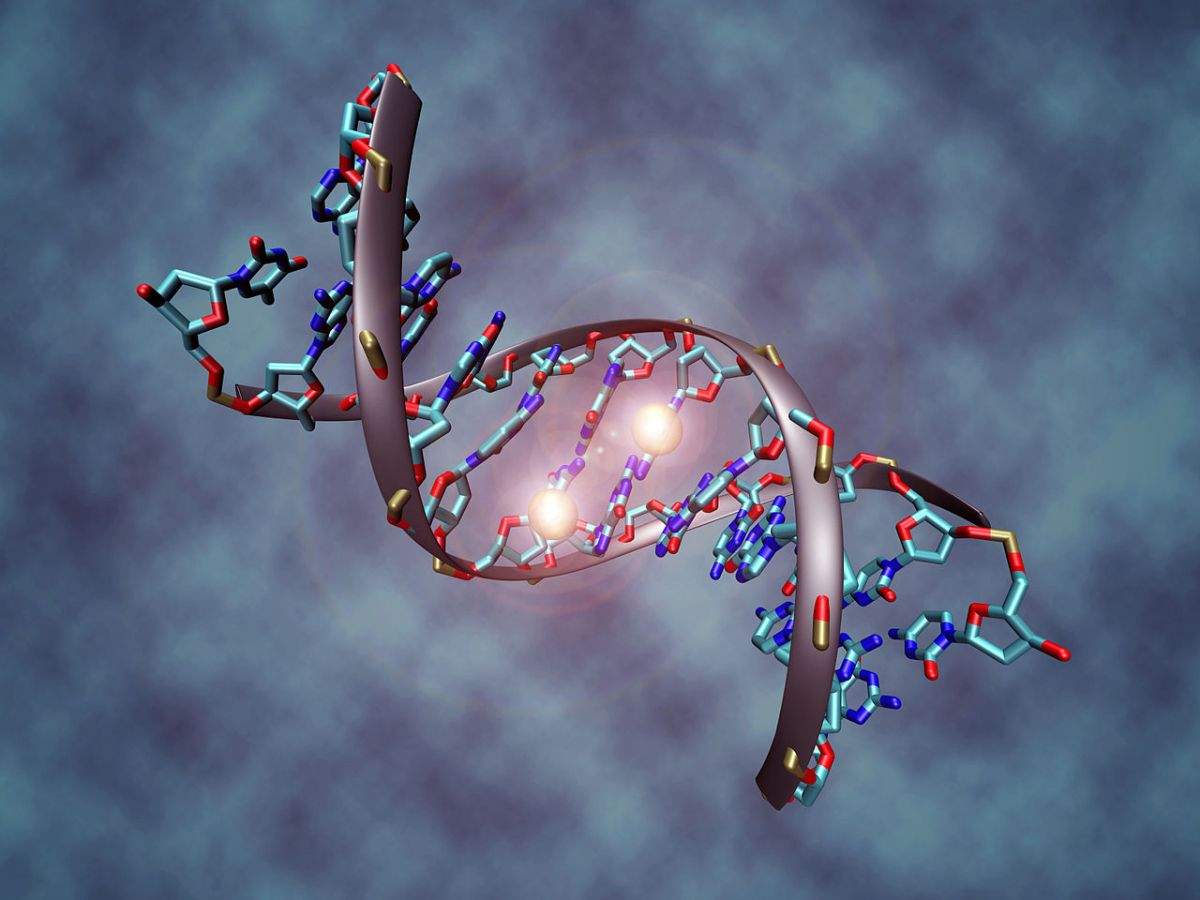
Ein methylierter DNA-Abschnitt zur Veranschaulichung. Die hellen Kugeln symbolisieren zwei Methylgruppen an zwei Cytosin Nukleinbasen. (© Christoph Bock (MPI für Informatik)/ wikimedia.org/ CC BY-SA 3.0)
Sind allein die Gene entscheidend für die Ausprägung von Merkmalen? Seit Jahren reift die Gewissheit, dass dem nicht so ist. Zu verdanken ist diese Einsicht der Epigenetik. Eines ihrer Teilbereiche ist die Erforschung der DNA-Methylierung. Nachdem die Grundzüge dieses Prozesses verstanden sind, macht sich die Pflanzenforschung nun auf, das Wissen über das Methylom in die Erforschung der phänotypischen Vielfalt innerhalb der Pflanzenzwelt zu integrieren.
Viele erinnern sich noch an die Aufbruchsstimmung und den Hype um die Jahrtausendwende, als das menschliche Erbgut im Rahmen des Humangenomprojekts (HGP) erstmals vollständig entziffert wurde. Im selben Zeitraum, wenn auch ohne vergleichbare mediale Aufmerksamkeit, gelang dies auch an Reis (Oryza sativa) und Arabidopsis (Arabidopsis thaliana). Experten sagten das Ende von Krebs, Fettleibigkeit und Schizophrenie sowie eine neue Form der Pflanzenzüchtung voraus. Stets lautete der Tenor: Alles steht in den Genen.
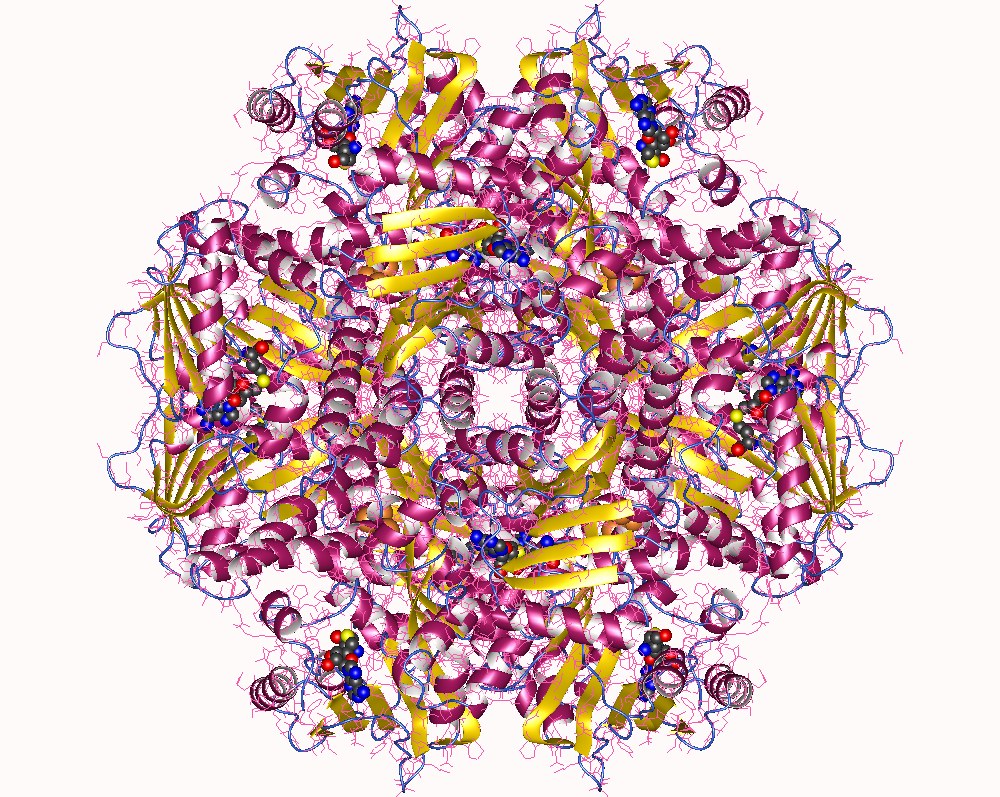
Visualisierung einer Methyltransferase. Enzyme ihrer Art sind damit beschäftigt, Methylgruppen an bestimmte Biomoleküle zu heften. Dies spielt bei bestimmten biochemischen Stoffwechsel- oder Produktionsprozessen eine Rolle oder aber im Zusammenhang mit der epigenetischen Regulation.
Bildquelle: © Astrojan/ wikimedia.org/ CC BY 4.0
Nur wenige Monate nach der Verkündung veröffentlichte das Fachmagazin „Science“ jedoch eine Sonderausgabe zum Thema Epigenetik, in der mit der Vorstellung aufgeräumt wurde, dass viele der Heilsversprechen allein durch das Lesen des genetischen Codes in Erfüllung gehen würden. Zwar spiele das Wissen um die Abfolge der genetischen Bausteine freilich eine Rolle, doch reiche dies längst nicht aus, um zu verstehen, wie genetische Informationen abgerufen und umgesetzt werden. Ohne Ausnahme gelte dies auch für Pflanzen.
Ist das Epigenom der Schlüssel?
Wer daher heute verstehen möchte, warum selbst innerhalb einer einzigen Pflanzenart eine enorme phänotypische Vielfalt möglich ist, muss sich über kurz oder lang mit dem Epigenom beschäftigen. Die Gesamtheit aller epigenetischen Veränderungen und Markierungen des Genoms. Unterschieden wird hier zwischen mehreren Ebenen, die gemeinsam haben, dass sie die Genaktivität beeinflussen, dies aber auf unterschiedliche Weise tun. Eine Ebene ist die DNA-Methylierung. Wie ein internationales Forscherteam nun in einem Kommentar im Fachmagazin „Trends in Plant Science“ unterstreicht, könnte das Wissen über diese dabei helfen, die Hintergründe der genetischen und phänotypischen Vielfalt bei Pflanzen noch besser zu verstehen.
Was passiert bei der Methylierung?
Um nachvollziehen zu können, warum dem so ist, lohnt ein Blick hinter die Kulissen: Bei der DNA-Methylierung handelt es sich um die Anhaftung einer Methylgruppe, ein einfaches Molekül aus einem Kohlenstoffatom und drei Wasserstoffatomen (CH3), an die DNA. Eine Aufgabe, die von speziellen Enzymen, den Methyltransferasen, übernommen wird, die diese vornehmlich an die Nukleinbase Cytosin heften.
Sie tun dies jedoch nicht wahllos, sondern bei Pflanzen am häufigsten dann, wenn auf das Cytosin (C) ein Guanin (G) folgt, die Sequenz also CG lautet. Darüber hinaus oftmals in zwei weiteren Sequenzkontexten, nämlich CHG und CHH, wobei H als Platzhalter für die Nukleinbasen Adenin (A), Thymin (T) oder Cytosin (C) steht. Alle drei Kombinationen tauchen besonders häufig in Promotoren auf, den Sequenzabschnitten, die dem proteincodierenden Abschnitt vorgelagert sind und den Startpunkt der Transkription bilden.
Springende Gene bändigen
Neben ihnen ist eine weitere Gruppe von Genen ebenfalls häufig betroffen, die Transposonen, auch springende Gene genannt. Diesmal erfolgt die Methylierung nicht nur im Zusammenhang mit den oben genannten Sequenzkontexten, sondern im Grunde entlang des gesamten Genkörpers. Vermutlich handelt es sich hier um eine Art zellulären Schutzmechanismus vor fremdem Erbgut.
Welche Folgen hat nun eine DNA-Methylierung? Um es kurz zu machen: Für sich allein genommen ist eine Methylgruppe nicht in der Lage, die Transkription eines Gens zu beeinflussen. Dies übernehmen Proteine, die von der Methylgruppe angelockt werden, an ihr andocken und den Promotor bzw. bei den Transposonen das komplette Gen stilllegen.
Eine weitere Ebene der epigenetischen Regulation: Die Visualisierung zeigt, wie sich die DNA der Taufliege (bunt) um eine Gruppe von Histonen (dunkelrot) windet. Überall dort, wo die DNA besonders dicht und eng um die Histone gewickelt ist, ist eine Transkription unmöglich. Um diese zu erlauben, muss sich das Gebilde erst einmal auflockern.
Bildquelle: © Sponk/ Wikimedia.org/ CC0
Methylierung und Selektion
Während in den letzten zwei Jahrzehnten die Grundzüge der DNA-Methylierung immer klarer wurden, begannen Forscher, über ihre Rolle in der Evolution nachzudenken. Zwar sind Methylierungen jederzeit reversibel (Demethylierung), aber dennoch über Generationen vererbbar. Sie verändern den Phänotyp und beeinflussen auf diese Weise die evolutionäre Fitness. Theoretisch wären damit die Voraussetzungen erfüllt, zum Spielball der Evolution zu werden.
Transkriptom und Methylom im Abgleich
2016 bestätigte dies die Studie eines internationalen Forscherteams mit Beteiligung aus Deutschland. Im Zuge des „1001 Genomes“-Projekts, bei dem es vorrangig um die Sequenzierung von mindestens 1.001 Arabidopsis-Genomen ging, wurden neben der DNA auch das Transkriptom und das Methylom sequenziert. Letzteres umfasst die Gesamtheit aller Methylierungen der DNA. Der Abgleich mit dem Transkriptom ergab, dass rund 25 % aller Gene unterschiedlich methyliert waren und sich dies im Transkriptom widerspiegelte. Zudem korrelierten bestimmte Methylierungsmuster mit bestimmten Standorten und Klimata. Klare Indizien für die Forscher, dass DNA-Methylierungen bei der Evolution eine Rolle spielen müssen, auf jeden Fall bei der Anpassung.
Verstehen, was vor sich geht
Die Analysen zeigten außerdem, dass das Methylom und somit auch das Epigenom (die Gesamtheit aller epigenetischen Veränderungen) nicht nur reguliert, sondern selbst reguliert wird. „Es gibt Gene, die die Epigenome in Pflanzen kontrollieren. Verschiedene Varianten dieser Gene sind in der Lage, das Epigenom so zu verändern, dass es der Pflanze hilft, in einer bestimmten Umgebung besser zu überleben. Es ist ein enormer Hypothesengenerator, wenn es darum geht, zu verstehen, was in der natürlichen Welt vor sich geht“, erklärt Joe Ecker, ein Beteiligten der Studie.
Genom, Transkriptom und Methylom im Akkord
Dies sehen die Forscher im aktuellen Fall genauso. „Das Genom, das Transkriptom und das Methylom machen es in Kombination möglich, die Hintergründe der Anpassung und der Evolution systematisch zu erforschen“, ist dort zu lesen. Das Ende der Fahnenstange ist damit jedoch noch lange nicht in Sicht. Schließlich dürfen die anderen Ebenen der epigenetischen Regulation nicht vergessen werden. Ein Beispiel sind Histonmodifikationen, die kurz gesagt über den Verpackungsgrad der DNA entscheiden und so die Transkription ebenfalls beeinflussen.

Carl von Linné war einer der Ersten, der sich im 18. Jahrhundert mit epigenetischen Veränderungen beschäftigte. Natürlich ohne es zu wissen. Seine Beobachtungen machte er damals am Echten Leinkraut (Linaria vulgaris).
Bildquelle: © Frank Vincentz/ Wikimedia.org/ CC BY-SA 3.0
Die Pflanzenforschung als Hotspot der Epigenetik?
Wir befinden uns somit am Anfang eines neuen Kapitels der Forschung, in dem epigenetische Veränderungen, Variationen und ihre Vererbung als neue Hauptakteure hinzukommen. Ihre Eigenschaften und Fähigkeiten besser kennen zu lernen, wird viele Forscher in den nächsten Jahren beschäftigen. Auch wird sich zeigen, ob und wie sich die Ergebnisse von Arabidopsis auf andere Organismen übertragen lassen werden.
Die Chancen, dass die Pflanzenforschung in den nächsten Jahren zu einem Hotspot der Epigenetik anvancieren wird, stehen jedoch nicht schlecht. „Pflanzen sind Meister der epigenetischen Regulation. Alle wichtigen epigenetischen Mechanismen werden von Pflanzen genutzt und oft in einem Maße verfeinert, das in anderen Reichen keine Parallele findet“, begründen dies Marjori Matzke und Ortrun Mittelsten Scheid vom Gregor Mendel Institute of Molecular Plant Biology in Wien.
Eine neue Informationsquelle für Pflanzenforscher und -züchter
Dass das Methylom zu einer wichtigen Informationsquelle für die Pflanzenforschung werden dürfte, ist also sehr wahrscheinlich. Auch Pflanzenzüchter würden davon profitieren, erklärt Ecker: „Züchter könnten epigenetische Informationen verwenden, um Eigenschaften einer Pflanze auszuwählen, genauso wie sie genetische Informationen nutzen. So nützlich individuelle Gene auch sind - die Vorstellung, dass man nach epigenetischen Varianten auswählen kann, ist etwas, dem man Aufmerksamkeit schenken sollte.“
Schon heute sind Züchtungsfirmen in der ganzen Welt eng in wissenschaftliche Netzwerke eingebunden, die sich mit der Aufklärung epigenetischer Prozesse beschäftigen. Diese Erkenntnisse in Züchtungsprogramme zu integrieren, stellt eine große Herausforderung dar, da noch nicht alle Gesetzmäßigkeiten verstanden sind. Wie es vor 150 Jahren mit den Mendelschen Regeln gelang, der Züchtung ein rationales Fundament zu geben, so arbeiten Pflanzenforscher daran, ein solches für die Epigenetik zu schaffen.
Quellen:
- Lang, Z. et al. (2016): The 1001 Arabidopsis DNA Methylomes: An Important Resource for Studying Natural Genetic, Epigenetic, and Phenotypic Variation. In: Trends in Plant Science, doi.org/10.1016/j.tplants.2016.09.001.
- Kawakatsu, T. et al. (2016): Epigenomic Diversity in a Global Collection of Arabidopsis thaliana Accessions. In: Cell, Vol. 166, doi.org/10.1016/j.cell.2016.06.044.
Weiterlesen auf Pflanzenforschung.de:
Titelbild: Ein methylierter DNA-Abschnitt zur Veranschaulichung. Die hellen Kugeln symbolisieren zwei Methylgruppen an zwei Cytosin Nukleinbasen. (© Christoph Bock (MPI für Informatik)/ wikimedia.org/ CC BY-SA 3.0)
