Klaus Mayer jongliert Milliarden von DNA-Bausteinen
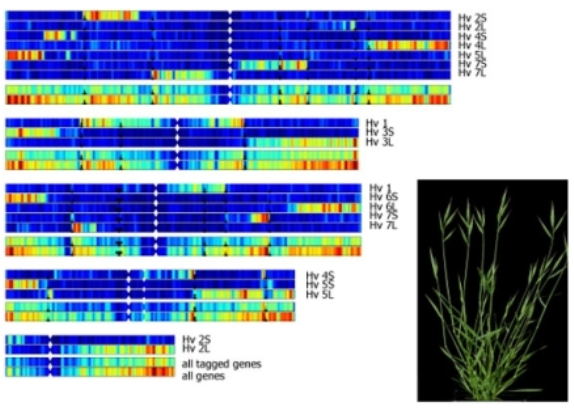
Ein Vergleich zwischen ähnlichen Genombereichen von Brachypodium und Gerste half bei der Entschlüsselung des gewaltigen Gerstengenoms. (Quelle: © Helmholtz-Zentrum München)
Es gibt noch viel zu tun für Klaus Mayer und seine Arbeitsgruppe: Die Gesamtheit aller Gene in einer Zelle ist noch nicht von allen Pflanzen und Tieren bekannt. Mayer und sein Team verfolgen ein Ziel: Die Etablierung einer synergetischen Plattform für verschiedensten Tier- und Pflanzenvarietäten.
"Die Entschlüsselung des Genoms von Nutzpflanzen wie Gerste und Weizen ist eine der großen Herausforderungen für die Bioinformatiker", erklärt Klaus Mayer, Gruppenleiter der 12-köpfigen Arbeitsgruppe "Pflanzliche Genomforschung" am Institut für Bioinformatik und Systembiologie am Helmholtz-Zentrum München. "Das Genom des Weizens ist mit seinen 15 Gigabasen - das entspricht 15 Milliarden DNA-Bausteinen - ungefähr fünf mal so groß wie das menschliche Genom", so der Forscher. Auch die Sequenz der Gerste lässt sich nicht lumpen: Immerhin hat es 1,8 mal mehr an Inhalt zu bieten als das menschliche Pendant.
Doch Größe ist nicht alles, die Nutzpflanzen protzen mit großen Genomen, in denen sich aber nicht viel mehr Gene befinden. "Über 90% des Genoms sind angefüllt mit ähnlichen, sich wiederholenden Elementen," erklärt Mayer. Diese Überreste der Evolution machen das Zusammensetzen der sequenzierten Einzelteile enorm schwierig, denn die Forscher haben wenig Anhaltspunkte, die Abschnitte in der richtigen Reihenfolge aneinander zu fügen oder dem richtigen Chromosom zuzuordnen.
Die Kunst, die richtige Reihenfolge zu finden
Die Entschlüsselung des 280 Megabasen großen Genoms von Brachypodium distachyon, der Modellpflanze für Getreide, ist den Münchnern in Kooperation mit internationalen Forschungseinrichtungen erfolgreich gelungen und stellt die Grundlage zur Erforschung von weiteren Nutzpflanzen dar. Die Sequenzierarbeit dafür wurde vom kalifornischen Joint Genome Institute durchgeführt und dauerte dank moderner Sequenziermaschinen lediglich zwei Wochen.
"Das Zusammenfügen des Sequenz-Puzzles am Computer und die Analyse von Genen und der Vergleich mit anderen Genomen nahm viel mehr Zeit in Anspruch," erinnert sich Mayer. Die Wissenschaftler können nun aus den Sequenzen folgern, dass mehrere zehntausend genetische Beziehungen zwischen Brachypodium, Reis und Weizen existieren.

Klaus Mayer arbeitet am Institut für Bioinformatik und Systembiologie des Helmholtz-Zentrums München.
Bildquelle: © Helmholtz-Zentrum München
Diese wissenschaftliche Leistung brachte eine Publikation im hochgeschätzten Wissenschaftsmagazin Nature (11. Februar 2009, Bd. 463, S. 763-767) ein. Die Sequenzen machen die Wissenschaftler frei zugänglich - eine Information, die für die Sequenzierung und Analyse von Gerste und Weizen wesentlich ist und von der mittelfristig auch Züchter profitieren. "Diese können beispielsweise gezielter Pflanzen züchten, die dem Klimawandel trotzen, resistenter gegen Krankheiten oder auf trockenen Böden wachsen", sagt der 46?jährige Wissenschaftler, der über Umwege zur Forschung gelangte.
Nach einer Ausbildung zum Chemielaborant machte er sein Abitur auf dem zweiten Bildungsweg und studierte anschließend Biologie in München. "Meinen einzigen Auslandsaufenthalt hatte ich während meiner Promotion. Den verbrachte ich als Bayer im schwäbischen Tübingen," erzählt der Biologe mit einem Schmunzeln. Den Neckar verließ er vor zwölf Jahren wieder und ist seitdem am Institut für Bioinformatik und Systembiologie, zuerst am Max?Planck Institut für Biochemie und dann am Helmholtz?Zentrum München.
Weitere Sequenzierungsprojekte von Nutzpflanzen und Tieren
Die Computer in Mayers Arbeitsgruppe werden selten abgeschaltet. In diversen vom Bundesforschungsministerium geförderten Projekten sind sie derzeit im Einsatz. So laufen die Analysen von Hühner- und Rindergenomen, die im Rahmen von SynBreed unterstützt werden. Darüber hinaus ist Mayer an der Sequenzierung des Gerstengenoms unter dem Dach der BMBF-Initiative "Genomanalyse im biologischen System Pflanze" (GABI) beteiligt. "Dieses Projekt ist bahnbrechend. Zusammen mit unseren Kollaborationspartnern haben wir einen Weg gefunden, um mit cleverer bioinformatischer Analyse einen Großteil der Gerstengene im Genom anzuordnen", sagt der Familienvater und Bergliebhaber Mayer.
Mit einer gehörigen Portion Ausdauer kommt Mayer seinem Ziel, möglichst viele Genome zu entziffern, Schritt für Schritt näher: Seine Vision ist es, eine synergetische Plattform für die verschiedensten Arten und Varietäten zu etablieren die erlauben, die molekularen Unterschiede zwischen verschiedenen Pflanzen und Tieren zu untersuchen.
- Der Beitrag wurde durch eine Kooperation mit der Redaktion von Biotechnologie.de unseren Lesern zur Verfügung gestellt. Den Beitrag können Sie dort in der Rubrik "Menschen" (unter "Aktuelles") lesen.
- Informationen zum Projekt "GABI-BARLEX" (Physische Karte des Gerstengenoms) finden Sie hier.
