Mehr als nur wertloser Schrott
Genom Editierung mit CRISPR/Cas erleichtert Annotation nichtcodierender DNA

Lange Zeit galten nichtcodierende DNA-Sequenzen als ohne jede Bedeutung. In den letzten Jahren wandelt sich jedoch das Bild. (Bildquelle: © MIKI Yoshihito/ wikimedia.org/ CC BY-SA 2.0)
Dass nichtcodierende DNA keineswegs so wertlos und überflüssig ist, wie die Bezeichnung „Junk-DNA“ aus den 1970er Jahren suggeriert, ist mittlerweile anerkannt. Schwierig gestaltet sich jedoch nach wie vor die Annotation, die funktionelle Zuordnung. Wie jüngste Beispiele nun zeigen, ließe sich diese Problem mit Hilfe von CRISPR/Cas lösen.
Lange Zeit galt sie als überflüssig, wertlos und ohne jede Funktion. Die Rede ist von jenen Teilen der DNA, die nicht für Proteine codieren. Sprich Sequenzen, die keine genetischen Informationen zur Herstellung von Proteinen (Proteinbiosynthese) enthalten. Beim Menschen machen diese in der Gesamtheit etwa 98 % des gesamten Erbguts aus und auch bei Pflanzen kann der Anteil beträchtlich sein. Es gehört zu den Verdiensten des internationalen ENCODE-Forschungsprojekts, dass sich die Wahrnehmung auch in der breiten Öffentlichkeit gewandelt hat. 2012 veröffentlichte das Konsortium eine Studie, in der endgültig mit der Vorstellung aufgeräumt wurde, es handele sich bei der nichtcodierenden DNA und der entsprechenden RNA schlichtweg um wertlosen Schrott bzw. „Junk“. Vielmehr ist davon auszugehen, dass ein Teil von ihr wichtige, wenn nicht sogar überlebenswichtige Funktionen ausübt, ein Steuerungsapparat gigantischen Ausmaßes ist.
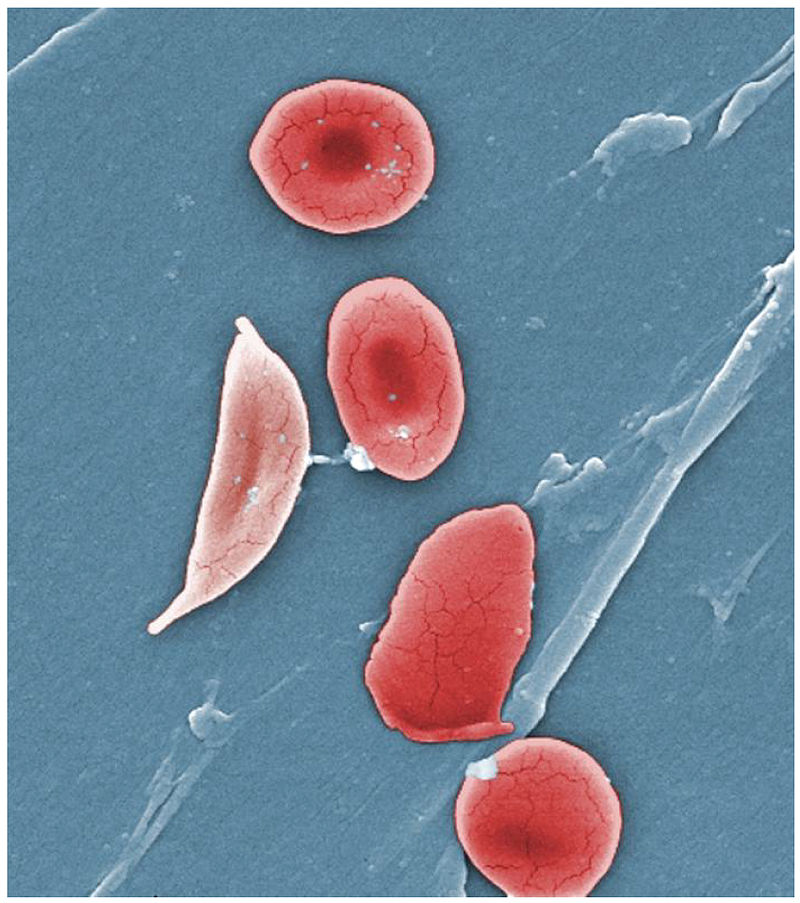
Dank des Rückgriffs auf die CRISPR/Cas-Methode gelang es Forschern 2015, die krankheitsauslösende Mutation ausfindig zu machen, die dazu führt, dass aus runden, weichen und beweglichen Blutkörperchen harte und spitze Sicheln werden.
Bildquelle: © OpenStax College/ Wikimedia.org/ CC BY 3.0
Die bekannte Unbekannte
In der Wissenschaft war die nichtcodierende DNA zum Zeitpunkt der Veröffentlichung längst kein unbeschriebenes Blatt mehr, insbesondere regulatorische Funktionen bereits bekannt. So zum Beispiel von Sequenzen, die die Transkription von Genen ermöglichen (Promotoren) bzw. verstärken (Enhancer) und zu den sogenannten Transkriptionsfaktoren gezählt werden. Nicht zu vergessen die Introns: Nichtcodierende Einschübe innerhalb eines Gens, die zwar transkribiert werden, danach aber durch spezielle Komplexe aus RNA oder Proteinen wieder herausgeschnitten werden (Spleißen). Sie haben Anteil daran, dass aus einem Gen verschiedene Proteinvarianten hervorgehen können.
Grenzen bisheriger Methoden
Nach wie vor basiere jedoch vieles von dem, was wir über die nichtcodierende DNA wissen oder zu wissen glauben, entweder auf Hypothesen oder auf Beobachtungen in äußerst künstlichen Umgebungen, kritisieren die beiden Genomforscher Jason Wright und Neville Sanjana nun in einer aktuellen Publikation. Ihnen fehlt es an Methoden zur Untersuchung der Funktion und Rolle besagter Sequenzen in biologischen Prozessen in ihrem natürlichen Kontext. Zwar seien Verfahren wie die MPRA-Methode (Massively Parallel Reporter Assay) verfügbar, die in der Lage sind, ausschließlich die transkriptionsregulatorischen Elemente aus einer DNA-Probe herauszufiltern, jedoch stoßen diese an Grenzen.
Zwei großes Mankos sind, dass bei der MPRA-Methode episomale Vektoren eingesetzt werden (ringförmige, bakterielle DNA-Moleküle), wodurch die Transkription unter anderen Bedingungen abläuft als in echt, u. a. außerhalb des endogenen Chromatin-Kontextes. Zum anderen, dass durch die Beschränkung auf mRNA-Moleküle posttranskriptionale oder etwaige Feedback-Mechanismen nicht berücksichtigt werden können.
Ist CRISPR/Cas die Lösung?
Wie jüngere Studien nun gezeigt haben, könnte eine Lösung dieses Problems in der Nutzung von CRISPR/Cas liegen. Jenem vielseitig einsetzbaren bakteriellen RNA-Protein-Komplex, mit dem DNA punktgenau geschnitten und verändert werden kann. Die Möglichkeiten, die sich aus dem neuen Anwendungsfeld ergeben, sind weitreichend und können mitunter Leben retten.

Nichtcodierende DNA beeinflusst auch die Blütenbildung, wie z. B. beim Blauen Gänseblümchen (Brachyscome iberidifolia).
Bildquelle: © CSRIO/ Wikimedia.org/ CC BY 3.0
Wie verschiedene genomweite Assoziationsstudien (GWAS) ergeben haben, ist die Ursache für eine Reihe von menschlichen Erkrankungen nicht in der codierenden, sondern nichtcodierenden DNA zu suchen. Ein populäres Beispiel ist die Sichelzellenanämie, eine erbliche Erkrankung des Blutes, die die roten Blutkörperchen (Erythrozyten) betrifft, genau genommen den blutroten Farbstoff, das Hämoglobin.
Die Suche nach der Nadel im Heuhaufen
Schuld ist eine Mutation des β-Globin-Gens (BCL11A), wodurch dieses in seiner Aktivität gehemmt ist. Die Folge ist, dass an der sechsten Aminosäurenposition des Hämoglobin-Proteinkomplexes statt der Aminosäure Glutamat (HbA) die Aminosäure Valin (HbS) vorzufinden ist. Eine kleine Veränderung, die jedoch dazu führt, dass die runden, glatten, weichen und beweglichen Blutkörperchen spitz, hart und sichelförmig werden. Das Problem bestand lange darin, dass der exakte Ort der Mutation, von dem nur bekannt war, dass er innerhalb eines Introns liegen muss, zum Leid vieler Betroffener nicht exakt lokalisiert werden konnte.
2015 gelang der Durchbruch
Mit Hilfe von CRISPR/Cas gelang es den Forschern, den Auslöser punktgenau dingfest zu machen. Hierfür setzten sie speziell programmierte CRISPR/Cas-Systeme auf drei infrage kommende BCL11A-Enhancer-Sequenzen in jungen und gesunden Erythrozyten an. Ihre Aufgabe bestand darin, diese nacheinander durch eine gezielt herbeigeführte Mutation auszuschalten, um so herauszufinden, welcher der drei Enhancer die Aktivität des BCL11A Gens entscheidend beeinflusst.

Keine Regel ohne Ausnahme: Der Zwerg-Wasserschlauch (Utricularia gibba) ist dafür bekannt, nur wenig nichtcodierende DNA zu besitzen. Ihr Anteil am Genom liegt gerade einmal bei 3 %.
Bildquelle: © Alex Popovkin/ Wikimedia.org/ CC BY 2.0
Tatsächlich gelang den Forschern der entscheidende Treffer. Verantwortlich für die krankheitsbildende Veränderung des Hämoglobins ist eine Mutation in der Sequenz eines GATA1-Transkriptionsfaktors. Ein Meilenstein für die Entwicklung neuer Therapieansätze und Medikamente und für die Übertragung des Ansatzes auf andere biologische Fragen.
Nichtcodierende DNA bei Pflanzen
Auch in der Pflanzenforschung steht die Erforschung der nichtcodierenden DNA im Fokus. Besondere Aufmerksamkeit genießt in diesem Kontext die Gruppe der langen nichtcodierenden RNA (im Englischen: long non-coding RNA, kurz lncRNA). Hierbei handelt es sich um RNA-Sequenzen bestehend aus mindestens 200 Nukleotiden, die das Transkript der nichtcodierenden DNA darstellen.
Wie verschiedene Studien unabhängig voneinander bestätigt haben, spielen diese in einer Reihe von biologischen Prozessen und Mechanismen bei Pflanzen eine wichtige Rolle. So stehen elementare Prozesse wie die Transkriptionsregulation, die Methylierung, das Spleißen, die Entwicklung der Zellkernstruktur, also essentielle Lebens- und Entwicklungsprozesse, bis hin zur Blütenbildung unter ihrem Einfluss.
Den Kinderschuhen entwachsen
Noch aber steckt die Forschung zu diesem Thema in den Kinderschuhen. Selbst bei den klassischen Modellsystemen der Pflanzenforschung ist nach wie vor vieles unklar. Diesen Rückstand möglichst schnell aufzuholen, war und ist das Anliegen von Michal Szczesniak. Ende 2015 ging die von ihm entwickelte Online-Plattform „CANTATAdb“ ans Netz, die Forschern freien Zugang zu den lncRNA-Daten von 10 Modellorganismen und weiteren wichtigen Pflanzen der Forschung gewährt: u. a. Arabidopsis thaliana, Reis (Oryza sativa), Kartoffel (Solanum tuberosum), Wein (Vitis vinifera), Soja (Glycine max), die Grünalge Chlamoyomonas reinhardtii sowie Mais (Zea mays).
Um eine Facette reicher
Wright und Sanjana sind zuversichtlich, dass in naher Zukunft weitere Enthüllungen über bislang verborgene Funktionen jener Sequenzen folgen werden, die lange Zeit als Schrott abgetan wurden. Vor diesem Hintergrund empfehlen sie, die Technologie dahingehend weiterzuentwickeln, möglichst bald auch längere Abschnitte auf Anhieb zu analysieren. Derzeit sei man darauf beschränkt, Abschnitte in der Länge von maximal einem Megabasenpaar (1 Millionen Basenpaare) zu untersuchen. Um die mitunter weitreichenden Verflechtungen und Interaktionen besser erforschen zu können, bedarf es eines größeren, bis hin zum genomweiten Blickwinkel. Unabhängig davon steht jedoch schon jetzt fest, dass das ohnehin schon breite Anwendungspotenzial von CRISPR/Cas nun um eine interessante und wichtige Facette reicher ist.
Quellen:
- Wright, J., Sanjana, N. (2016): CRISPR Screens to Discover Functional Noncoding Elements. In: Trends in Genetics (13. Juli 2015), doi.org/10.1016/j.tig.2016.06.004
- Chekanova, J. (2015): Long non-coding RNA and their functions in plants. In: Plant Biology, Vol. 27, doi:10.1016/j.pbi.2015.08.003
- Szczesniak, M. et al. (2015): CANTATAdb: A Collection of Plant Long Non-Coding RNAs. In: Plant & Cell Physiology, Vol. 57, doi:10.1093/pcp/pcv201
Zum Weiterlesen auf Pflanzenforschung.de:
Titelbild: Lange Zeit galten nichtcodierende DNA-Sequenzen als ohne jede Bedeutung. In den letzten Jahren wandelt sich jedoch das Bild. (Bildquelle: © MIKI Yoshihito/ wikimedia.org/ CC BY-SA 2.0)
