RNA-Editierung wird modernisiert
Forscher setzen auf SNAP-Protein statt Cas13b
Modell des ADAR-Eznyms, das aus dem Nukleosid Adenosin das Nukleosid Inosin macht. (© Emw/Wikimedia.org/CC BY-SA 3.0)
Die CRISPR/Cas9-Technologie hat bekanntermaßen eine Revolution bei der gezielten DNA-Veränderung ausgelöst. Auch auf RNA-Ebene rücken solche Eingriffe nun in greifbare Nähe. Sie eröffnen neue Möglichkeiten, von denen auch die Pflanzenzüchtung profitieren dürfte.
Gezielt bestimmte Abschnitte des Erbguts verändern zu können, war lange der Traum der Genetiker. Mit der CRISPR/Cas9-Technologie scheint er wahr geworden zu sein. Diese enzymatische Genschere erlaubt präzise Eingriffe in die DNA. Doch die Diskussion ist noch nicht abgeschlossen, ob auch vereinzelt unbeabsichtigte DNA-Veränderungen auftreten (sogenannte „off-target“-Effekte). So gesehen könnten DNA-Eingriffe einen Nachteil haben: Wenn ungewollte Veränderungen stattfinden, dann sind sie von Dauer.
Anders ist das bei Eingriffen in die mRNA: sie führen zwar auch zu veränderten Proteinen, aber das Erbgut bleibt unverändert. Da mRNAs in der Zelle relativ schnell wieder abgebaut werden, tritt der ursprüngliche Zustand nach einer gewissen Zeit wieder von alleine ein. RNA-Veränderungen sind somit immer reversibel.
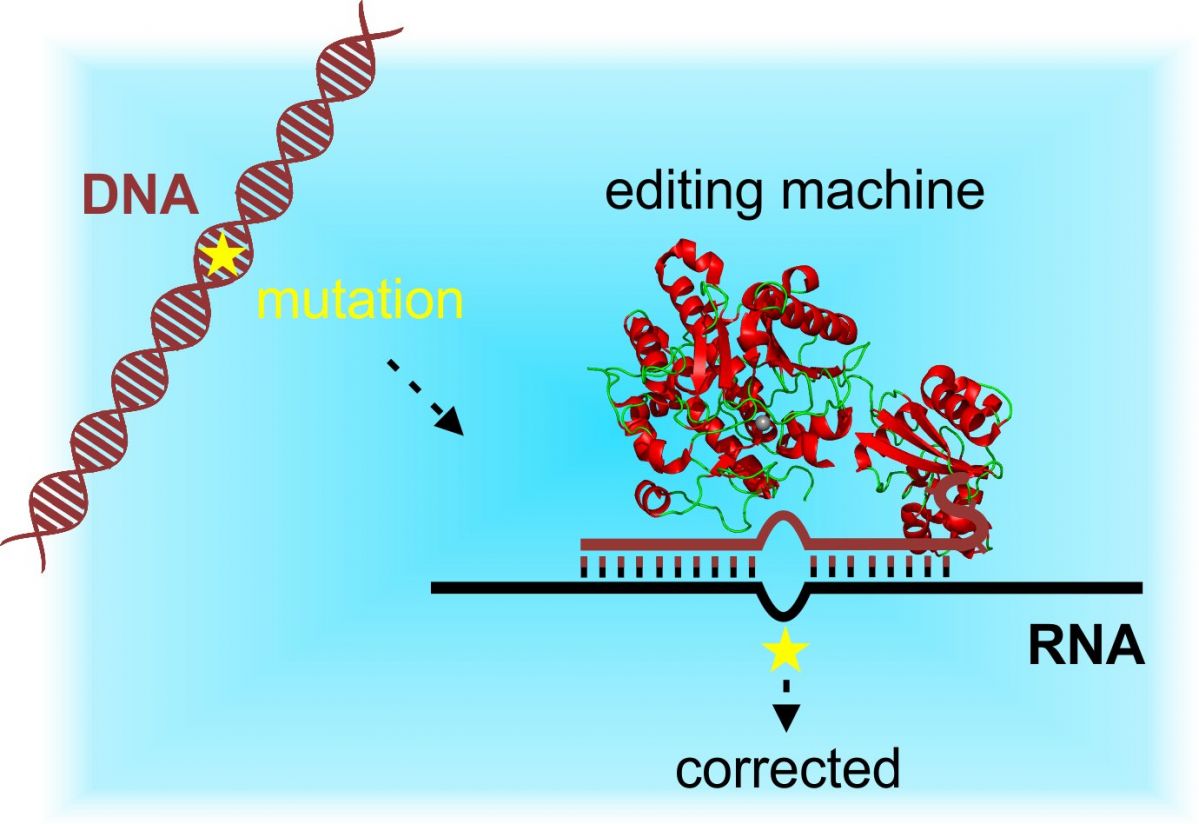
Schema zum Konzept der zielgerichteten RNA-Editierung: Die Arbeitsgruppe von Professor Thorsten Stafforst entwickelte eine Editierungsmaschine, die es ermöglicht, krankheitsverursachende Mutationen durch RNA-Editierung zu reparieren.
Bildquelle: Abbildung: Paul Vogel
Zwei neue elegante RNA-Editierungsmethoden haben Forscher im Jahr 2017 vorgestellt. Anstatt die RNA-Sequenz zu schneiden, bewirkt eine chemische Modifikation durch ein Enzym die gezielte Umwandlung von einer Base in eine andere. Beide Ansätze verwendeten Variationen des CRISPR-Cas-Systems. Jetzt haben Tübinger Forscher gemeinsam mit US-Wissenschaftlern eine weitere Methode vorgestellt, um einzelne Basen der RNA zu verändern. Sie nutzt das SNAP-Protein und arbeitet zuverlässiger als die älteren Ansätze.
Aus A wird G
„Unsere Methode der RNA-Editierung beruht auf einem Proteinkonstrukt, das mit Hilfe einer kleinen Führungs-RNA zum RNA-Zielmolekül gelangt und dort einzelne Basen umwandelt. So wird die Bauanleitung des kodierten Proteins umgeschrieben“, erklärt Thorsten Stafforst von der Universität Tübingen. Gemein ist der neuen und den älteren Methoden der Einsatz von Enzymen, die Aminogruppen von einem Molekül entfernen (Deaminasen). Das Enzym namens ADAR (adenosine deaminases acting on RNA) macht aus dem Nukleosid Adenosin das Nukleosid Inosin. Bei der Proteinbiosynthese wird Inosin als Guanosin gelesen. Aus einem A in der DNA wird somit ein G in der präRNA für die Translation. So lassen sich neue Start- oder Stop-Codons erzeugen, das Splicing verändern oder die Struktur des Proteinprodukts beeinflussen.
Damit die Deaminase ADAR nicht wahllos Veränderungen in der RNA vornimmt, haben die Forscher sie mit der Führungs-RNA (guide-RNA, kurz: gRNA) kovalent verbunden. Deren Sequenz ist komplementär zu der zu editierenden RNA-Sequenz und sorgt dafür, dass ADAR hauptsächlich an der gewünschten Stelle der RNA aktiv wird. Um das Konstrukt aus ADAR und gRNA zu verbinden, kam die SNAP-Tag-Technologie zum Einsatz. Das sogenannte SNAP-Protein ist ein genetisches Standardwerkzeug, das dazu entwickelt worden ist, einen Liganden kovalent an ein bestimmtes Zielprotein zu binden.
Wildtyp präziser als hyperaktive Mutanten
In ihrer Studie nutzten die Forscher zwei unterschiedliche ADAR-Enzyme, jeweils als Wildtyp-Form und als hyperaktive Mutante. Die Wildtyp-Form bewirkte bei 40 bis 80 Prozent des Zielproteins die geplante Veränderung, die hyperaktive Variante bei 65 bis 90 Prozent. Etwa drei Tage nach Versuchsbeginn nahm der Anteil der editierten Proteine langsam ab – mutmaßlich weil die Konzentration des ADAR-gRNA-Komplexes infolge der Zellteilung sank. Die hyperaktive Form erzielte ihre halbmaximale Wirkung mit einem Zwölftel der beim Wildtyp dafür nötigen Konzentration.
Als Stärke ihres Systems stellen die Forscher heraus, dass es kaum zu Veränderungen bei anderen Proteinen außer dem Zielprotein kommt, wie die Forscher mittels der sogenannten Gesamt-Transkriptom-Shotgun-Sequenzierung analysierten. Dies sei auf die SNAP-Methode zurückzuführen. Lediglich bei Adenosin-reichen Tripletts, insbesondere bei CAA, gab es einige nicht gezielte Editierungen, wie die Auswertung des Transkripts des GAPDH-Gens zeigte. Durch chemische Veränderungen der gRNA konnte das Team die ungewollten Editierungen für dieses Transkript auf unter zehn Prozent senken. Im Vergleich mit dem Wildtyp wiesen die hyperaktiven ADAR-Enzyme deutlich erhöhte Fehlerquoten auf. Eine der Wildtyp-Formen erwies sich als besonders präzise, da von den 344 in verschiedenen Transkripten betroffenen Nicht-Ziel-Sequenzen nur in 20 Fällen mehr als 50 Prozent des Proteinprodukts editiert waren.
#####2#####Methode zum Patent angemeldet
Trotz dieser Fehler-Editierungen konnten die Forscher bei den betroffenen Zellen keine Probleme feststellen. Morphologie, Verdoppelungsrate und die Transkriptmenge wiesen keine Unterschiede auf. Allerdings ist dem Team bewusst, dass die Präzision der Methode noch verbessert werden muss. Einen ersten Ansatz verfolgte die Studie bereits: Senkten die Forscher die Expression des Wildtyp-ADARs um den Faktor 25, sanken die fehlerhaften Veränderungen deutlich. Weitere Verbesserungen erhoffen sich die Biochemiker künftig von chemischen Modifikationen der gRNA und der ADAR-Enzyme. „In Zukunft möchten wir auf die Proteinkonstrukte völlig verzichten und stattdessen auf die natürlich vorkommenden Enzyme zurückgreifen, um diese für die gerichtete RNA-Editierung einzuspannen“, so Stafforst zu seinen Plänen für das weitere Vorgehen. Auf die neue Methode hat das Forscherteam ein Patent angemeldet.
Im Vergleich mit dem 2017 vorgestellten System CRISPR/Cas13b hat sich der neue Ansatz bereits als doppelt so effizient und deutlich präziser erwiesen. Das deutet darauf hin, dass der Zielmechanismus mittels gRNA wesentlichen Einfluss auf die Präzision hat. Für medizinische Anwendungen sei zudem der humane Ursprung des SNAP-Proteins von Vorteil.
Mögliche Anwendungen in der Pflanzenzüchtung
In die Pflanzenzüchtung hat die RNA-Editierung noch nicht Einzug gehalten. Entsprechende Enzyme, die einzelne Nukleoside umwandeln, sind allerdings auch in den Plastiden von Samenpflanzen und den Mitochondrien höherer Landpflanzen bekannt. Es ist daher zu erwarten, dass ähnlich wie bei CRISPR/Cas auch die neuen Ansätze der RNA-Editierung für die Pflanzenzüchtung adaptiert werden können. Denkbar wäre zum Beispiel, bestimmte Abwehrmechanismen der Pflanze auf diese Weise erst beim Befall durch einen Schädling zu aktivieren. Zunächst aber dürfte noch einige Arbeit nötig sein, um die Genauigkeit dieses Präzisionswerkzeugs noch weiter zu verbessern.
Quelle:
Vogel, P. et al. (2018): Efficient and precise editing of endogenous transcripts with SNAP-tagged ADARs. In: Nature Methods, 15, 535-538, (2. Juli 2018), doi: 10.1038/s41592-018-0017-z.
Zum Weiterlesen auf Pflanzenforschung.de:
- CRISPR/Cas: Baukasten zur Editierung von Genomen wird erweitert
- CRISPR/Cas9 in bester Gesellschaft - C2c2 ergänzt Cas9 auf RNA-Ebene
- Wie CRISPR/Cas funktioniert - Eine einfache Technologie verbessert die Effizienz der Genom Editierung
Titelbild: Modell des ADAR-Eznyms, das aus dem Nukleosid Adenosin das Nukleosid Inosin macht. (© Emw/Wikimedia.org/CC BY-SA 3.0)
