Tool mit neuen Möglichkeiten: MapMan4
So lassen sich Proteinfunktionen schneller identifizieren

MapMan4 zeigte, dass in der parasitären Pflanze Cuscuta - auch Teufelszwirn genannt (hier: rot) - viele klassische Gene der Photosynthese nicht mehr vorhanden sind. (Bildquelle: © arenysam / Fotolia.com)
„Rohe Daten“ wie Sequenzinformationen von Proteinen und Genen sagen oft nur wenig über die Funktionen dieser Biomoleküle im Stoffwechsel aus. Mit „Mercator“ und „MapMan4“ haben Wissenschaftler Plattformen geschaffen, mit denen Forscher ihre „omics“-Daten in einen biologischen Kontext bringen können.
Seit die Genomsequenzierung für viele Labore erschwinglich geworden ist, schnellt die Zahl der sequenzierten Pflanzengenome in die Höhe. Doch der genetische Code verrät nur wenig über die tatsächlich ablaufenden Stoffwechselvorgänge in einer Pflanze. Um herauszufinden, welches Gen für welches Protein codiert und welche Funktionen dieses Protein in der Pflanze übernimmt, haben Wissenschaftler zwei Programme erschaffen.
Das Prozedere beginnt mit dem Programm „Mercator“. Hier gibt der Forscher seine Gen- bzw. Proteinsequenzen ein und erhält nach wenigen Minuten eine Klassifizierung dieser Sequenzen. Mit dieser Klassifizierung lässt sich beispielsweise auch herausfinden, ob Gene oder Genfamilien im untersuchten Organismus verloren gegangen sind oder sich im Erbgut ausgebreitet haben.
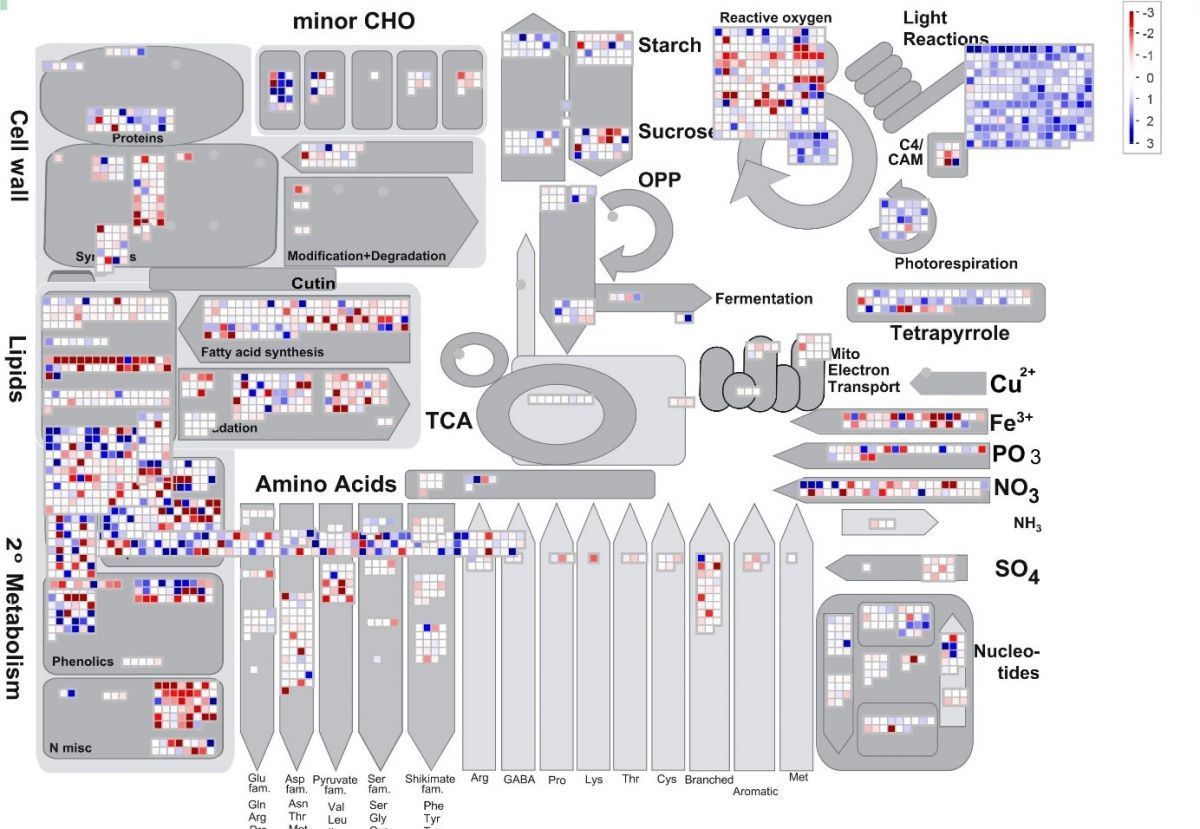
In MapMan4 werden Omics-Daten zu bestimmten Stoffwechselbereichen zugeordnet. Die blauen und roten Farbtöne spiegeln den veränderten Expressionszustand eines Gens wider.
Bildquelle: © MapMan4 / Dr. Björn Usadel
„Unsere Untersuchungen dazu haben wir an der Pflanze Cuscuta durchgeführt“, erklärt Björn Usadel vom Forschungszentrum Jülich und der RWTH Aachen Universität. Die gelbe, parasitäre Pflanze betreibt weniger Photosynthese als ihre Artgenossen. „Hier haben wir ganz deutlich gesehen, dass einige Gene, die normalerweise in den Plastiden eine wichtige Rolle spielen, gar nicht mehr vorhanden sind.“
Omics-Daten auf Stoffwechselwegen visualisieren
In MapMan4 lassen sich die eigenen Omics-Daten auf Stoffwechselwegen visualisieren. Unter omics-Daten versteht man alle in einer Zelle zu einem bestimmten Zeitpunkt identifizierten Moleküle einer bestimmten Substanzklasse, etwa Proteine, mRNA oder Metaboliten. „Das Programm klassifiziert, ob beispielsweise ein untersuchtes Gen für einen Transkriptionsfaktor codiert, der eine Rolle bei Kältestress spielt“, so Usadel weiter. Das entsprechende Gen wird dann anschaulich in funktionellen Stoffwechsel- und Prozesskarten dargestellt und ermöglicht dem Wissenschaftler so eine leichte Einordnung der Gen- bzw. Proteinfunktion im gesamten Stoffwechselkontext der Pflanze.
„Wissenschaftler können ihre meist komplexen Daten mit MapMan4 visualisieren und auswerten. Bei einer Transkriptionsstudie wäre beispielsweise unmittelbar ersichtlich, dass die Glykolyse in diesem Moment in der Zelle hochgefahren und einige Transkriptionsfaktoren im Gegenzug gedrosselt wurden“, erklärt Usadel. Doch vollständig ist das System noch lange nicht, denn noch längst sind nicht alle Genfunktionen bekannt.
Universell nutzbar
„Mit MapMan haben wir eine Struktur geschaffen, die theoretisch für alle Pflanzen funktioniert, jedenfalls in den konservierten Bereichen“, so Usadel. Aber spezielle Metaboliten – beispielsweise von Medizinalpflanzen – deckt das Programm im Regelfall bisher noch nicht ab.
Hohe Qualität durch Präzision
Die Modellpflanze Arabidopsis thaliana ist derzeit die am besten untersuchte Pflanze. Trotzdem ist für etwa 20 Prozent der Gene noch keine Funktion bekannt. Auch die bekannten Gene sind noch nicht alle in MapMan4, der aktuellsten Version von MapMan, verfügbar. Das hat einen guten Grund: „Wir warten mit der Aufnahme eines Gens so lange, bis wir ihm eine sehr präzise Funktion zuordnen können“, erklärt Usadel. Diese Zuordnung basiert in den allermeisten Fällen auf der händischen Durchsicht von aktuellen, wissenschaftlichen Veröffentlichungen.

Selbst in der am besten bisher untersuchten Pflanze Arabidopsis thaliana sind 20 % der Gene hinsichtlich ihrer Funktion noch unbekannt. In MapMan4 werden Gene erst hinzugefügt, wenn ihre Rolle im Stoffwechsel detailliert bekannt ist.
Bildquelle: © GABI
Ontologie „in Eimern“
Eine Ontologie ist eine Definition von Kategorien, Eigenschaften und Beziehungen zwischen Daten und Einheiten. Das kann man beispielsweise auch für Gene und ihre Funktionen machen. Gene, die funktional zusammengehören, sind bei MapMan4 in sogenannten „Bins“ (dt. Eimern) zusammengefasst. Die einzelnen Bins sind mit Nummern versehen, in manchen Fällen existieren auch Sub-Bins. Am Ende der Ontologie steht die spezifische Funktion eines Gens oder des entsprechenden Proteins – beispielsweise ein Transkriptionsfaktor für Gen XY.
„Anhand des Bin-Systems kann sich der Nutzer leicht orientieren, auf welcher Annotationsebene er sich gerade befindet“, erklärt Usadel. Im Gegensatz zu anderen Programmen bietet MapMan4 ein Komplettpaket an. Usadel dazu: „Man beginnt mit den Proteinsequenzen einer Pflanze. Für diese erstellt Mercator dann eine Klassifizierung und einen Annotationstext, der die Funktion der Proteine beschreibt. Schließlich kann man in MapMan quantitative Genexpressionsdaten auf funktionellen Stoffwechsel- und Regulationswege darstellen und untersuchen.“
Gegenüber den Vorgängerversionen sei MapMan4 sehr viel systematischer und feiner in der Analyse geworden, berichtet Usadel – weitere Entwicklungen laufen.
Frei nutzbar sind Mapman und Mercator hier: http://mapman.gabipd.org/de und https://plabipd.de/portal/mercator4
Quelle:
Schwacke, R. et al. (2019): MapMan4: a refined protein classification and annotation framework applicable to multi-omics data analysis. In: Molecular Plant, (Januar 2019), doi: 10.1016/j.molp.2019.01.003.
Zum Weiterlesen auf Pflanzenforschung.de:
- Exomsequenzierung von Gerste - Der extremen Anpassungsfähigkeit der Gerste auf der Spur
- Genomsequenzierung für Eilige - Neue Methode spart Zeit und Geld
- Reiche Ernte - 10 Jahre Arabidopsis Genom Sequenzierung
- DNA-Sequenzierungsmethoden - Motoren der Genomforschung
Titelbild: MapMan4 zeigte, dass in der parasitären Pflanze Cuscuta - auch Teufelszwirn genannt (hier: rot) - viele klassische Gene der Photosynthese nicht mehr vorhanden sind. (Bildquelle: © arenysam / Fotolia.com)
