Vernetzte Pflanzenproteine
Am Beispiel von Arabidopsis erstellen Forscher das erste Gen-Interaktiom einer Pflanze (Quelle: © Qgroom / wikimedia.org; gemeinfrei).
Erstes Pflanzen-Interaktom erstellt. Das Netzwerk der zellulären Wechselwirkungen zwischen allen Proteinen einer Pflanze gleicht einer Landkarte mit Verkehrsknotenpunkten. Wenige stark vernetzte Proteine stabilisieren das System, sind aber gleichzeitig Ziele von Krankheitserregern.
Das gesamte Genom der Modellpflanze Arabidopsis thaliana ist bereits seit elf Jahren bekannt. Auf deren Basis konnten weitreichende genetische sowie molekulare Kenntnisse aufgedeckt werden. Dennoch sind im intensiv erforschten Modellsystem Arabidopsis die Funktionen von mehr als 60 Prozent aller Protein-kodierenden Gene bis heute nicht aufgeklärt.
Als wichtigen Schritt zu einem besseren und komplexeren Verständnis hat eine Gruppe internationaler Forscher nun eine Karte erstellt, die das Netzwerk der Protein-Protein-Wechselwirkungen im Modellsystem aufzeigt. Bisher gab es solche als Interaktom bezeichneten Karten nur für Bakterien, einzellige Pilze und einige wenige tierische Organismen. In einem weiteren Schritt wurden Pflanzenproteine den Proteinen von typischen Pflanzenschädlingen ausgesetzt. Die Reaktion der pflanzlichen Eiweiße auf die pathogenen Proteinen wurde in diese Netzwerkbetrachtungen integriert.
Stark vernetzte Proteine erhalten den zellulären Prozess
In einer Serie von Veröffentlichungen im Fachmagazin „Science“ haben die Wissenschaftler ihre Ergebnisse vorgestellt. Die Forschergruppe „Arabidopsis Interactome Mapping Consortium“ (AIMC) testete die Wechselwirkungen von 8.000 Arabidopsis thaliana-Proteinen in Hefezellen, was etwa 30 Prozent der Protein-kodierenden Gene entspricht. Die Hefezellen waren so verändert, dass sie sich teilten, sowie zwei Proteine zusammen reagierten. Auf diese Weise konnte der Beweis für eine Protein-zu-Protein-Reaktion optisch beobachtet werden. Die Forscher kombinierten alle möglichen Paarungen untereinander. Am Ende identifizierten sie für 2.700 Proteine insgesamt 6200 Protein-zu-Protein-Wechselwirkungen. Die mit diesen Ergebnissen erstellte Netzwerkkarte zeigte, dass sich um eine relativ kleine Zahl von Proteinen regelrechte Knotenpunkte von Wechselwirkungen gruppierten. Deutlich mehr Proteine zeigten nur wenige Interaktionen mit anderen Proteinen. Diese stark vernetzten Proteine stellen zentrale Knotenpunkte im Interaktom dar. Werden sie entfernt, bricht nach Meinung der Forscher das Netzwerk zusammen. Würden dagegen wenig vernetzte Proteine entfernt, erwarten die Forscher keine signifikanten Störungen von wichtigen zellulären Prozessen.
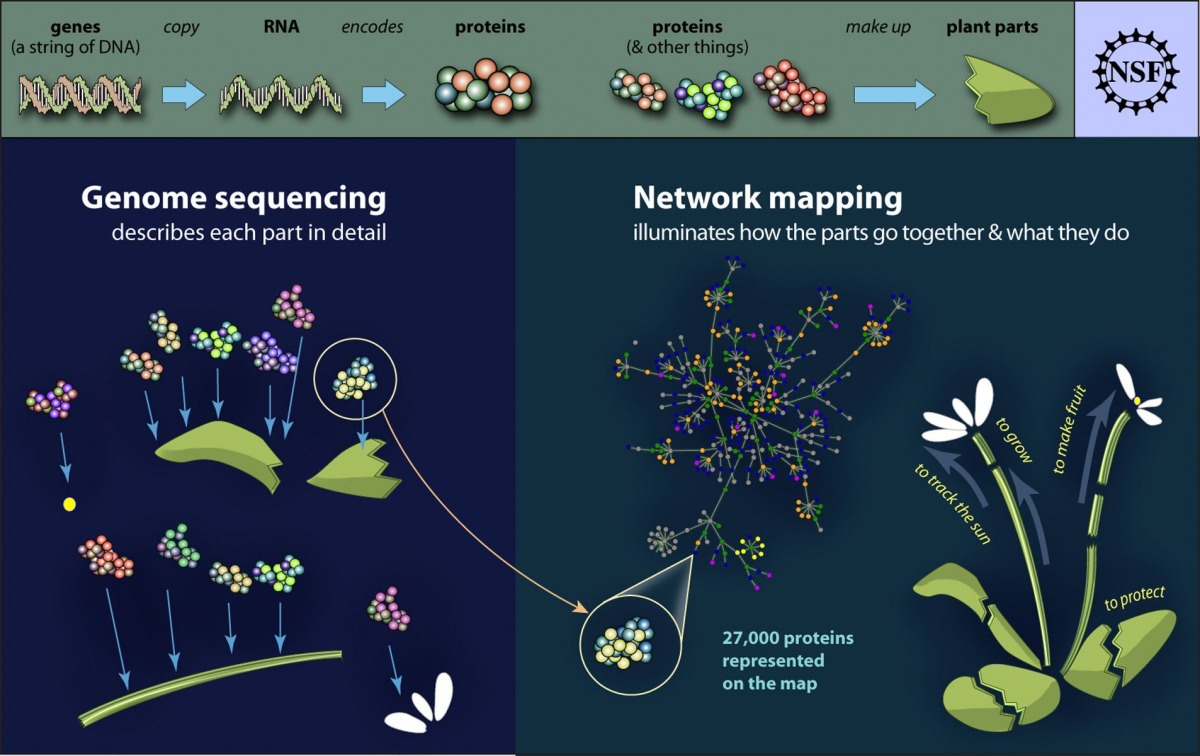
Proteine sind die Bausteine des Lebens. Die Interaktionsmuster der Proteine untereinander ermöglichen die Vielfalt in der Natur. Hochpräzise steuern sie sämtliche Lebensprozesse der Organismen. Die Grundlage für die vollständige Erfassung des Interaktoms ist die genetische Information. Das Interaktom ist eine Voraussetzung für die umfassende Aufklärung der Gen- bzw. Proteinfunktionen. Damit legte die Modelpflanze Arabisopsis thaliana erneut einen wichtigen Grundstein für zukünftige Forschungs- und Entwicklungsarbeiten an Kulturpflanzen.
Bildquelle: © Zina Deretsky, National Science Foundation, USA
Welchen Einfluss hatten Protein-Netzwerke während der Evolution?
Einige der im Genom von Arabidopsis in mehrfacher Kopie vorliegenden Gene (duplizierte Gene) wurden stammesgeschichtlich vor bereits 700 Millionen Jahren vervielfältigt. Bisher waren Wissenschaftler davon ausgegangen, dass solche Duplikate über einen so langen Zeitraum stark mutieren und sich in Struktur und Funktion weit von den anderen Duplikaten entfernen. Die AIMC-Forscher stellten nun jedoch fest, dass die meisten Genduplikate mit den gleichen Proteinen interagieren und mit diesen funktionale Komplexe bilden. Sie schließen daraus, dass das Protein-Netzwerk auf noch unbekannte Weise die unterschiedliche Entwicklung der Duplikate einschränkt.
Krankheitserreger greifen die Knotenpunkte an
In einer weiteren Studie wollten Forscher wissen, wie das Interaktom reagiert, wenn Krankheitserreger in die Zelle eindringen. Sie paarten daher Proteine des Arabidopsis-Interaktoms mit Effektor-Proteinen der Krankheitserreger. Effektor-Proteine greifen speziell das „Immunsystem“ der Pflanze an und führen so letztlich zur Infektion. Manche Krankheitserreger produzieren 20 bis 30 solcher Effektoren. In der aktuellen Studie wurden Effektorproteine des Bakteriums Pseudomonas syringae sowie des Eipilzes Hyaloperonospora arabidopsis mit den Pflanzenproteinen gepaart. Die Forscher fanden ihre Theorie bestätigt, dass Effektorproteine beider Krankheitserreger nicht etwa die speziellen Proteine des „Immunsystems“ angreifen, sondern die stark vernetzten Proteine im Interaktom. Auf diese Weise wird das „Immunsystem“ der Pflanze instabil und eine Infektion kann sich ausbreiten.
Obwohl sich die evolutionären Wege der beiden Pathogene bereits vor etwa 2 Millionen Jahren trennten, attackierten sie 18 gleiche Knotenpunkte. Als die Forscher die Gene dieser 18 Proteine ausschalteten, zeigten die Mutanten eine veränderte Immunantwort. Die Forscher schließen daraus, dass die Effektorproteine sich auf Proteine in der Wirtspflanze konzentrieren, welche für deren Immunantwort eine zentrale Rolle spielen.
Das bessere Verständnis über die Wechselwirkungen der Proteine in der Zelle kann sowohl im Pflanzenschutz neue Möglichkeiten eröffnen als auch bei der Entwicklung resistenter Pflanzensorten. Außerdem erhoffen sich die Forscher auf dieser Grundlage zukünftig weitere Erkenntnisse darüber, wie sich Interaktome durch die Entwicklung vielzelligen Lebens verändert haben bzw. wie sie auf Umwelteinflüsse reagieren.
Quellen:
- M. Shaid Mukhtar et al. (2011): Independently Evolved Virulence Effectors Converge onto Hubs in a Plant Immune Systeme Network, In: Science, 29 July 2011, Vol. 33, pp. 596-601, DOI: 10.1126/science.1203659, (Abstract).
- Arabidopsis Interactome Mapping Consortium (2011): Evidence for Network Evolution in an Arabidopsis Ineractome Ma, In: Science, 29 July 2011, Vol. 33, pp. 601-60, DOI: 10.1126/science.1203877 5, (Abstract)
- Christian R. Landry (2011): A Cellular Roadmap for the Plant Kingdom. Science 333, 532; DOI: 10.1126/science.1209753 (Abstract).
Anregungen zum Weiterlesen:
