Virtuelle Mutationen per Mausklick
Machine Learning in der Pflanzenforschung

Forscher haben das genregulatorische Netzwerk rekonstruiert, das im Spross der Arabidopsis durch Stickstoff aktiviert wird. (Bildquelle: © GABI)
Die Modellierung genregulatorischer Netzwerke (GRN) macht dank „Machine Learning“ große Fortschritte. Für Forscher und Züchter öffnen sich dadurch neue Perspektiven und Möglichkeiten, gewünschte Pflanzeneigenschaften noch besser zu optimieren. Experten sprechen bereits von GRN-Engineering und virtuellen Mutationen.
Weltweit arbeiten Forscher und Züchter daran, die Stickstoffeffizienz (N) bei Nutzpflanzen zu optimieren. Ein Ziel ist es, höhere Erträge bei gleichzeitig reduzierter N-Düngergabe zu ermöglichen. Davon würden nicht nur Landwirte, sondern auch die Umwelt profitieren. Das Wissen über die Gene, mit denen Pflanzen Stickstoff wahrnehmen, aufnehmen, assimilieren und verstoffwechseln, ist mittlerweile stark gewachsen und wird in der Züchtung häufig genutzt. Was bislang aber fehlte, waren detaillierte Kenntnisse über das genetische und regulatorische Netzwerk: Wann werden welche Gene ein- und ausgeschaltet, welche Proteine werden synthetisiert und welche Transkriptionsfaktoren steuern diese Gene. Eine neue Publikation bringt nun Licht ins Dunkel.
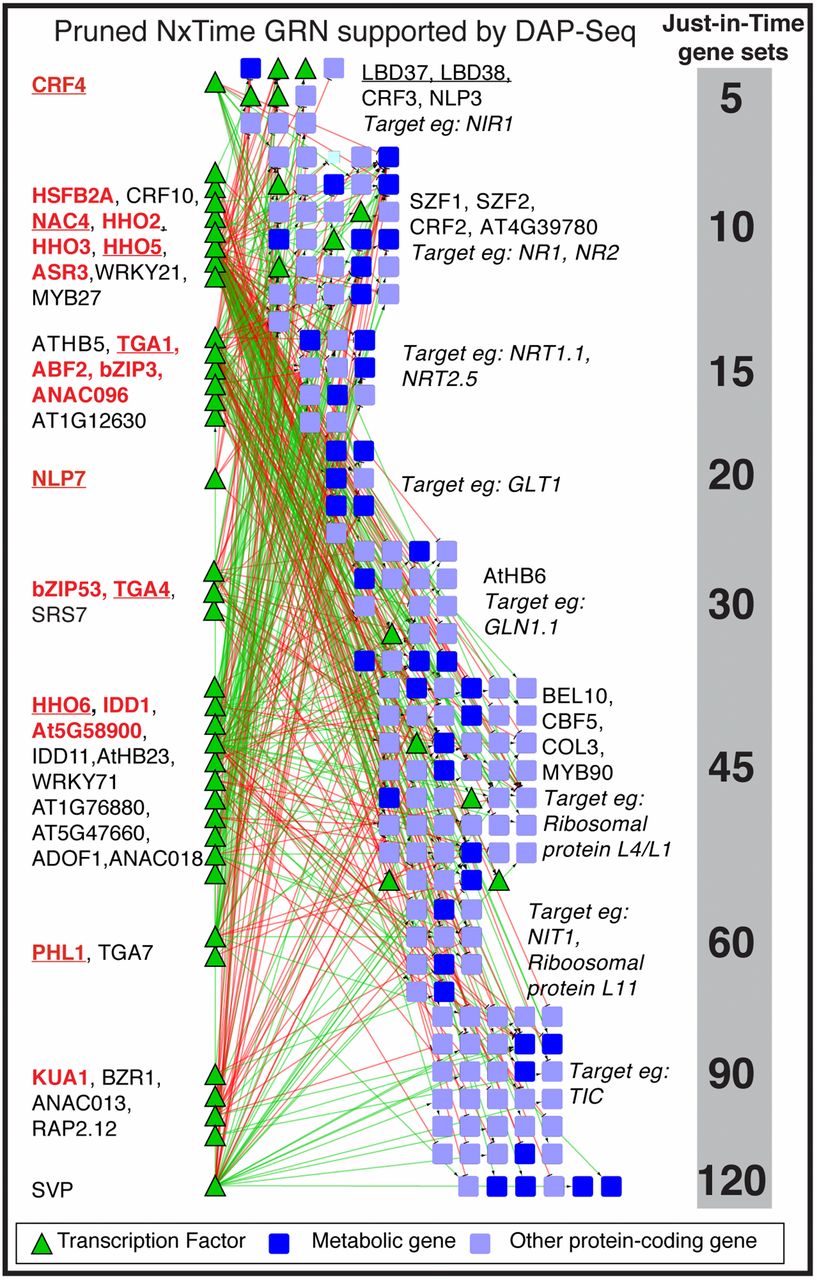
Das Schaubild zeigt, wann die Transkriptionsfaktoren (grüne Dreiecke) aktiv sind und auf welche Gene (blaue Kästchen) sie wirken. In der rechten grauen Spalte stehen die Minuten ab dem Zeitpunkt der Stickstoffgabe.
Bildquelle: © Kranthi Varala et al. (2018)/PNAS/CC BY-ND-NC
De Novo Modellierung mit „künstlicher Intelligenz“ möglich
Am Anfang stand die Frage, welche genetischen Prozesse im Spross junger Arabidopsispflanzen (Arabidopsis thaliana) ablaufen, wenn diese zuvor Stickstoff in Form von Kalium- und Ammoniumnitrat erhalten haben. Um diese Frage zu beantworten, entnahmen die Forscher über zwei Stunden mehrere Proben und analysierten das Transkriptom, also die Gesamtheit der aktiven Gene zum jeweiligen Zeitpunkt. Darauf aufbauend rekonstruierten sie mithilfe des Computers das genregulatorische Netzwerk (GRN) von Grund auf neu.
CRF4 steht an der Spitze der Kaskade
Zuerst fischten sie aus der Fülle der Daten die relevanten 172 Transkriptionsfaktoren und 2.174 Gene heraus. Anschließend setzte der Computer diese über einen Machine-Learning-Ansatz zu einer zusammenhängenden und funktionalen Karte zusammen. Dabei prüfte er Schritt für Schritt, welche Transkriptionsfaktoren gerade aktiv sind, auf welche Gene sie wirken und welche Wirkung sie auf die Expression haben bzw. haben könnten. Denn der Learning-Aspekt bestand darin, dass für die Berechnung immer auch das Expressionsmuster aus den vorherigen Schritten berücksichtig wurde.
Dabei kam heraus, dass am Anfang der N-Signalkaskade ein eigentlich als Kältesensor bekannter Transkriptionsfaktor steht: CRF4. Dieser reguliert eine Gruppe von Genen, die bei der Aufnahme und Assimilation von Stickstoff eine Rolle spielen. In den ersten 15 Minuten sind diese besonders aktiv. Danach treten für eine halbe Stunde Gene zur Energiegewinnung in den Vordergrund, bis nach 45 Minuten schließlich Gene das Ruder übernehmen, die den Stickstoff in den Pflanzenzellen verstoffwechseln.
Beziehungen sind das A und O von Netzwerken
Die Möglichkeit der Rekonstruktion von genregulatorischen Netzwerken ist ein wichtiger Schritt für die Pflanzenforschung und -züchtung. Bisher musste man sich entweder auf kleine Ausschnitte fokussieren (zeitlich und bezogen auf die Zahl der Elemente) oder man musste sich damit zufrieden geben, die einzelnen Bausteine zu kennen, nicht aber ihre kausalen Zusammenhänge. Dabei sind genau sie es, die das Netzwerk lebendig und wichtige Ansatzpunkte sichtbar machen. Gemeint sind Knotenpunkte im genetischen Netzwerk, über die sich gewünschte Eigenschaften und Merkmale am effizientesten beeinflussen lassen.
Modelle von genregulatorischer Netzwerken dienen aber nicht nur als Anschauungsmaterial. Sie bieten z. B. auch die Möglichkeit, virtuelle Mutationen per Mausklick zu setzen. Damit lässt sich am Computer theoretisch durchspielen, was passieren würde, wenn bestimmte Elemente im Netzwerk ausgeschaltet werden.
Bei der Systembiologie geht es nicht mehr nur darum zu verstehen, welche Funktionen die Komponenten einer Pflanzenzelle haben, sondern welche Interaktionsnetzwerke und -muster sie bilden.
Bildquelle: ©LadyofHats/Wikimedia.org/Public Domain
Systembiologie in der Pflanzenforschung
Das Wissen über die genetischen Netzwerke öffnet der Züchtung neue Wege. GRN-Engineering lautet das Stichwort. Das Konzept empfiehlt Forschern und Züchtern, sich künftig nicht mehr nur mit einzelnen Genen oder Allelen zu befassen, sondern verstärkt mit Gen-Kombinationen.
GRN-Engineering greift auf die Idee der Systembiologie zurück. Diese beschäftigt sich mit den intra- und interzellulären Abläufen, um Organismen in ihrer Gesamtheit zu verstehen. Dafür setzt sie u. a. bei den einzelnen Komponenten einer Zelle oder eines Organismus an und fragt, wie diese interagieren und so das Verhalten und die Funktion der Zelle beeinflussen.
Experten sind sicher, dass die Systembiologie die Pflanzenforschung und -züchtung radikal verändern und voranbringen wird. Sie versprechen sich von einem ganzheitlicheren Verständnis des genetischen und zellulären Backgrounds, bestehende Merkmale noch besser stabilisieren und optimieren zu können. Aber auch völlig neue und sogar bisher pflanzenuntypische Merkmale könnten damit entwickelt werden.
Quellen:
- Varala, K. et al. (2018): Temporal transcriptional logic of dynamic regulatory networks underlying nitrogen signaling and use in plants. In: PNAS, (16. Mai 2018), https://doi.org/10.1073/pnas.1721487115
- Lavarenne, J. et al. (2018): The Spring of Systems Biology-Driven Breeding. In: Trends in Plant Science, (12. Mai 2018), https://doi.org/10.1016/j.tplants.2018.04.005
Zum Weiterlesen auf Pflanzenforschung.de:
- Goldgräberstimmung bei pflanzlichen Medikamenten
- Datennetzwerk für die Pflanzenforschung
- den Altvorderen lernen
Titelbild: Forscher haben das genregulatorische Netzwerk rekonstruiert, das im Spross der Arabidopsis durch Stickstoff aktiviert wird. (Bildquelle: © GABI)
