Von KillerRed bis dTomato: Leuchtproteine erobern die Pflanzenforschung

Leuchtproteine revolutionieren die Erforschung zellulärer Prozesse (Quelle: © Sven Hoppe / fotolia.com)
Fluoreszierende Proteine aus Meerestieren haben die Erforschung zellulärer Prozesse revolutioniert. Mit ihnen lassen sich Proteine und sogar RNA-Moleküle im Nanobereich sichtbar machen und Zellen manipulieren. Auch in der Pflanzenforschung kommen sie mit immer ausgefeilteren Techniken zum Einsatz.
Als der Meeresbiologe Osamu Shimomura aus der Qualle Aequorea victoria das „green fluorescent protein“ (GFP) isolierte, ahnte er noch nicht, dass seine Entdeckungen zu einer wissenschaftlichen Revolution führen würden. Er wollte damals verstehen, warum die Pazifikqualle grün fluoreszierte, wenn man sie störte und entdeckte dabei das GFP, ein Molekül, das unter UV-Licht hell grün strahlte.
Eine Revolution für die Zellbiologie
In den vergangenen Jahrzehnten erlangte das Molekül als grün fluoreszierender zellulärer Marker eine überragende Bedeutung für die medizinische und biologische Forschung. Die Neukombination von Genen im Labor machte es möglich, das Quallen-Gen in andere Gensequenzen einzubauen und damit tierische, pflanzliche und bakterielle Proteine grün aufleuchten zu lassen.
Fast jedes Protein lässt sich heutzutage auf diese Weise an GFP koppeln und sichtbar machen. Dadurch kann beispielsweise beobachtet werden, ob zelluläre Proteine an die Zellmembran andocken oder in den Zellkern transportiert werden, wann sie auf- und abgebaut werden. Mit dem neuen, leuchtenden Gen lassen sich diese Prozesse sogar in Echtzeit in lebenden Zellen verfolgen und die Genaktivität kann bestimmten Organen zugeordnet werden. Mit ultraviolettem und blauem Licht lässt sich das GFP einfach durch Zellmembranen und Zellwände hindurch von außen anregen, ohne diese zu zerstören.

Quallen verfügen über fluoreszierende Proteine, die sie im Dünkeln leuchten lassen. Forschern ist es gelungen, einige dieser Proteine zu isolieren (Quelle: © HaPi76 / pixelio.de).
Für ihre Entdeckung und Weiterentwicklung des GFP als Reportermolekül erhielten Osamu Shimomura und die Zellbiologen Martin Chalfie und Roger Tsien 2008 den Nobelpreis für Chemie.
Neue Farben, neue Tricks
Seit der Entdeckung des GFP hat die Anwendung von Leuchtproteinen eine rasante Entwicklung erlebt. Als die Proteinstruktur des GFPs bekannt wurde, machten sich Wissenschaftler bald daran, Aminosäuren in dem farbgebendem Chromophor-Komplex des Proteins auszutauschen und zu ersetzen. Bereits wenige Mutationen bewirkten, dass das GFP in anderen Wellenlängen des Farbspektrums strahlte. Auch wurden aus anderen Meerestieren neue fluoreszierende Reporter isoliert, wie beispielsweise das rot fluoreszierende Protein (RFP oder auch DsRed), das in der Koralle Discosoma striata entdeckt wurde.
Mittlerweise gibt es über 50 GFP- und RFP-Varianten, die die gesamte Farbpalette des sichtbaren Lichts abdecken. Je nach Farbe heißen sie mCherry, dTomato, Citrine oder mBanana. Die diversen Farbschattierungen ermöglichen es, mehrere Proteine oder Zellorganellen gleichzeitig abzulichten.
Einige der neuen Varianten besitzen darüber hinaus aber auch ganz neue Funktionen. Mit KillerRed aus der Anthomedusa-Qualle lassen sich Zellen beispielsweise per Lichtsignal abtöten. Werden Zellen, in denen KillerRed an einen Zellfaktor gekoppelt ist, mit Licht im gelben Wellenbereich bestrahlt, so produziert das Protein Sauerstoffradikale, die Zellorganellen angreifen und schließlich zum Tod der Zelle führen. Dieser Zelltod, der quasi per Fernsteuerung durch ein Lichtsignal eingeleitet werden kann, ist beispielsweise als mögliche Krebstherapie interessant, um Tumorzellen zu zerstören.
Leuchtende Gene in der Pflanzenforschung
Auch in der Pflanzenforschung eröffnete das leuchtende Reportermolekül ganz neue technische Möglichkeiten. Mit ihm ließen sich erstmals Proteine und Strukturen in lebenden Pflanzenzellen in hoher mikroskopischer Auflösung sichtbar machen und filmen. Mittels Fluoreszenzmikroskopie, bei der die Reporter durch Laser oder Licht aus einem bestimmten Wellenbereich zum fluoreszieren angeregt werden, lassen sich beispielsweise Transportprozesse von Hormonen an Wurzel und Sprossachse live verfolgen.
Bei der Erzeugung gentechnisch veränderter Pflanzen wird GFP darüber hinaus genutzt, um Keimlinge oder Zellen auszuwählen, in denen die Übertragung fremder Gene erfolgreich geglückt ist und um bestimmte Zelltypen zu markieren. Mit der sogenannten Fluorescence Activated Cell Sorting (FACS)-Technik lassen sich GFP-markierte Zellen aus einer Pflanze gezielt isolieren.
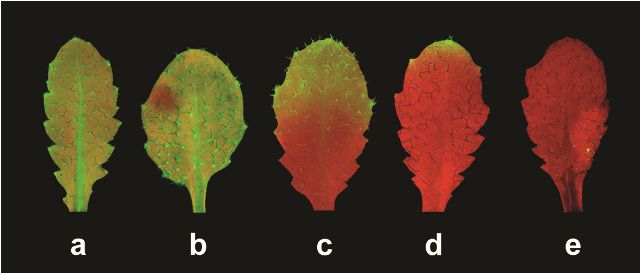
Ausbreitung der Transgeninaktivierung: Tritt bei GFP-exprimierenden Pflanzen (a) die Transgeninaktivierung auf, so beginnt dies als lokales Ereignis (b) wobei Ort und Zeitpunkt der Inaktivierung selbst bei genetisch identischen Pflanzen unterschiedlich sind. Silencing breitet sich mit der Zeit aus (c, d), sodass ganze Blätter (e) und schließlich die gesamte Pflanze betroffen sind. Dieser Prozess kann Tage bis Wochen dauern. (Quelle: © Schmidt / Max-Planck-Institut für molekulare Pflanzenphysiologie).
GFP als Biosensor
Unter Bestrahlung von UV-Licht, sind GFP-positive Pflanzen allerdings auch mit bloßem Auge in intakten Pflanzen im Gewächshaus auszumachen. Aus diesem Grund wird das Protein auch als Biosensor eingesetzt, um zu erforschen, wie Pflanzen auf Umweltreize reagieren. Dazu koppeln Pflanzengenetiker GFP an entsprechende Genschalter, die durch Signale aus der Umwelt ein oder ausgeschaltet werden. Werden beispielsweise Stressgene durch Kälte oder Trockenheit aktiviert, so beginnen bestimmte Pflanzenorgane grün zu fluoreszieren. GFP fungiert dabei auch als Indikator der Genaktivität und zeigt wo und wann bestimmte Stressgene in der Pflanze aktiv werden.
Um GFP als Stress-Biosensor zu nutzen, entwickelten Wissenschaftler sogar eine spezielle Stresssensitive GFP-Variante. Das sogenannte redox-sensitive GFP (roGFP) reagiert auf die Anwesenheit reaktiver Sauerstoffradikale, die beispielsweise bei Trockenstress in Pflanzenzellen entstehen. Durch Einbau zweier Cystein-Aminosäuren in das ursprüngliche GFP, wechselt roGFP zwischen zwei Anregungswellenlängen. Werden die Cysteine durch Sauerstoffradikale oxidiert, verändert sich die Proteinstruktur des roGFP und es fluoresziert stärker bei einer Anregung mit blauem Licht von 400 Nanometer (nm). Befindet sich das Protein in nicht oxidiertem Zustand, benötigt man längerwelliges Licht von 490 nm, um eine maximale Fluoreszenz zu erreichen. Je nach dem Gehalt der schädlichen Sauerstoffradikale in der Zelle, fluoresziert das roGFP demnach entweder stärker bei der einen oder anderen Wellenlänge. In der Forschung wird diese Methode vor allem genutzt, um zu untersuchen, wie stressanfällig unterschiedliche Pflanzengewebe und Zellorganellen unter den verschiedensten Umweltbedingungen sind.
Schaltbare Leuchtproteine, der Schlüssel zur Nanoskopie
Mittlerweile ist es gelungen, die fluoreszierenden Reportermoleküle so weiterzuentwickeln, dass sie gezielt durch das Einstrahlen von Licht gesteuert werden können. Der Fluoreszenzreporter EosFP ändert beispielsweise bei Aktivierung mit violettem Licht seine Farbe von grün nach rot. Auf diese Weise können gleiche Zellstrukturen und Proteine über einen bestimmten Zeitraum hinweg unterschiedlich farbig markiert werden, um beispielsweise zu verfolgen, wie die Zellen während der Organentwicklung ihre Positionen ändern.
Darüber hinaus werden immer neue Techniken entwickelt, um mit Hilfe photoschaltbarer Leuchtproteine Ultrastrukturen im Nanobereich live zu beobachten. Aufgrund der Beugung der Lichtwellen ist die Auflösung in der Fluoreszenzmikroskopie auf etwa 200 nm beschränkt, so dass sehr kleine Farbstoffmoleküle mit einer Größe von zirka einem Nanometer nur in Form eines „verwaschenen“ Lichtflecks erkannt werden. Nur mit aufwändiger Eletronenmikroskopie war es bisher möglich, solche superhochaufgelösten Strukturen abzubilden. Die exakte Position von Proteinkomplexen, die eine Größe von nur einigen Nanometern besitzen und deren Interaktionen mit anderen Proteinen konnte daher mit der Fluoreszenzmikroskopie bisher nicht aufgelöst werden.
Physikalische Grenzen überwinden
Photoschaltbare Proteine haben es jedoch möglich gemacht, diese physikalische Grenze zu überwinden. Techniken wie die Photoaktivierte Lokalisationsmikroskopie (PALM) und die Stochastisch Optische Rekonstruktionsmikroskopie (STORM) basieren beide auf Fluoreszenzmarkern, die mit Lichteinstrahlung reversibel ein- und ausgeschaltet werden können. Nur einige wenige Leuchtmoleküle verbleiben dabei in ihrem fluoreszierenden Zustand. Ihre Position wird mathematisch exakt bestimmt bevor auch sie mit Licht wieder ausgeschaltet werden. Darauf folgt der nächste Zyklus, in dem wieder andere Reportermoleküle in ihre fluoreszierende Form überführt werden. Aus tausenden von Lokalisationen entsteht schließlich das superhochaufgelöste Bild.
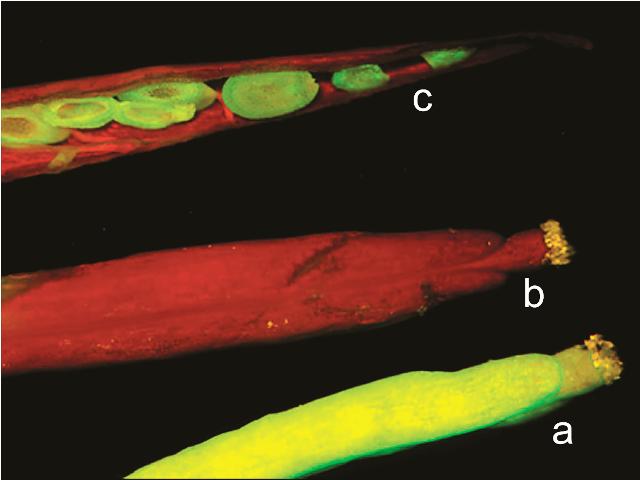
Die Transgeninaktivierung ist reversibel. Die unter (b) und (c) gezeigten Schoten transgener Arabidopsispflanzen wiesen Inaktivierung der GFP-Transgene auf, die Samen in der geöffneten Schote (c) zeigten jedoch GFP-Fluoreszenz und somit die Reaktivierung der GFP-Transgene in den Nachkommen. Zum Vergleich ist unter (a) die Schote einer Pflanze mit hoher und stabiler GFP-Expression gezeigt (Quelle: © Schmidt / Max-Planck-Institut für molekulare Pflanzenphysiologie).
Zu den reversibel schaltbaren Fluoreszenzmarkern, die für STORM und PALM genutzt werden, gehört beispielsweise die kürzlich entwickelte GFP-Variante Dreiklang. Dreiklang lässt sich erstmals durch Wellenlängen außerhalb des optischen Bereiches kontrollieren, das bedeutet das Anregung und Photoaktivierung bei diesem Reporter erstmals entkoppelt sind. Diese Eigenschaft macht es besonders geeignet für die STORM-Technik.
Kleinste pflanzliche Strukturen mit 3D-SIM ablichten
In der Pflanzenforschung ist es mit PALM und STORM allerdings noch nicht geglückt, superhochaufgelöste zu Bilder an intakten Zellen aufzunehmen. Ein technisches Problem bereitet die etwa 700 nm dicke Zellwand von Pflanzenzellen, durch die Zellorganellen zu weit von dem Deckgläschen entfernt liegen, um sie mit diesen Methoden hochaufgelöst abzulichten.
Erste superhochauflösende Bilder aus lebenden Pflanzenzellen konnten jedoch mit der Dreidimensionalen-Struktur-Illuminations-Mikroskopie (3D-SIM) erzeugt werden. Bei der 3D-SIM Mikroskopie werden mehrere Bilder pro Aufnahmebereich aus unterschiedlichen Winkeln geschossen und nachbearbeitet. Aus den entstehenden Schattenmustern lassen sich schließlich die kleinsten Strukturen einer Zelle berechnen.
Mit dieser Methode gelang es Wissenschaftlern kürzlich, feinste Strukturen von Plasmabrücken in den Siebzellen von Tabakpflanzen aufzunehmen, durch die der Stoffaustausch zwischen Pflanzenzellen stattfindet.
RNA-Moleküle live verfolgen mit Spinach
Mit einer neuen Technik ist es jetzt auch erstmals möglich geworden RNA-Moleküle, die bei der Umsetzung von genetischer Information in Proteine als Zwischenmolekül gebildet werden, grün fluoreszieren zu lassen. Wissenschaftler isolierten eine Komponente aus dem GFP, die sobald sie an RNA-Moleküle bindet, grün zu leuchten beginnt. Den künstlich herstellten RNA-Farbstoff Komplex tauften die Forscher „Spinach“.
Spinach lässt sich an RNA-Moleküle von Zellen koppeln, um deren Verteilung und Funktion in Zellen zu untersuchen. Der neue Marker kann möglicherweise auch dafür genutzt werden, um Wechselwirkungen mit anderen RNA-Molekülen und RNA-bindenden Proteinen zu untersuchen. Die Abstände von Biomolekülen zueinander können mit dem sogenannten Förster-Resonanzenergietransfer (FRET) gemessen werden, bei dem die Energie eines Farbstoffes auf einen zweiten übertragen wird. Je mehr Energie das fluoreszierende Donormolekül abgibt, desto näher befindet es sich an seinem potentiellen Interaktionspartner.
Die Entwicklung des Spinach-Komplexes wurde bereits mit Begeisterung von einigen Wissenschaftlern aufgenommen. Möglicherweise werden die neuen fluoreszierenden RNA-Moleküle die Erforschung der RNA in ähnlicher Weise vorantreiben wie die Entdeckung des GFPs die Erforschung von Proteinen revolutioniert hat.
Quellen:
- Brakemann T. et al. (2011). A reversibly photoswitchable GFP-like protein with fluorescence excitation decoupled from switching. Nature Biotechnology 29: doi:10.1038/nbt.1952.
- Paige J. et al. (2011). RNA Mimics of Green Fluorescent Protein. Science 333: doi 10.1126/science.1207339.
- Fitzgibbon J. et al. (2010). Super-Resolution Imaging of Plasmodesmata Using Three-Dimensional Structured Illumination Microscopy. Plant Physiology 153: 1453-1463, doi: 10.1104/pp.110.157941.
- Nienhaus G. et al. (2010). Photoaktivierbare fluoreszente Proteine für die Zellbiologie. BIOspektrum 4: 432-434.
Weiterlesen auf Pflanzenforschung.de:
