Willkommen in der Pseudozeit
Algorithmus liefert neue Einblicke in die Zelldifferenzierung
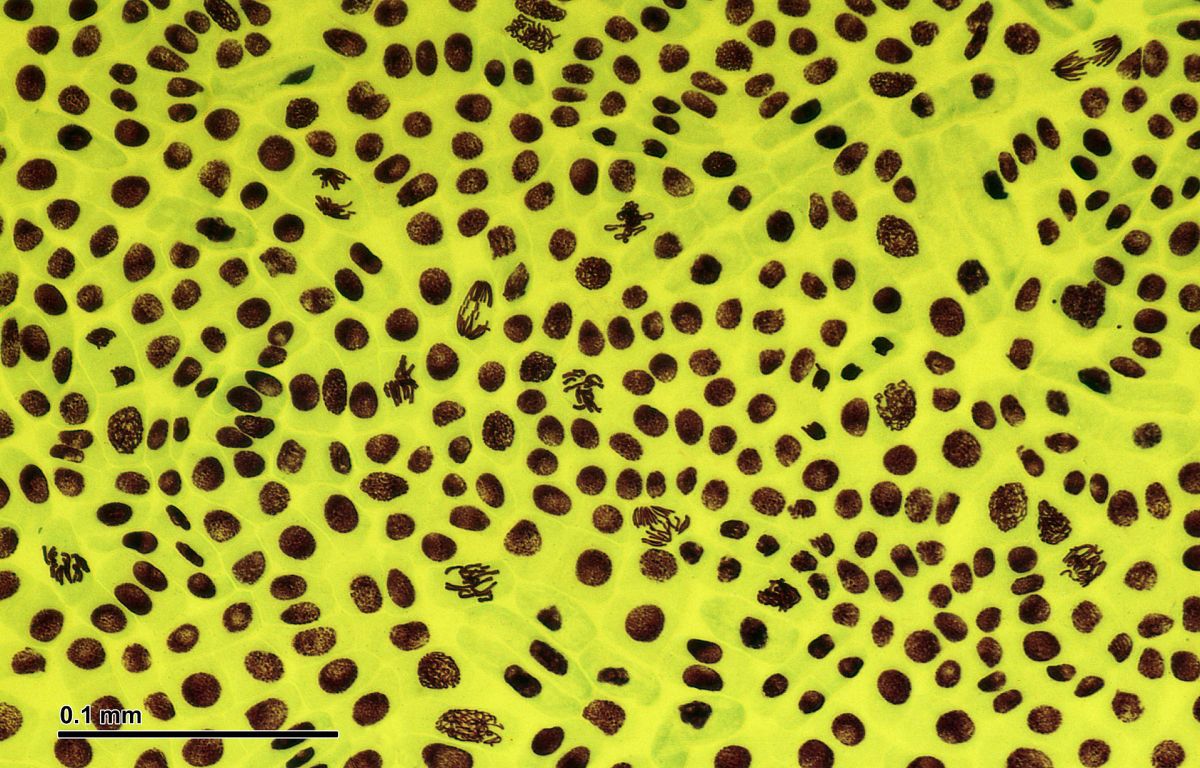
Blick durch das Mikroskop auf das Wurzelmeristem der Ackerbohne. Es ist das Bildungsgewebe, in dem laufend neue Zellen gebildet werden und sich anschließend differenzieren. (Bildquelle: © Josef Reischig/ wikimedia.org/ CC BY-SA 3.0)
Das Genexpressionsprofil einer Zelle liefert Hinweise über ihr Entwicklungsstadium. Deshalb werden bei der Erforschung der Zelldifferenzierung Methoden der RNA-Sequenzierung eingesetzt. Bisherige Methoden der Massenanalyse stießen jedoch angesichts der teils vielfältigen Verästelungen und Abzweigungen an ihre Grenzen. Dies gilt jedoch nicht mehr für eine neue Software, die von Forschern aus Deutschland entwickelt wurde. Sie liefert neue Einblicke in die Zellentwicklung.
Egal ob Pflanze, Pilz oder Tier, ob in Gewebe oder Organen. Wer sich die Zelldifferenzierung wie einen wohl geordneten Prozess vorstellt, der gleichmäßig für kontinuierlichen Nachschub an spezialisierten Zellen sorgt, lässt sich von der Ruhe täuschen, die Schaubilder in Lehrbüchern ausstrahlen. Von der Analogie her ähnelt dieser meist eher einem sich permanent wiederholenden fliegenden Massenstart. „So starten nie alle Zellen einer Probe gleichzeitig ihre Entwicklung und brauchen unterschiedlich lange. Daher haben wir es immer mit einem dynamischen Gemisch zu tun. Daraus die Abfolge mehrerer Schritte eines Prozesses zu konstruieren, ist enorm schwer, zumal die Zellen nur für eine Messung zur Verfügung stehen“, erklärt Erstautorin Laleh Haghverdi die Tücken. Denn trotz aller Kenntnisse zu den Ursachen, Faktoren und Mechanismen, ist es nach wie vor schwierig, den Prozess der Zelldifferenzierung im Detail nachzuvollziehen.
Eine Koproduktion aus München

Hier kann mit bloßem Auge das Ergebnis der Zelldifferenzierung beobachtet werden. Zu sehen ist das Apikalmeristem eines jungen Geldbaums (Crassula ovata), eine beliebte Zimmerpflanze. Die Bilder wurden im Abstand von 14 Tagen aufgenommen und zeigen, wie sich an der Spitze des Sprosses neue Blätter bilden.
Bildquelle: © Daniel,levine/ wikimedia.org/ CC BY-SA 3.0
Gemeinsam mit Wissenschaftlern der Technischen Universität München haben Forscher des Helmholtz-Zentrums München nun eine Software entwickelt, die in der Lage ist, Proben mit Tausenden Zellen, die sich alle in unterschiedlichen Entwicklungsstadien befinden, individuell zu analysieren und die Ergebnisse anschließend zu einem großen Bild zusammenzusetzen. Die Probe aufs Exempel erfolgte am relativ einfachen Beispiel der Blutbildung (Hämatopoese), bei der aus blutbildenden Stammzellen unterschiedliche Blutzelltypen entstehen. Bevor es um die Software im Speziellen geht, soll zunächst aber beantwortet werden, wie es überhaupt möglich ist, festzustellen, in welchem Entwicklungsstadium sich eine Zelle befindet. Anders als bei Menschen fehlt es ihnen schließlich an markanten Merkmalen, wie eine sich im Laufe der Entwicklung verändernde Körpergröße, -gestalt oder-behaarung.
Wenn äußere Merkmale zur Bestimmung fehlen
Rückschlüsse liefert stattdessen das Genexpressionsprofil, also das Abbild der zum Untersuchungszeitpunkt aktiven Gene. Hintergrund ist, dass sich diese je nach Entwicklungsstadium unterscheiden, bestimmte Gene also für einen bestimmten Abschnitt in der Differenzierungsphase stehen. Damit bei der Gen-Analyse auch tatsächlich nur die aktiven berücksichtigt werden, werden für die Bestimmung des Stadiums nur die transkribierten RNA-Sequenzen herangezogen bzw. sequenziert. Für Biologen handelt es sich bis hierhin mehr oder weniger um ein Kinderspiel, anders jedoch als das, was folgt.
Die Summe der einzelnen Teile
Die Herausforderung besteht nämlich darin, die ermittelten Transkriptomdaten aus allen individuellen Zellen zu einem großen Bild zusammenzusetzen, das der Wirklichkeit des Differenzierungsprozesses entspricht. Somit ist nicht vorgesehen, die Entwicklung einer einzelnen Zelle über den gesamten Entwicklungsprozess zu verfolgen, sondern den zellulären Differenzierungsprozess durch das richtige Aufreihen mehrerer Zellen in unterschiedlichen Entwicklungsstadien abzubilden. Da, wie Haghverdi erklärte, nie alle Zellen gleichzeitig starten würden, ist die Wahrscheinlichkeit stets hoch genug, dass in einer einzigen Probe genügend Zellen aus allen Entwicklungsstadien vertreten sind.
Schicksalswege von Zellen nachempfinden
Der Vorteil der neu entwickelten Methode im Vergleich zu anderen ist, dass sie die Verzweigungen und Verästelungen während der Zelldifferenzierung sichtbar macht, ähnlich wie bei einem Familienstammbaum. Darüber hinaus, dass sie den gesamten Prozessablauf, die verschiedenen Entwicklungsstadien sowie die jeweiligen Endpunkte detaillierter und klarer abbildet. Fabian Theis spricht in diesem Zusammenhang vom Nachzeichnen der Schicksale der Zellen. Der Clou besteht u. a. darin, dass der Ablauf der Zelldifferenzierung nicht mehr nur vom eigentlichen Start aus betrachtet wird, sondern zugleich auch rückwärts, von den jeweiligen Endpunkten aus.
Ein anderes Verständnis von Zeit
Kernstück der Software ist der von den Forschern entwickelte Algorithmus „Diffusion Pseudotime“, kurz DPT. Das Geheimnis hinter seinem kryptischen Namen ist schnell erklärt. Denn was der DPT-Algorithmus im Endeffekt tut, ist eine diffuse Menge zellulärer Transkriptomdaten mithilfe von Regeln der Wahrscheinlichkeitsrechnung in die richtige Reihenfolge zu bringen. Dass hierbei von „pseudotime“, zu Deutsch Pseudozeit, die Rede ist, liegt daran, dass die zugrunde liegende Probe wie erwähnt ein Schnappschuss aller vorhandenen Entwicklungsstadien zu einem einzigen Zeitpunkt ist. Den Differenzierungsprozess an einem Zeitstrahl in Minuten oder Sekunden abzubilden, wäre daher selbst beim besten Willen nicht möglich, da diese nicht erfasst wurden. Für die korrekte Darstellung des Ablaufs des Differenzierungsprozesses ist dies jedoch im Grunde auch gar nicht nötig.

Kaum zu glauben, dass das mal ein Maiskolben war. Schuld ist der Schadpilz Ustilago maydis, der ausschließlich Maispflanzen befällt und den sogenannten Maisbeulenbrand auslöst. Bei der Kolonisierung auf der Oberfläche spielt die Zelldifferenzierung des Schädlings eine entscheidende Rolle. In Mexiko gilt sein Werk übrigens als Delikatesse, wird sogar "Mexikanischer Trüffel" genannt.
Bildquelle: © Hans-Martin Scheibner/ wikimedia.org/ CC BY-SA 3.0
Was leistet der neue Algorithmus?
Nichtdestotrotz eröffne der DPT-Algorithmus neuen Möglichkeiten, erklärt Theis: „Wir können beispielsweise zeigen, wie sich im von uns gewählten Beispiel ein relativ einheitlicher Verband von Blutstammzellen zu unterschiedlichen Zelltypen entwickelt. Während die einen zu roten Blutkörperchen (Erythrozyten) werden, differenzieren andere zu endothelartigen Zellen.“ Bei Letzteren handelt es sich um die nach innen gerichteten Zellen von Blutgefäßen, die im Grunde alle Gefäße des Herz-Kreislauf-Systems mit einer einzelligen Schicht auskleiden.
Zu den Hintergründen der Geschichte vordringen
Theis´ Beispiel macht deutlich, dass die Beschränkung auf das Maß der Pseudozeit keineswegs als Nachteil zu werten ist. Allein die Möglichkeit, die verschiedenen Abzweigungen im Verlauf der Zelldifferenzierung nachvollziehen zu können, erlaubt es, diese besser zu erforschen. Hier geht es weniger darum, zu bestimmen, wann, sondern was, in welcher Reihenfolge und warum passiert.
So zählt zu den wichtigsten Fragen, welche Faktoren, z. B. chemische Signale im Cytoplasma oder Transkriptionsfaktoren, einzeln oder im Zusammenspiel bestimmen, dass die eine Zelle den einen Entwicklungspfad einschlägt, ihr Nachbar jedoch einen völlig anderen. Es ist ein Unterschied, zu wissen, welche unterschiedlichen Zelltypen aus einem Stammzellendepot hervorgehen, andererseits die Hintergründe zu kennen.
Einsatz in der Pflanzenforschung
Einblicke in die Zelldifferenzierung zu erhalten, ist auch für die Pflanzenforschung von großem Interesse. Einerseits natürlich wegen des Erkenntnisgewinns an sich, andererseits aufgrund der neuen Einflussmöglichkeiten, die sich daraus ergeben, z. B. in Bezug auf Zelltypen, die ertragsrelevant sind oder die pflanzliche Fitness und Robustheit beeinflussen. Auch die Architektur einer Pflanze, ließe sich gezielt verändern, sodass sie effizienter mit Ressourcen wie Licht, Wasser oder Nährstoffen umgeht.
Ein weiteres Anwendungsfeld ist die Erforschung der Zelldifferenzierung bei pathogenen Organismen, z. B. Schadpilze, bei denen sie mitunter bei der Kolonisierung ihrer Wirtspflanzen eine Rolle spielt. „Künftig wollen wir uns vor allem Prozesse anschauen, die bisher unverstanden oder möglicherweise noch gar nicht entdeckt sind“, erklärt Theis mit Blick auf die Zukunft. Und noch eins macht die vorliegende Studie deutlich: Biologie und Informationstechnologie verschmelzen mehr und mehr zu einer Einheit.
Quelle:
Haghverdi, L. et al. (2016): Diffusion pseudotime robustly reconstructs lineage branching. In: Nature Methods, (29. August 2016), doi:10.1038/nmeth.3971.
Zum Weiterlesen auf Pflanzenforschung.de:
- Von der Zelle zur Wurzel
- Neuer Zusammenhang beim Pflanzenwachstum entdeckt
- Molekularer Schalter entdeckt
Titelbild: Blick durch das Mikroskop auf das Wurzelmeristem der Ackerbohne. Es ist das Bildungsgewebe, in dem laufend neue Zellen gebildet werden und sich anschließend differenzieren. (Bildquelle: © Josef Reischig/ wikimedia.org/ CC BY-SA 3.0)
