Metatranskiptomanalyse des Wurzelmikrobioms
„Besiedlungsgene“ identifiziert
Bakterien können Pflanzenwurzeln besiedeln. Dafür müssen sie bestimmte Gene aktivieren, um eine erfolgreiche Lebensgemeinschaft mit der Pflanze eingehen zu können (Symbolbild). (Bildquelle: © Pflanzenforschung.de)
Pflanzliche Sekrete machen den Wurzelraum zu einem Hotspot mikrobieller Aktivität. Doch welche Gene ermöglichen es Bakterien, dort erfolgreich zu siedeln? Mit Hilfe einer neuen Methode gibt es jetzt erste Antworten.
Zahlreiche Bakterien und Pilze besiedeln den Raum um die Wurzeln, teilweise die Wurzeln selbst: das Wurzelmikrobiom. Für die Pflanze ist das mit zahlreichen Vorteilen verbunden. Die Mikroorganismen spielen eine entscheidende Rolle bei der Nährstoffaufnahme, indem sie Pflanzen bei der Umwandlung und Aufnahme von Nährstoffen wie Stickstoff und Phosphor unterstützen, die sonst für die Pflanze schwer zugänglich wären. Sie tragen zur Pflanzengesundheit bei, indem sie pathogene Mikroben abwehren und das Immunsystem der Pflanzen stärken. Außerdem können sie das Wachstum fördern, indem sie Wachstumshormone produzieren und stressmindernde Bedingungen schaffen. So helfen sie, Pflanzen unter widrigen Umweltbedingungen widerstandsfähiger zu machen.
Transkriptomanalyse mit hohen Hürden
Aber wie Pflanzen und Bakterien im Wurzelraum genau miteinander interagieren, ist bislang noch nicht im Detail verstanden. Die individuellen Genaktivitäten der Mikroorganismen anhand der Analyse der gebildeten Transkripte (mRNAs) in der natürlichen Umgebung zu erforschen, scheiterte bisher an mehreren Hürden. Zum einen sind da die rRNAs und mRNAs der Pflanze, die es von den mRNAs der Mikroorganismen zu unterscheiden gilt. Zum anderen sind die Unterschiede der mRNA-Sequenzen eng verwandter Mikroorganismen oft zu gering, um sie sicher auseinanderzuhalten und korrekt zu kartieren. Mit einem Kniff und modernen Analysemethoden ist es nun einem Team der Universität Köln und des Max-Planck-Instituts für Pflanzenzüchtungsforschung in Köln gelungen, Licht in die teils seit 450 Millionen Jahren etablierten Abläufe bei der mikrobiellen Besiedelung von Pflanzenwurzeln zu bringen.
Künstliches Mikrobiom war die Lösung
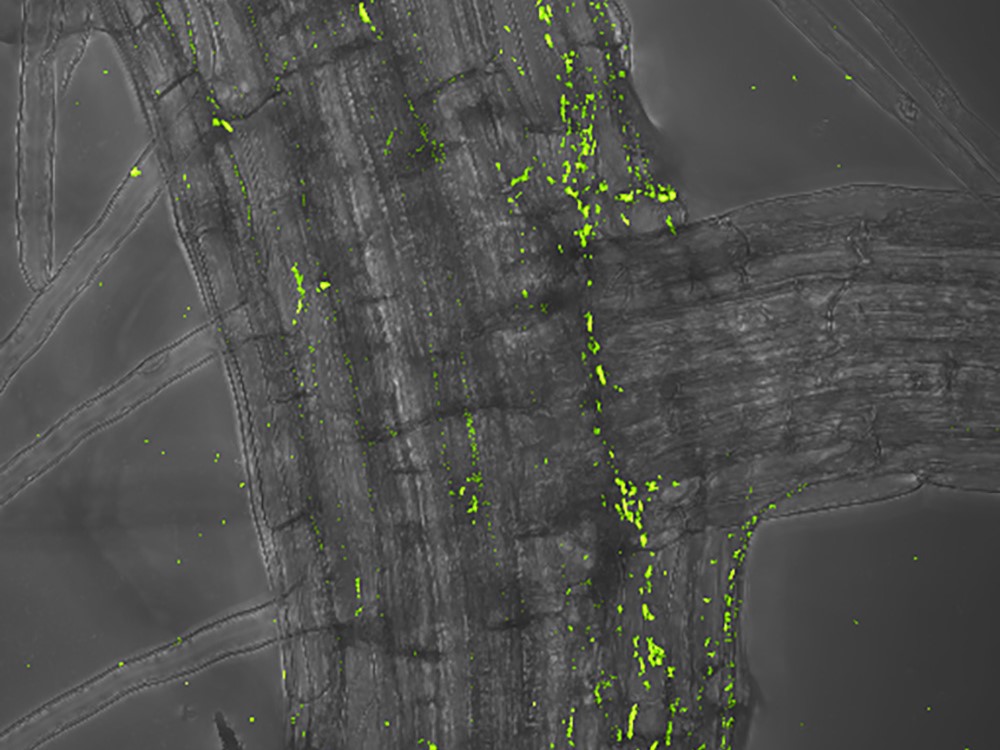
Rhodanobacter-Bakterium, hier grün markiert, besiedelt die Wurzel einer Ackerschmalwand.
Bildquelle: © Jana Ordon
Dazu haben die Forscher:innen zunächst ein künstliches Mikrobiom erstellt. Sie besiedelten die keimfreien Wurzeln von Arabidopsis thaliana mit 84 Bakterien- und 22 Pilzarten, die repräsentativ für ein natürliches Mikrobiom sind. Der Clou dabei: Von allen 106 Arten war das Genom bereits sequenziert. Dieser Ansatz begrenzte nicht nur die mögliche Transkriptom-Vielfalt, sondern erleichterte es, das Meta-Transkriptom richtig zu kartieren.
Zusätzlich entfernte das Forschungsteam große Teile der pflanzlichen RNA vor der Analyse mittels einer Methode, die als „rRNA-Depletion“ bezeichnet wird. Dadurch konnte Nicht-rRNA gegenüber rRNA um mehr als den Faktor 25 angereichert werden. Die anschließende Transkript-Sequenzierung und die Kartierung ermöglichten es, rund 42 Prozent der Reads aus Bodenproben direkt um die Wurzeln (dieser Bereich wird auch als Matrix bezeichnet) und knapp 9 Prozent der Reads aus Wurzelproben individuellen mikrobiellen Genomen zuzuordnen. Damit war die Methode erfolgreich darin, einen größeren Teil des Transkriptoms der mit der Pflanze assoziierten Bakterien und Pilze zu erfassen, aber noch weit davon entfernt, das gesamte Meta-Transkriptom der künstlichen Mikrobengemeinschaft abzubilden. Immerhin: Im Direktvergleich zeigte sich, dass mittels RNA-Analyse mehr individuelle Mikroorganismen-Stämme nachgewiesen werden konnten als mit einem DNA-basierten Ansatz, der die 16S-rRNA-Gene von Bakterien bzw. die ITS-Regionen in den Pilzgenomen als Marker nutzt.
Das Ergebnis: Die bakterielle Mikrobiom-Gemeinschaft wird dominiert von den Ordnungen Xanthomonadales, Actinomycetales, Pseudomonadales und Rhizobiales. Hypocreales und Glomerallales waren bei den Pilzen am häufigsten.
Kontakt zur Pflanze verändert das Transkriptom
Anschließend untersuchten die Forscher:innen, welche Gene die Mikroorganismen bei Kontakt mit dem pflanzlichen Wirt herauf- oder herunterregulieren. Das Ergebnis: Ca. 2000 bakterielle Gene zeigten verstärkte Aktivität, etwa 1000 Gene eine geringere. Die generell aktivsten Gene waren in ihrer Funktion assoziiert mit der Bildung von Pili, der Synthese von Glucosamin-6-Phosphat (ein Schlüsselzwischenprodukt in der Biosynthese von Chitin und Peptidoglykan als Komponenten der Zellwände vieler Bakterien), dem Zellwandumbau, der Eisenaufnahme, der bakteriellen Pathogenese oder der Ausscheidung von Antibiotica.
Bei den Pilzen fanden die Forscher:innen nur etwa 100 Gene mit einer veränderten Aktivität. Hochreguliert waren z.B. Gene für mutmaßliche Effektoren, Proteine der Cerato-Platanin-Familie, Peptidasen und Proteasen sowie mutmaßliche Virulenzfaktoren, die wichtig sind, um einen Wirt zu besiedeln.
Aktivitätsveränderungen sind oft über Artgrenzen hinaus konserviert
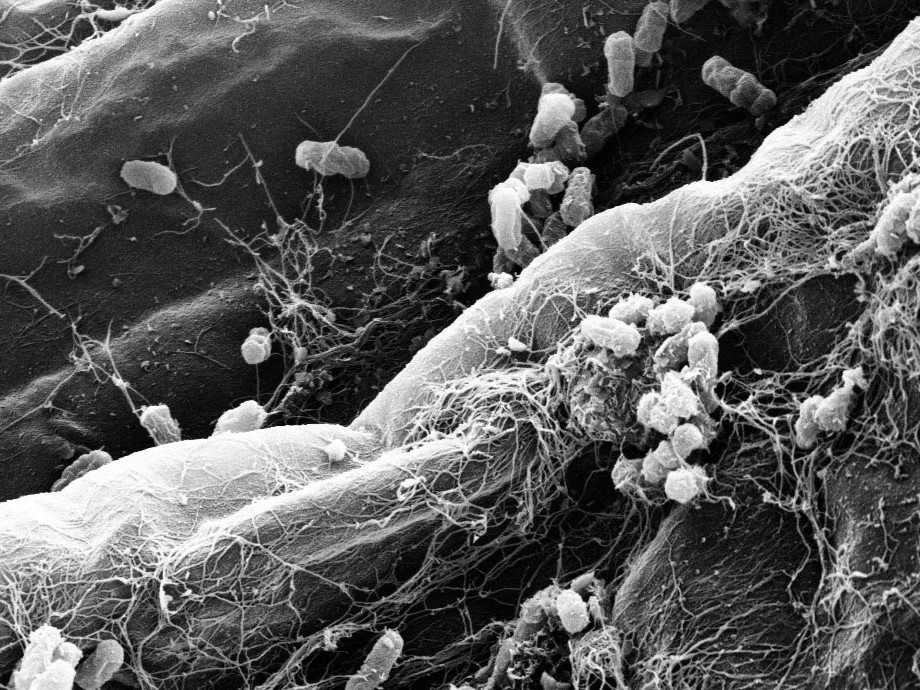
Besiedlung der Wurzeln durch Bakterien der Wurzelmikrobiota.
Bildquelle: © S. Hacquard
Viele der durch den Pflanzenkontakt umregulierten Genaktivitäten waren individuell, doch es zeigten sich auch einige Gemeinsamkeiten: Die Zugehörigkeit zu einer bestimmten Bakterienklasse war für 13 Prozent der Transkriptom-Unterschiede entscheidend, auf Ebene der Bakterien-Ordnung bereits 23 Prozent, bei der Familie 40 Prozent und bei der Art 55 Prozent. Ob die Probe aus Wurzelgewebe oder der Matrix stammte, korrelierte nur mit knapp sieben Prozent der Transkriptom-Unterschiede.
Effiziente Phosphataufnahme für die Besiedelung wichtig
Orthologe Gene, die in zahlreichen Bakteriengruppen zur Wurzelbesiedelung hochreguliert werden, dienen vor allem der Translation, der Energieproduktion und -umwandlung, der Replikation und Reparatur sowie dem Zellwandumbau. Demnach sind bei den Bakterien vor allem solche Funktionen für die Ausbildung eines Wurzelmikrobioms evolutionär konserviert, die aktives Wachstum und eine hohe Protein-Biosyntheserate ermöglichen. Weiterhin fanden sich unter den Top 200 der aktivierten orthologen Gene solche, die mit Transport- sowie Sekretionsprozessen assoziiert sind.
Die Bedeutung einzelner hochregulierter Gene testeten die Forscher:innen mithilfe von Knock-out-Stämmen von Rhodanobacter. Ein Ergebnis: Insbesondere das evolutionär konservierte Gen typA (kodiert für ein GTP-Bindungsprotein) und die pst-Familie (kodieren für einen Phosphat-Transporter) sind für die Besiedlung der Wurzel essenziell. Eine effiziente Phosphataufnahme könnte somit einer der Schlüssel sein, damit Bakterien eine Pflanzenwurzel erfolgreich besiedeln können.
Methodischer Fortschritt mit klaren Grenzen
Insgesamt konnte die Studie zeigen, dass es ein breites Spektrum an Genen gibt, die Bakterien nutzen, um den Wurzelraum zu besiedeln, und dass einige davon über phylogenetisch weite Distanzen konserviert sind. Der in dieser Arbeit verwendete methodische Ansatz hat es erstmals ermöglicht, komplexe genregulatorische Zusammenhänge von Bakterien in einem Mikrobiom zu analysieren. Zugleich zeigt die Studie die heute noch bestehenden Grenzen auf: Möglich war diese Analyse nur für eine künstliche und zuvor sequenzierte mikrobielle Gemeinschaft.
Wer noch mehr über das Wurzelmikrobiom und seine vielfältigen Interaktionen erfahren möchte, hier ein Hinweis: Eine vorangegangene Studie des Max-Planck-Instituts für Pflanzenzüchtungsforschung in Köln in Zusammenarbeit mit einem internationalen Forscherteam hat natürliche chemische Strategien identifiziert, die Bakterien nutzen, um bakterielle Konkurrenten fernzuhalten und sich erfolgreich auf Pflanzenwurzeln zu vermehren.
Quellen:
- Vannier, N., et al. (2023): „Genome-resolved metatranscriptomics reveals conserved root colonization determinants in a synthetic microbiota.“ In: Nature Communications, online (13.12.2023). doi:10.1038/s41467-023-43688-z.
- Getzke, F. et al. (2023): “Co-functioning of bacterial exometabolites drives root microbiota establishment”. In: Proceedings of the National Academy of Sciences of the United States of America, PNAS (2023). doi: 10.1073/pnas.2221508120
Zum Weiterlesen auf Pflanzenforschung.de:
- Multikulti im Ackerboden - Vielfältige Bodenmikrobiome können Pflanzen schützen
- Globalisierung auch in der Mikrobenwelt - Das Bodenmikrobiom verliert an Vielfalt – weltweit!
- Der Mikrobiota-Schnelltest - Neues Protokoll zur Kultivierung von Bakterien im Wurzelraum
Titelbild: Bakterien können Pflanzenwurzeln besiedeln. Dafür müssen sie bestimmte Gene aktivieren, um eine erfolgreiche Lebensgemeinschaft mit der Pflanze eingehen zu können (Symbolbild). (Bildquelle: © Pflanzenforschung.de)
