Gute Prognose
Künstliche Intelligenz erkennt Promotorstärke

Das Team entwickelte ein neuronales Netzwerkmodell, mit dem vorhergesagt werden kann, wie sich Änderungen an nicht kodierenden DNA-Sequenzen in Hefe auf die Genexpression auswirkt. Das Modell erstellt die hier gezeigten Karten. (Bildquelle: © Martin Krzyw
Promotoren entscheiden darüber, wie häufig ein Gen transkribiert und das entsprechende Protein gebildet wird. Ein Forschungsteam hat nun ein neuronales Netz darauf trainiert, die Aktivität eines Gens anhand der Promotorsequenz vorherzusagen.
Das Erbgut eines Organismus’ zu verstehen, bedeutet nicht nur, den jeweiligen DNA-Sequenzen Proteine und den Proteinen Funktionen zuordnen zu können. Wichtig ist auch die Vorhersage, wie stark einzelne Gene transkribiert werden. Belastbare Prognosen waren bislang schwierig, weil dahinter komplexe biochemische Wechselwirkungen stecken. Ein Forschungsteam hat nun mit einem Hochdurchsatz-Experiment Daten erzeugt, anhand derer sie eine Künstliche Intelligenz (KI), ein neuronales Netz, trainiert haben. In der Folge konnte diese KI mit hoher Zuverlässigkeit aus der Sequenz von Promotoren die Expressionsstärke des zugehörigen Gens vorhersagen.
30 Millionen Promotoren getestet
Für ihre Experimente haben die Fachleute die einzellige Bäckerhefe Saccharomyces cerevisiae genutzt. Sie haben gemessen, wie stark das Gen yfp, das für ein gelb fluoreszierendes Protein kodiert, in der Zelle exprimiert wird. Allerdings haben sie das nicht nur für eine Zelle, sondern für mehr als 30 Millionen Hefezellen gemacht – ein vor wenigen Jahren noch undenkbarer Ansatz. In jeder dieser Zellen regulierte ein anderer exakt 80 Basenpaare langer Promotor die Genaktivität. So konnten diese Forscher:innen für 30 Millionen unterschiedliche Promoter-Sequenzen die individuell resultierende Expressionsstärke des yfp-Gens bestimmen.
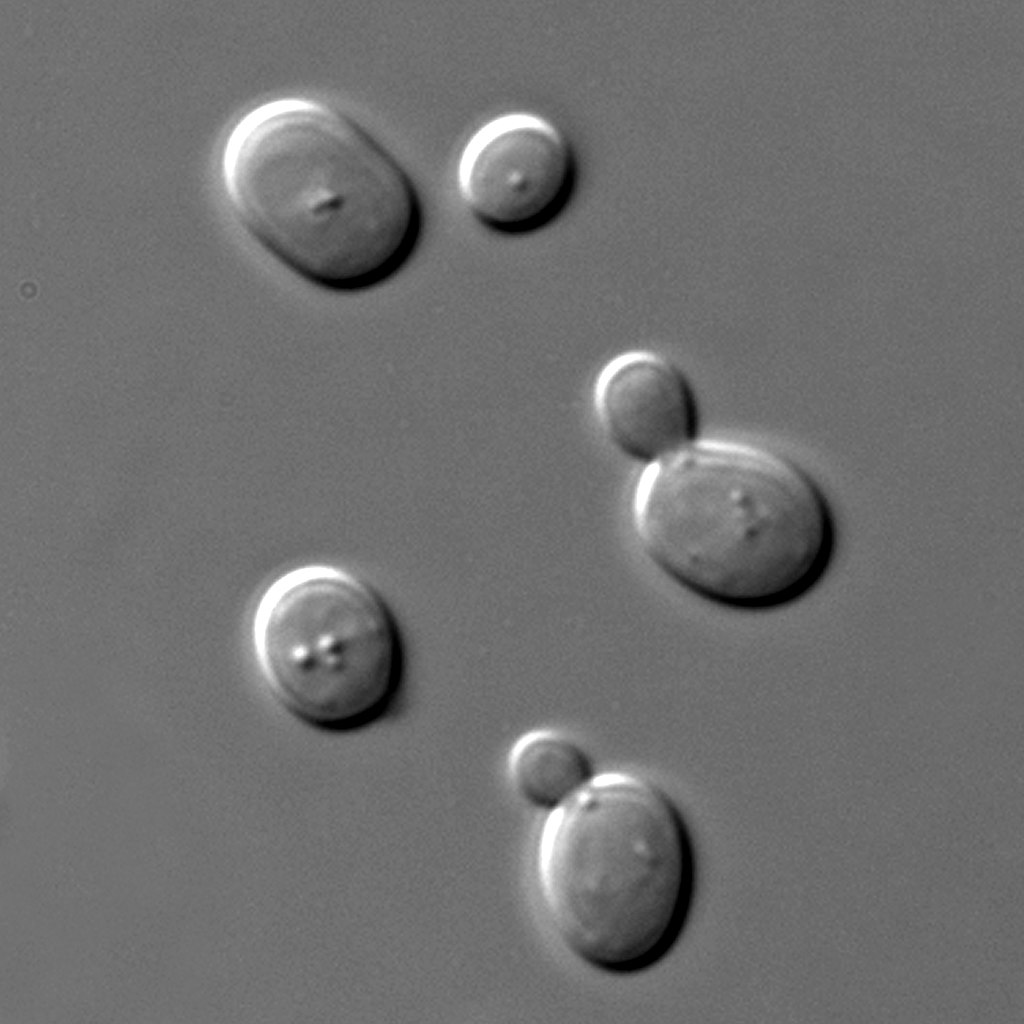
Für die Experimente hat das Team die einzellige Bäckerhefe (Saccharomyces cerevisiae) genutzt.
Bildquelle: © Masur / wikimedia.org / Public domain
Prognosefehler halbiert
Für einen Menschen wäre es kaum möglich, daraus Gesetzmäßigkeiten abzuleiten. Die Forscherinnen und Forscher haben sich jedoch nicht selbst an diese Aufgabe gemacht, sondern ein „Convolutional Neural Network“, eine bestimmte Form der Künstlichen Intelligenz, mit diesen Daten gefüttert. Anschließend prüfte das Forschungsteam, um die KI tatsächlich aus den Daten Gesetzmäßigkeiten ableiten konnte. Dazu generierten die Fachleute mehrere Zehntausend zufällige Promotorsequenzen, ließen die KI die Expressionsstärke für jede Promotorsequenz vorhersagen und glichen diese experimentell ab. Dabei zeigte sich, dass die Prognosen des neuronalen Netzwerks mit einem Korrelationkoeffizienten von 0,96 tatsächlich eine sehr hohe Genauigkeit aufwiesen. Gegenüber bisherigen konventionellen Vorhersagemodellen hat sich der Fehler damit nahezu halbiert.
Schon drei Mutationen maximieren die Expression
Die Studie ging im Anschluss noch einen Schritt weiter. Das Forschungsteam präsentierte der KI weitere zufällige Sequenzen als Promotoren und nutzte die Vorhersagefähigkeit des Programms, um in zehn simulierten Evolutionsschritten die Sequenzen so zu verändern, dass sie entweder eine extrem starke oder extrem geringe Expression bewirkten. 500 dieser Sequenzen überprüften die Forscherinnen und Forscher im Experiment – und fanden erneut die Vorhersage mit hoher Zuverlässigkeit bestätigt. Bei rund einem Fünftel der so erzeugten Promotoren übertraf die Genaktivität die aller ursprünglichen Sequenzen. Weitere Untersuchungen zeigten, dass meist bereits drei oder vier Mutationen einer beliebigen Ausgangssequenz als Promotor ausreichend waren, um eine sehr hohe oder sehr niedrige Expression zu erzielen.
Eine weitere Erkenntnis ist evolutionsbiologisch bemerkenswert. Demnach hat die Evolution rund 70 Prozent der Hefegene so „stabilisiert“, dass einzelne Mutationen nicht zu einer erheblichen Veränderung der Expressionsstärke führen, sondern lediglich zu graduellen Unterschieden.
Nur an Einzellern erprobt
Auch für die Pflanzenforschung und Pflanzenzüchtung verspricht dieser Ansatz Potenzial. Denkbar wäre, Gene mit einer gewünschten Expressionsrate „auf dem Reißbrett“ zu designen. Zum gegenwärtigen Zeitpunkt gibt es jedoch drei Einschränkungen: Erprobt ist die Methode nur an einem Einzeller. Die Genregulation in höheren Organismen ist weit komplexer, sodass sich erst zeigen muss, ob die KI auch unter diesen Bedingungen so belastbare Vorhersagen tätigen kann – zumal hier die Expression auch gewebeabhängig sein könnte.
Zweitens sind Promotoren generell nicht die einzigen sequenzabhängigen Faktoren, die die Expression beeinflussen. Und nicht zuletzt geht mit der Vorhersage durch die KI keinerlei Erklärung einher, wie es zur jeweiligen Expressionsrate kommt. Mehr logisches Verständnis über den Zusammenhang zwischen Promotor-Sequenz und Expressionsrate kann ein solcher Ansatz daher nicht liefern.
Quelle:
Vaishnav, E.D. et al. (2022): The evolution, evolvability and engineering of gene regulatory DNA. In: Nature, 603, 455–463, (9. März 2022), doi: 10.1038/s41586-022-04506-6.
Zum Weiterlesen auf Pflanzenforschung.de:
- Regulation der Genaktivitäten besser verstehen - Prognosemodelle für Transkriptionsfaktoren werden zunehmend genauer
- Ferngesteuert - Auch Pflanzen nutzen CREs für die Genregulation
- Das zweite Gedächtnis - Epigenetischer Code für Stressabwehr ist vererbbar
Titelbild: Das Team entwickelte ein neuronales Netzwerkmodell, mit dem vorhergesagt werden kann, wie sich Änderungen an nicht kodierenden DNA-Sequenzen in Hefe auf die Genexpression auswirkt. Das Modell erstellt die hier gezeigten Karten. (Bildquelle: © Martin Krzywinski)
