Die Emanzipation des Embryos
De-novo-Silencing nach Epigenom-Reset aufgeklärt
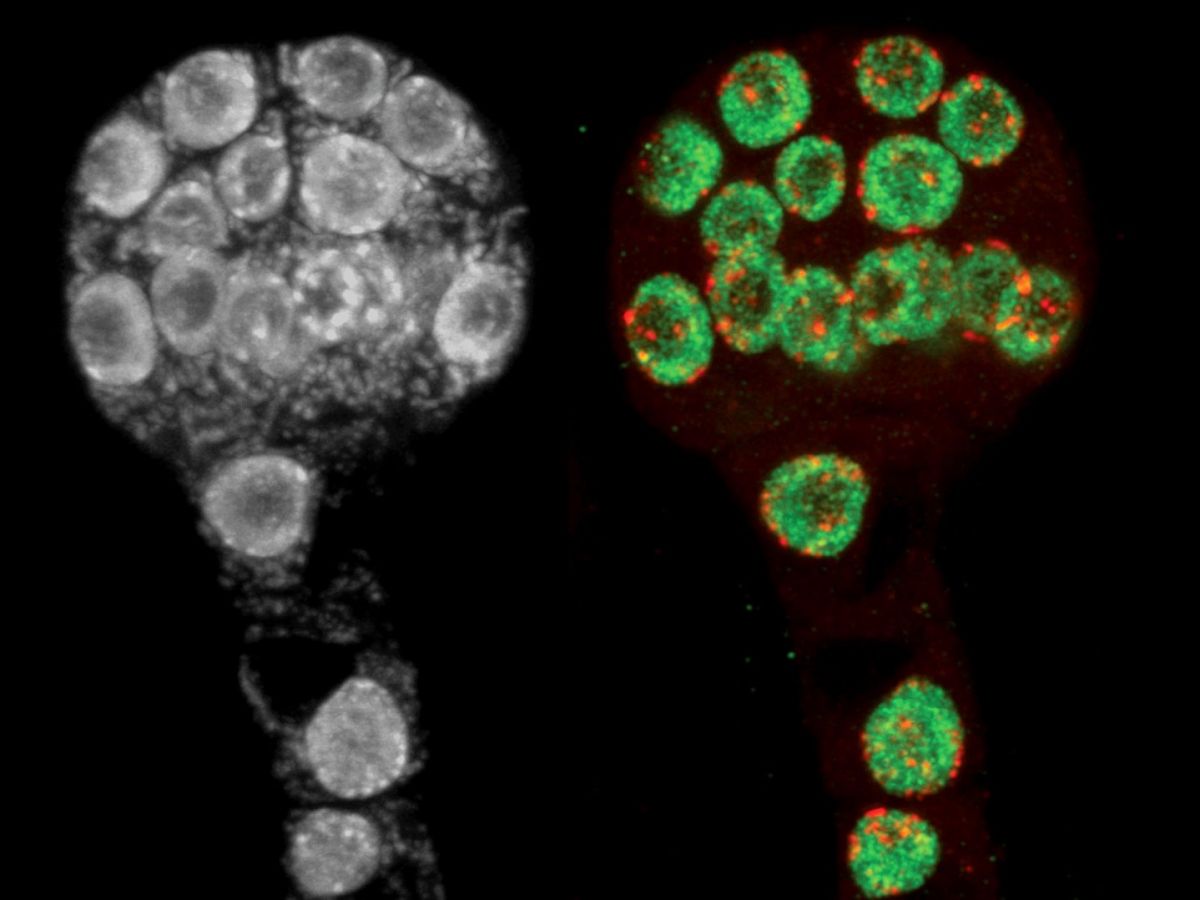
Die Fotos zeigen frühe Embryonen der Ackerschmalwand mit epigenetischen Veränderungen in grün und rot rechts bzw. grau links. (Bildquelle: © Daniel Grimanelli and Jean-Sébastien Parent/Martienssen lab, CSHL/2021)
Während der Embryogenese löschen junge Pflanzen epigenetische Modifikationen ihrer Eltern aus dem Erbgut. Dies macht den Weg frei für eine ungestörte Entwicklung. Doch in diesem „frischen“ Genom müssen für eine reibungslose Embryogenese auch gleich wieder einige Gene epigenetisch stillgelegt werden. Eine wichtige Rolle spielen dabei das Gen SUVH9 und kurze RNA-Moleküle. Die Wissenschaftler sehen im gefundenen Mechanismus auch eine Begründung, warum die Artenvielfalt der Blütenpflanzen so außerordentlich hoch ist.
Im Laufe eines Pflanzenlebens kommt es zu zahlreichen epigenetischen Veränderungen im Erbgut. Bestimmte DNA-Sequenzen werden methyliert und so Genaktivitäten dauerhaft abgeschaltet. Diese Modifikationen sind Folge der erlebten Umwelteinflüsse wie Klima, Krankheiten oder Schädlingsbefall, auf die die Pflanzen reagieren. Für die Nachkommen der Pflanzen sind einige dieser epigenetischen Modifikationen jedoch schädlich, da manche der stillgelegten Gene im Jugendstadium wieder benötigt werden. Als Embryo kann es daher überlebenswichtig sein, sich von den Eltern wieder „zu emanzipieren“: Während der Embryogenese entfernen junge Pflanzen zahlreiche epigenetische Modifikationen, müssen aber auch einige Gene für ihren Entwicklungsprozess erneut stilllegen.

Ohne das Protein SUVH9 können Embryonen der Ackerschmalwand elterliche epigenetische Veränderungen nicht zurücksetzen und haben dadurch Nachteile in der Entwicklung.
Bildquelle: © Parent/Martienssen lab, CSHL/2021
Gegen epigenetischen Kollateralschaden
„Wenn Sie an einen Baum denken, an die Blüten, die hundert Jahre nach der Keimung entstehen, dann sind diese offensichtlich einen langen Weg von der ursprünglichen Eichel entfernt und eine furchtbare Menge epigenetischer Veränderungen könnte in dieser Zeit geschehen sein“, erläutert Forschungsleiter Rob Martienssen vom Cold Spring Harbor Laboratory. Der Reset des Embryos sei daher wichtig, um nicht „epigenetischen Kollateralschaden“ zu erben.
Unklar war bislang jedoch, wie es dem Embryo gelingt, nach dem Reset die richtigen Stellen der DNA zu identifizieren, die erneut methyliert werden müssen. Wie dieses De-novo-Silencing funktioniert, konnten Forscher nun am Beispiel eines wichtigen Gens der Modellpflanze Arabidopsis thaliana zeigen.
In Eukaryoten ist das sogenannte konstitutive Heterochromatin gekennzeichnet durch die Anwesenheit von methylierten Cytosinen und der Zwei- oder Dreifachmethylierung der Aminosäure Lysin 9 im Histon H3 (H3K9). Um die Methylierung aufrecht zu erhalten, bindet normalerweise – abhängig von bestimmten Basenmustern – eine von drei H3K9-Methyltransferasen an ein methyliertes Cytosin. Anschließend leiten kurze RNA-Moleküle bestimmte DNA-Methyltransferasen zu diesen methylierten Cytosinen, um die Arbeit zu vollenden.
Beim De-novo-Silencing liegen jedoch keine methylierten Cytosine mehr vor. Wie also finden die DNA-Methyltransferasen ihr Ziel? Laut Studie ist dafür eine neue Familie von H3K9-Methyltransferasen verantwortlich, die im Embryo durch kleine RNA-Moleküle zu den richtigen Cytosinen geführt werden.
Ein neuer Akteur muss her
„Es tat sich plötzlich die Tür auf für einen ganz neuen Akteur, den es im Standardmodell nicht gab“, erinnert sich Erstautor Jean-Sebastien Parent. Mithilfe von Knock-out-Mutanten, Bisulfit-Sequenzierung und unterschiedlichen biochemischen Analysen hatte das Team festgestellt, dass keine der üblichen drei H3K9-Methyltransferasen an der Zweifachmethylierung von H3K9 beteiligt ist. Gleichzeitig schienen ARGONAUTE-Proteine eine Rolle zu spielen und damit auch kurze RNA-Sequenzen, denen diese Proteine als Transporter dienen. Das wiederum verwies auf ein noch zu identifizierendes Molekül, das bislang nicht als H3K9-Methyltransferase bekannt war.

Das SUVH9-Protein könnte der Grund für die große Artenvielfalt im Pflanzenreich sein.
Bildquelle: © Capri23auto / Pixabay
In den Fokus des Forschungsteams geriet daher das Protein SUVH9. Es galt trotz der engen Verwandtschaft bislang nicht als aktive H3K9-Methyltransferase, da es zwar über die nötige SET- und Prä-SET-Domänen verfügt, ihm aber die Post-SET-Domäne fehlt. Trotzdem besitzt die als inaktiv erachtete SET-Domäne das für die Methylierung entscheidende reaktive Tyrosin.
Ein weiterer Hinweis war, dass diese augenscheinlich inaktive SET-Domäne inklusive des reaktiven Tyrosins auch in anderen Arten hochkonserviert ist – das deutet auf eine bis heute vorhandene „echte Funktion“ hin. Untersuchungen an weiteren Mutanten belegten schließlich die Beteiligung des Proteins und insbesondere des reaktiven Tyrosins am De-novo-Silencing.
Schutz vor Schäden durch Transposons
Von analogen Mechanismen in tierischen Zellen ist bekannt, dass oftmals erst die Komplexbildung mit einer zusätzlichen Untereinheit das Protein in seinen reaktiven Zustand versetzt. Und tatsächlich fanden die Forscher ein solches Molekül in Form eines kurzen RNA-Moleküls. Dieses Molekül leitet SUVH9 an die richtigen Genomstellen, wo SUVH9 dann die Methylierung einführt. Diese Stellen liegen auf Transposons und sorgen dafür, dass benachbarte Gene zur richtigen Zeit stummgeschaltet werden. Gleichzeitig verhindert die wiederhergestellte Methylierung, dass die Transposons im Genom umherspringen und dadurch andere Gene willkürlich unterbrechen.
Die Wissenschaftler glauben zudem, dass das SUVH9-Protein der Grund für die große Artenvielfalt im Pflanzenreich ist. Indem es schädliche Transposons daran hindert, andere Gene durch Insertion zu beschädigen, ermöglichte das Protein die Entwicklung unterschiedlichster Arten. Parent dazu: „Eines der großen Rätsel bei Blütenpflanzen war bis heute, wie sie es schaffen, so vielfältig zu werden und so viele verschiedene Arten so schnell in der Evolutionsgeschichte hervorzubringen. Und wir glauben, dass wir jetzt einen Teil eines molekularen Mechanismus gefunden haben, der diese Art von Flexibilität ermöglicht.“
Quelle:
Parent, J-S. et al. (2021): Small RNAs guide histone methylation in Arabidopsis embryos. In: Genes and Development, 35:1-6, (20. Mai 2021), doi: 10.1101/gad.343871.120.
Zum Weiterlesen auf Pflanzenforschung.de:
- Das zweite Gedächtnis - Epigenetischer Code für Stressabwehr ist vererbbar
- Evolution unter der Lupe - Pflanzen reagieren schneller auf Veränderungen als gedacht
- Der Zahn der Zeit - Epimutationen verraten das Alter von Bäumen
Titelbild: Die Fotos zeigen frühe Embryonen der Ackerschmalwand mit epigenetischen Veränderungen in grün und rot rechts bzw. grau links. (Bildquelle: © Daniel Grimanelli and Jean-Sébastien Parent/Martienssen lab, CSHL/2021)
