Nachschub für die Photosynthese
Wie eine Aminotransferase die Produktion von Chlorophyllvorläufern reguliert

Chlorophyll ist ein natürlicher Farbstoff, der für die grüne Farbe von Pflanzen sorgt. (Bildquelle: © Jeon Sang-O / Pixabay)
Die Photosynthese ist der vielleicht wichtigste Hebel, um den Ertrag von Pflanzen zu steigern. Ein deutsches Pflanzenforschungsteam hat nun einen zentralen Mechanismus im Syntheseweg des Chlorophyllvorläufers 5-Aminolevulinsäure aufgeklärt.
Chlorophyll, Häm, Phytochromobilin und Sirohäm: Alle diese für Pflanzen elementaren Moleküle beruhen auf der Tetrapyrrol-Biosynthese, die ihrerseits mit der 5-Aminolevulinsäure (ALA) beginnt. ALA gilt daher als limitierender Faktor dieses Synthesewegs und damit letztlich auch der Chlorophyllproduktion.
Doch auch ALA selbst unterliegt einer Limitierung, wie ein deutsches Pflanzenforschungsteam nun am Beispiel der Ackerschmalwand Arabidopsis thaliana zeigen konnte: Die GSA-Aminotransferase (GSAAT) beeinflusst, wie viel Glutamyl-tRNA-Reduktase (GluTR) frei verfügbar ist, um ALA zu erzeugen. Eine zentrale Rolle spielt dabei ein Multiproteinkomplex.

Ob die Ackerschmalwand ausreichend Chlorophyll produziert, damit sich die Blätter grün färben, hängt von einem nun erstmals beschriebenen Multiproteinkomplex im Syntheseweg der 5-Aminolevulinsäure ab.
Bildquelle: iStock.com / kovalvs
Wissenslücke im Syntheseweg
Im Groben sind die Schritte der ALA-Synthese in Pflanzen gut verstanden: Aktiviertes Glutamat in Form von Glutamyl-tRNA wird von GluTR zu Glutamat-1-Semialdehyd (GSA) reduziert. GSAAT transaminiert das Molekül zu ALA. Das Ganze geschieht in den Plastiden. Zahlreiche regulatorische Interaktionen mit GluTR konnte die Pflanzenforschung in der Vergangenheit bereits aufdecken. Doch über die Interaktionen von GSAAT hatte die Forschung bislang wenig herausgefunden.
Liegt in einer Pflanze ein Mangel an GSAAT vor, färben sich ihre Blätter gelblich – ein Zeichen des resultierenden Mangels an Chlorophyll und damit auch an ALA. Normalerweise akkumuliert sich GSAAT in den Stromata der Chloroplasten. Dort katalysiert das Enzym die Übertragung der C2-Aminogruppe von Glutamat-1-Semialdehyd an die C1-Position. Pyridoxaminphosphat initiiert diese Transaminierung.
Wie jedoch GluTR das Enzym GSAAT mit dessen Substrat versorgt, blieb unklar, denn ein Komplex aus den beiden Proteinen konnte in Pflanzen nicht nachgewiesen werden. Jetzt kennen die Forscher:innen die Erklärung dafür: Ein dritter Partner ist an diesem Proteinkomplex als verbindendes Gerüst beteiligt: das GluTR-bindende Protein (GBP). GBP besitzt die nötigen Bindungsstellen, um auf der einen Seite GluTR und auf der anderen Seite GSAAT anzulagern und so deren Interaktion zu ermöglichen. Ohne GBP sind die potenziellen direkten Bindungen der beiden Proteine zu schwach für eine Reaktion.
Schlüsselgen in zwei Isoformen
Interessanterweise gibt es in A. thaliana zwei Isoformen von GSAAT, deren Gene GSA1 und GSA2 zu 90 Prozent identisch sind. Das Forschungsteam untersuchte daher auch, inwiefern sich die beiden Enzymvarianten in ihrer Aktivität unterscheiden. Demnach ist GSA1 vor allem in trockenen Samen, Wurzeln und alternden Blättern aktiv, GSA2 hingegen in grünen Blättern und Trieben. In Knockout-Mutanten konnte GSA2 besser den Verlust von GSA1 kompensieren als umgekehrt. Außerdem fiel auf, dass das Ausschalten von GSA2 zu erhöhten Mengen an GluTR und GBP führte. Die Aktivität von GSAAT war jedoch geringer und der Chlorophyllverlust größer. Eine Überexprimierung von GSA1 konnte diese Effekte neutralisieren.
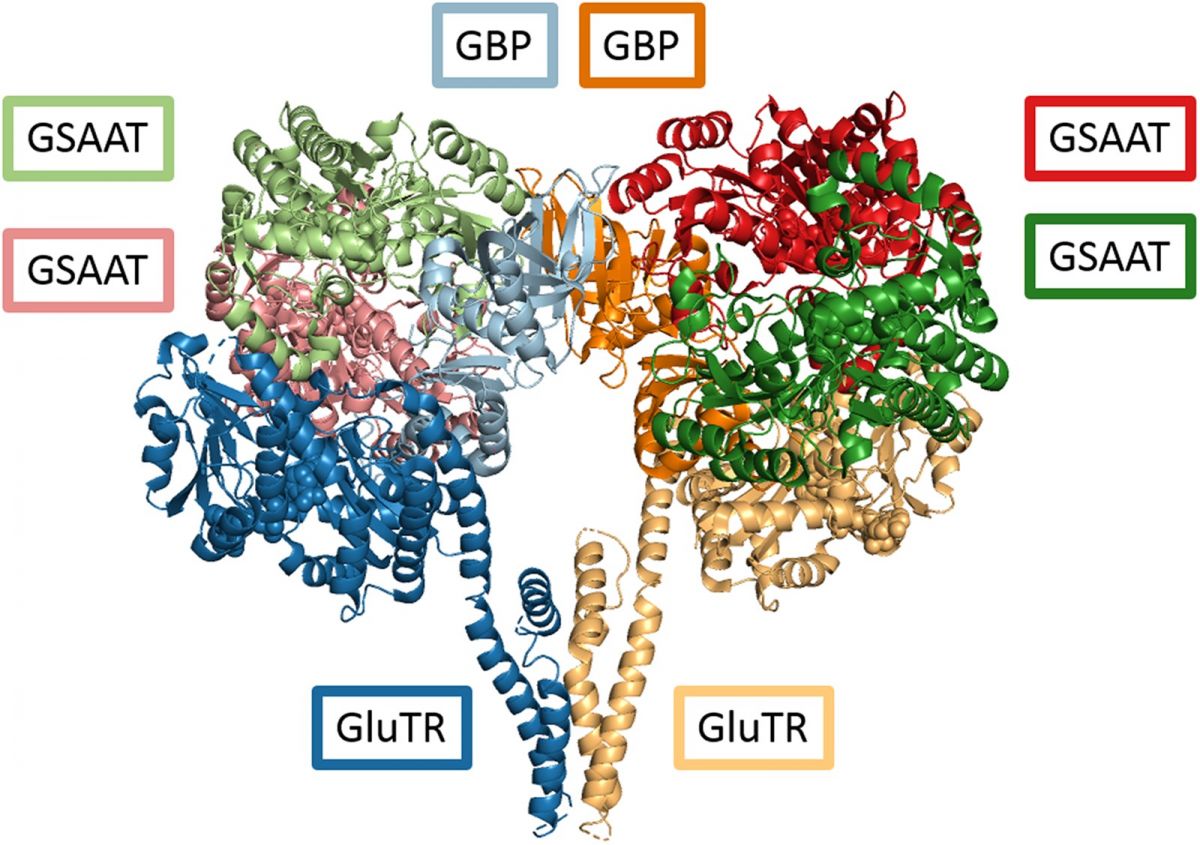
Modell des multimeren Proteinkomplexes: Pflanzen regulieren die Synthese von Chlorophyll durch eine Kontrolle der Enzyme.
Bildquelle: © Sinha et al., The Plant Cell, 2022; CC-BY
Die Forscher:innen folgern daraus, dass beide Isoformen trotz kleinerer Unterschiede wahrscheinlich funktional äquivalent sind. In jedem Fall sind beide Isoformen für eine normale ALA-Synthese und damit eine ausreichende Menge Chlorophyll erforderlich, wenngleich beide Knockout-Varianten voll funktionsfähige Photosynthese-Komplexe bildeten.
Nicht zuletzt beobachteten die Forscher:innen, dass bei einem Mangel an GSA2 überschüssiges GluTR an die Membran gebunden gespeichert wird, da GluTR und GSAAT bei der ALA-Synthese wohl in einem stöchiometrischen Verhältnis vorliegen müssen. Membrangebunden ist das Enzym geschützt vor der Proteolyse durch die Clp-Protease, die gelöstes GluTR angreift.
Freies Häm stoppt ALA-Synthese
Ob die ALA-Synthese überhaupt abläuft, hängt dabei übrigens vom Häm ab, wie eine frühere Studie zeigte: Liegt freies Häm vor, bindet dieses an den Komplex aus GluTR und GBP und löst diesen auf.
Damit hat die Pflanzenforschung einen weiteren regulatorischen Mechanismus im Zusammenhang mit der Photosynthese aufgedeckt. Dieses Verständnis bringt nicht nur die Grundlagenforschung voran: Die natürliche Photosynthese hat nur eine Effizienz von 0,5 bis 1,5 Prozent. Die Züchtungsforschung verspricht sich große Ertragssteigerung davon, sollte es gelingen, diese Effizienz signifikant zu erhöhen. Die neuen Details über den Syntheseweg des Chlorophylls können dazu ein Baustein sein.
Quelle:
Sinha, N. et al. (2022): Glutamate 1-semialdehyde aminotransferase is connected to GluTR by GluTR-binding protein and contributes to the rate-limiting step of 5-aminolevulinic acid synthesis. In: The Plant Cell, 2022:00:1-18, (16. August 2022), doi: 10.1093/plcell/koac237.
Zum Weiterlesen auf Pflanzenforschung.de:
- Photosynthese 2.0 - Von der Jagd nach mehr Effizienz bis zum künstlichen Blatt
- Photosynthese optimieren - Neuer CETCH-Zyklus erhöht die Photosyntheserate
- Scotty, weniger Energie! - Pflanzen mit Defekt im Proteasom bilden mehr Chlorophyll und sparen Energie
- Optimierte Photosynthese für höhere Erträge - Das Projekt „EnCroPho“
Titelbild: Chlorophyll ist ein natürlicher Farbstoff, der für die grüne Farbe von Pflanzen sorgt. (Bildquelle: © Jeon Sang-O / Pixabay)
