Schneller züchten dank Künstlicher Intelligenz
KI sagt Genexpressionsmuster voraus

Die KI konnte in den Genomen von Kultur- und Wildtomate Sequenzen finden, welche die unterschiedlichen Eigenschaften der Pflanzen erklären. (Bildquelle: © Lisa Redfern / Pixabay)
Ob und wann Gene aktiv sind, wird durch genregulatorische Sequenzen bestimmt. Jetzt hat ein Forschungsteam gezeigt, dass Künstliche Intelligenz erstaunlich präzise vorhersagen kann, wie genregulatorische Sequenzen auf „ihre“ Gene wirken. Das wird auch die Züchtung beschleunigen.
Das Aussehen und die Eigenschaften eines Organismus werden durch seine Gene bestimmt - so ein gerne verwendeter Lehrsatz. Aber ganz so einfach ist es dann doch nicht. Denn es kommt auch darauf an, zu welchem Zeitpunkt und in welcher Intensität diese Gene überhaupt aktiv sind, also in mRNA und dann in Proteine übersetzt werden.
Das wird unter anderem durch sogenannte cis-regulatorische Elemente (CREs) bestimmt. Dabei handelt es sich um kurze DNA-Motive, die sich in räumlicher Nähe eines Gens befinden und von Transkriptionsfaktoren (TF) gebunden werden können. Erst dann wird das Gen transkribiert, also in mRNA umgeschrieben. Und auch die mRNA selbst kann noch verschiedenen Regulationsmechanismen unterliegen: Intron-Splicing, mRNA-Kapping und Polyadenylierung sind alles Prozesse, die mitbestimmen, ob die mRNA letztendlich für die Biosynthese eines funktionalen Proteins genutzt wird.
Bisherige Methoden sind lückenhaft

Die genetische Vielfalt innerhalb einer Art (hier Mais) ist teilweise enorm. Mit Hilfe von KI könnten Zusammenhänge zwischen Genotyp und Phänotyp effizienter gefunden werden.
Bildquelle: © CLM-bv / Pixabay
Für die Züchtung neuer Pflanzensorten mit verbesserten Eigenschaften ist es wichtig, den Einfluss von CREs auf ihre Gene möglichst gut zu verstehen. Die bisherigen Methoden, mit denen ForscherInnen solche CREs ausfindig machen, sind jedoch lückenhaft. Ein Team um Dr. Jędrzej Jakub Szymański vom Leibniz-Institut für Pflanzengenetik und Kulturpflanzenforschung (IPK) hat jetzt „Deep Learning“-Modelle entwickelt: Sie können vorhersagen, wie stark bestimmte CREs die Aktivität von Genen beeinflussen. Die von den Forschern angewandte KI-Technologie ist vergleichbar mit derjenigen, die im Bereich des „Computersehens“ eingesetzt wird - wo es darum geht, Gesichtszüge in Bildern zu erkennen und auf Emotionen zu schließen.
Deep-Learning-Modelle gehören zu den KI-Algorithmen, die zunächst mit einem gewaltigen Datensatz trainiert werden. Dafür nutzten die Forscher:innen die genomischen Daten von der Ackerschmalwand (Arabidopsis thaliana), Tomate (Solanum lycopersicum), Mais (Zea mays) und Sorghum-Hirse (Sorghum bicolor). Sie fütterten den Algorithmus mit den Sequenzdaten jedes Gens inklusive der angrenzenden nicht-codierenden Genombereiche, denn dort befinden sich die CREs. Außerdem gaben sie dem Deep-Learning-Modell Daten über die Häufigkeit der einzelnen mRNA-Spezies im Blattgewebe, die aus RNA-Sequenzierungsexperimenten stammen.
Akkurate Vorhersagen durch Deep-Learning-Modelle
Bei der ersten Berechnung erhielt das Modell zunächst die Daten für einzelne Chromosomen einer Art und sollte auf dieser Basis Vorhersagen für die Genexpressionsraten der anderen Chromosomen liefern. Die Erfolgsrate dieser Einzel-Spezies-Referenz (single-species reference SSR) lag zwischen 78 und 84 Prozent. Als nächstes trainierten sie das Modell mit Daten von jeweils drei Spezies und ließen die KI für die vierte Spezies Vorhersagen treffen (multi-species reference MSR). Die Performance der MSR war ähnlich gut wie die der SSR.
„Diese Modelle waren nicht nur in der Lage, die Genaktivität anhand von Sequenzen genau vorherzusagen, sondern auch festzustellen, welche Sequenzteile diese Vorhersagen ermöglichen“, erklärt Dr. Jedrzej Jakub Szymanski.
Algorithmus funktioniert über Artgrenzen hinweg
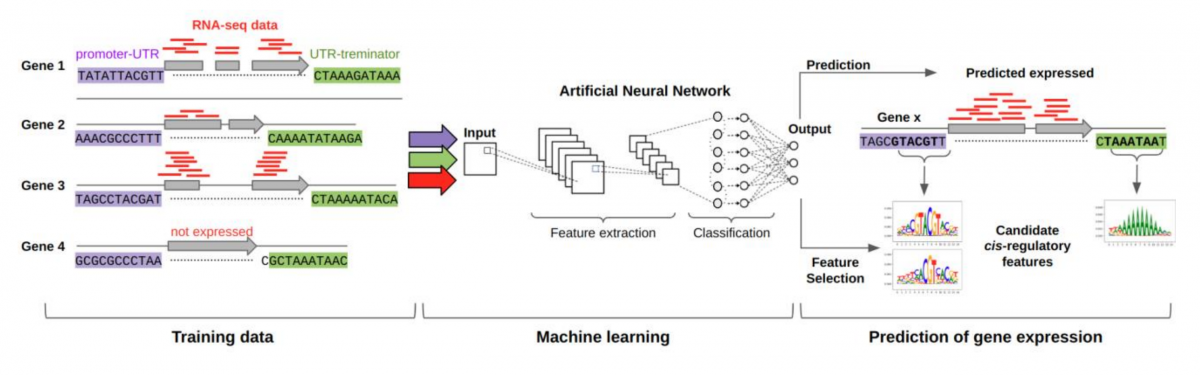
Schema der Vorgehensweise: Die KI wurde mit Sequenzdaten von Genen und deren experimentell bestimmten Transkriptionsraten trainiert. Das neuronale Netzwerk der KI konnte dann bald für andere Gene die Genexpressionsraten vorherbestimmen.
Bildquelle: © IPK Gatersleben
Die Genauigkeit sank etwas, wenn die SSR-Modelle Aussagen über eine andere Spezies treffen sollten. Am schlechtesten schnitten dabei die Sorghum-Hirse-Modelle ab, die Aussagen über Tomate treffen sollten (66 %). Das deutet darauf hin, dass es in jeder Art zwei Klassen von regulatorischen Sequenzen gibt: artspezifische und konservierte CREs, die also über Artgrenzen hinweg vorhanden sind.
Doch 66 Prozent Genauigkeit sind immer noch beachtlich. „Wir waren wirklich erstaunt über die Effektivität. Innerhalb weniger Tage Training haben wir viele bereits bekannte regulatorische Sequenzen wiederentdeckt und festgestellt, dass etwa 50 Prozent der identifizierten Sequenzmerkmale völlig neu waren. Die Modelle ließen sich sogar hervorragend auf Pflanzenarten anwenden, für die sie nicht trainiert wurden. Das macht sie für die Analyse neu sequenzierter Genome so wertvoll“, sagt Dr. Jedrzej Jakub Szymanski.
Überzeugende Ergebnisse auch bei nah verwandten Arten
Als letztes sollte der Algorithmus zeigen, ob er auch Unterschiede zwischen nah verwandten Arten aufdecken kann, obwohl er vorher nicht mit Daten dieser Spezies trainiert worden war. Dazu verwendeten die Forscher:innen das MSR-Modell, das mit Daten von Arabidopsis, Sorghum und Mais trainiert wurde und ließen es die Genome der kultivierten Tomate (Solanum lycopersicum) und der Wildtomate (Solanum penellii) vergleichen.
Tatsächlich gelang ihm das sehr gut. Obwohl die Genome sich sehr ähneln, konnte der Algorithmus zahlreiche phänotypische und metabolische Unterschiede der Pflanzen auf bestimmte regulatorische Sequenzvariationen in den jeweiligen Genomen zurückführen.
Modell ist öffentlich nutzbar
Das Team hat seine Arbeit in der Zeitschrift „Nature Communications“ veröffentlicht und gleichzeitig die Modelle öffentlich zugänglich gemacht. Über eine Webschnittstelle können die Modelle auch von anderen Forschungsgruppen direkt genutzt werden.
Dr. Simon Zumkeller, Mitautor und Evolutionsbiologe am FZ Jülich, sagt: „Die von uns vorgestellten Analyseansätze bieten Möglichkeiten, die Genregulation in Pflanzen besser und sogar auf evolutionärer Ebene zu untersuchen. Auch für die praktische Anwendung gibt es mit der von uns beschriebenen Methode eine neue Basis. Mit ihr nähern wir uns der routinemäßigen Identifizierung regulatorischer Genelemente in bekannten und neu sequenzierten Genomen, in verschiedenen Geweben und unter verschiedenen Umweltbedingungen.“ Die Forscher:innen sind sich sicher: Die neuen KI-Verfahren erweitern die Möglichkeiten, „intelligente Nutzpflanzen“ mit verbesserter Qualität und geringeren negativen Auswirkungen auf die Umwelt zu züchten.
Quelle:
Peleke, F.F., Zumkeller, S.M., Gültas, M. et al. Deep learning the cis-regulatory code for gene expression in selected model plants. Nat Commun 15, 3488 (2024). https://doi.org/10.1038/s41467-024-47744-0
Zum Weiterlesen auf Pflanzenforschung.de:
- Raps trifft auf Künstliche Intelligenz – Das Projekt „DeepIntegrate“
- Genom-Editierung wird präziser – Künstliche Intelligenz verbessert die Erfolgsrate beim Prime Editing
- Exakte Ertragsprognosen durch KI – Maschinelles Lernen kann sogar neue Züchtungsziele identifizieren
Titelbild: Die KI konnte in den Genomen von Kultur- und Wildtomate Sequenzen finden, welche die unterschiedlichen Eigenschaften der Pflanzen erklären. (Bildquelle: © Lisa Redfern / Pixabay)
