Kampf gegen Pilze und Viren
Das Projekt „IdeMoDeResBar“ will die Gerste stärker machen

Die Gerste steht im Fokus des BMBF-Forschungsprojektes IdeMoDeResBar. (Bildquelle: © Robert Hoffie)
Nicht alle Pflanzen sind Krankheitserregern schutzlos ausgesetzt, manche haben wirkungsvolle Abwehrstrategien entwickelt. Sie besitzen Resistenzgene im Erbgut, die sie gegen die Angreifer immun machen. Genau diese Gene sucht das Konsortium des Forschungsprojekts „IdeMoDeResBar“, um damit moderne Gerstensorten gegen Viren und Pilze zu wappnen.
Pusteln an den Stängeln oder Flecken auf den Blättern sind deutliche Hinweise, dass Gerste (Hordeum vulgare) von einem Schaderreger befallen ist. Pilze und Viren verursachen zahlreiche Krankheiten, die teils zu dramatischen Schädigungen und Ertragseinbußen führen. Im Kampf gegen diese Bedrohungen hilft die Sichtung und Analyse der natürlichen genetischen Vielfalt des Getreides: Viele Akzessionen sind von Natur aus widerstandsfähiger, was sie einer Vielfalt von Verteidigungsgenen verdanken, die sich im Laufe der Evolution über fortlaufende gegenseitige Anpassungen zwischen Pflanzen und ihren Pathogenen herausgebildet hat.

Diese infizierte Gerstenpflanze zeigt deutliche Symptome der Rhynchosporium-Blattfleckenkrankheit.
Bildquelle: © LfL, Sonja Eibel
Einige dieser Gene sind bereits bekannt, aber noch sind nicht alle Zusammenhänge auf molekularer Ebene geklärt. Hinzu kommt, dass die Umwelt und das Zusammenspiel zwischen Schaderregern und Pflanzen sehr komplex sind. Es gibt beispielsweise verschiedene Rassen oder Typen eines Krankheitserregers und ein Resistenzgen schützt die Pflanze nicht immer gegen alle Erregertypen. Erschwerend kommt hinzu, dass Erreger die schützende Wirkung eines Resistenzgens durchbrechen können. „Und üblicherweise wird eine Pflanze gleichzeitig von mehreren Pathogenen bedroht“, betont der Pflanzenzüchtungsforscher Jörg Schondelmaier. Darunter leidet der Ertrag.
Um Resistenzmechanismen und dafür verantwortliche Gene bei der Gerste detailliert zu untersuchen, haben mehrere Forschungspartner ihre Expertise im Verbundprojekt „IdeMoDeResBar“ gebündelt. Dieses Akronym leitet sich vom englischen Projekttitel „Identification, modification and deployment of genetic factors controlling resistance to important pathogens in barley“ ab. IdeMoDeResBar wird vom Bundesministerium für Bildung und Forschung (BMBF) im Programm „Pflanzenzüchtungsforschung für die Bioökonomie“ gefördert. Es befindet sich derzeit in der zweiten Projektphase, die noch bis Anfang 2023 läuft. Die erste Phase lief von 2016 bis 2019.
Partner und Ziele des Projektes
- Saaten-Union Biotec GmbH: Dr. Jörg Schondelmaier (Projektkoordinator)
- Leibniz-Institut für Pflanzengenetik und Kulturpflanzenforschung (IPK) Gatersleben: Dr. Jochen Kumlehn, Robert Hoffie
- Julius Kühn-Institut (JKI) – Institut für Resistenzforschung und Stresstoleranz: Prof. Dr. Frank Ordon, Dr. Dragan Perovic, Yaping Wang, Yu Cai, Claudia Breitkreuz
- Bayerische Landesanstalt für Landwirtschaft (LfL): Dr. Günther Schweizer, Dr. Bianca Büttner, Sonja Eibel, Petra Greim
- Forschungszentrum Jülich (FZJ): Prof. Dr. Björn Usadel (nur Phase 1)
Im Forschungsprojekt werden wirtschaftlich relevante Gerstenkrankheiten, die verantwortlichen Krankheitserreger und dagegen gerichtete Resistenzgene genauer erforscht:
- Gerstengelbmosaikvirosen, ausgelöst durch BaYMV (Barley yellow mosaic virus) und BaMMV (Barley mild mosaic virus), die von der bodenbürtigen, pilzähnlichen Mikrobe Polymyxa graminis L. auf Pflanzen übertragen werden,
- Zwergrost, verursacht durch den Rostpilz Puccinia hordei und die
- Rhynchosporium-Blattfleckenkrankheit, die auf den Schadpilz Rhynchosporium commune zurückgeht.
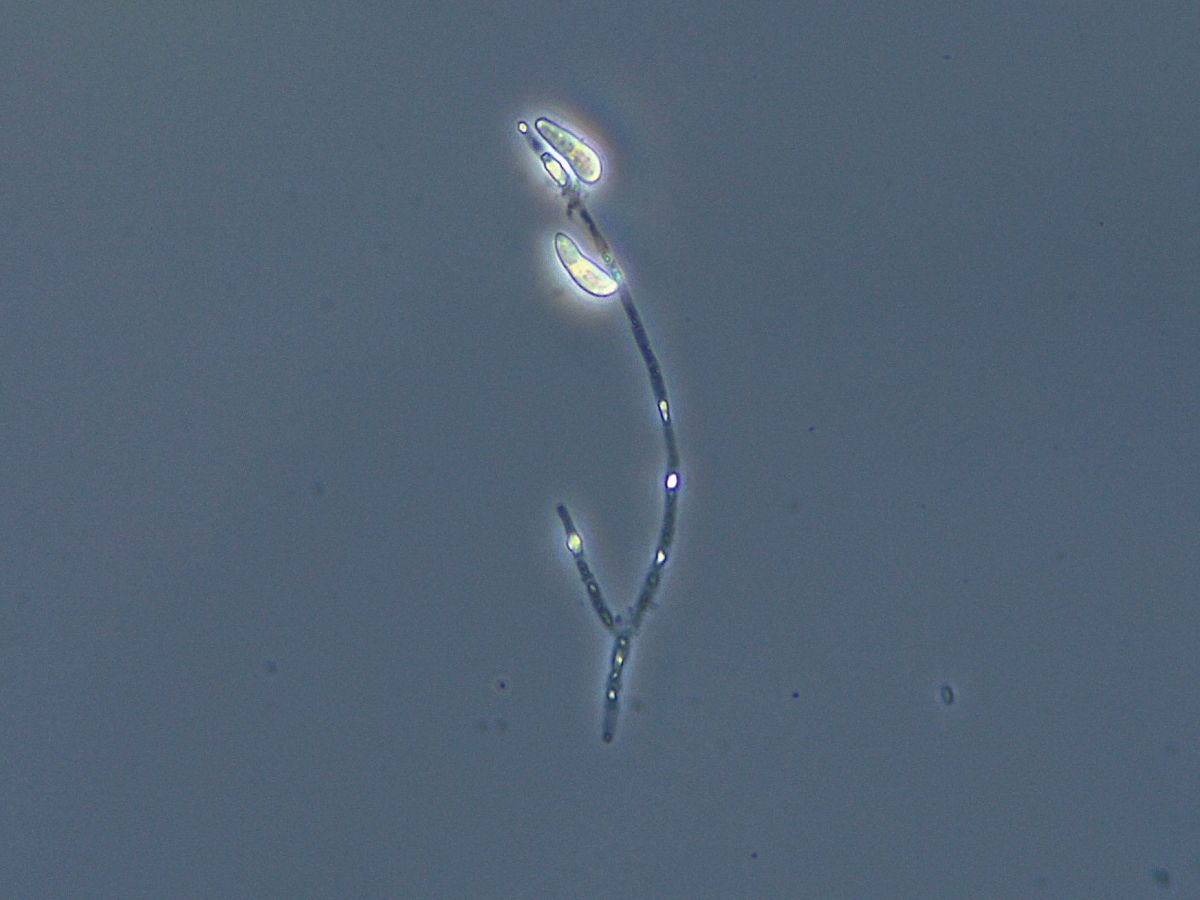
Sporen des Pilzes Rhynchosporium commune: Nicht immer sind Infektionen auch äußerlich sichtbar. Dann hilft nur eine Analyse im Labor.
Bildquelle: © LfL, Sonja Eibel
„Viele Infektionen sind gar nicht so einfach nachzuweisen, oft fehlen klare äußerliche Kennzeichen an den Pflanzen. Das ist ein Grund, warum die Forschung auf diesem Gebiet viel Zeit beansprucht. Denn man benötigt molekulare Nachweismethoden“, erklärt Projektleiter Schondelmaier.
Übergeordnetes Ziel ist es, neue Kandidatengene, neue resistenzvermittelnde Genvarianten (Allele) und geeignete molekulare Selektionsmarker für resistenzvermittelnde Gene zu identifizieren, die in der Gerstenzüchtung eingesetzt werden können.
Das Vorgehen
Mehrere Gene im Blick
Das Julius Kühn-Institut (JKI) für Resistenzforschung und Stresstoleranz in Quedlinburg hat zwei Resistenzgene im Blick: In einer serbischen Landrasse wurde das Gen RphMBR1012 identifiziert, dass die Pflanze vor Zwergrost schützt. Eine Variante eines zweiten Gens wirkt gegen Barley mild mosaic virus (BaMMV) und nennt sich rym15. Sie stammt aus einer japanischen Gerstensorte.
Die Bayerische Landesanstalt für Landwirtschaft arbeitet am Resistenzgen Rrs1, das eine spanische Gerstenlandrasse vor der Rhynchosporium-Blattfleckenkrankheit bewahrt. Schließlich kommt noch das Team vom IPK in Gatersleben hinzu, das sich auf mehrere Resistenzgene gegen das Gelbmosaikvirus wie rym11 konzentriert.
Resistenzgene im Genom verorten
Bei Resistenzgenen weiß man zunächst nicht, wo sie sich im Genom befinden. Doch genau das ist wichtig, um später solche Resistenzgene gezielt in andere Sorten einkreuzen zu können. Der erste Schritt ist daher eine sogenannte Kartierung. Die Projektpartner nutzen für die Feinkartierung der Resistenzgene das aktuellste Gerstenreferenzgenom. Dabei wird die Stelle des Gens im Genom immer weiter eingegrenzt. So werden schließlich relevante Kandidatengene für eine Krankheitsresistenz identifiziert und einem Genort im Gestengenom zugeordnet.
Im Anschluss werden die Kandidatengene einzeln untersucht. Für ihre Identifizierung werden neuste Sequenziermethoden mit anschließenden bioinformatischen Analysen eingesetzt. Ob es sich tatsächlich um die gesuchten Resistenzgene handelt, zeigen nachfolgende phytopathologische Experimente.
Resistenzmechanismen verstehen
Dafür werden die Kandidatengene auch mit Methoden der Genomeditierung (CRISPR/Cas-System) untersucht und deren Gensequenz gezielt bearbeitet. Für die Überprüfung der Genfunktion werden die per Genomeditierung modifizierten Pflanzenzellen zunächst zu Keimlingen angezogen und nun gezielt mit dem Pathogen infiziert, beispielsweise mit Sporen eines Rostpilzes. Hat sich die Resistenzreaktion des Keimlings gegen den Erreger verändert, so haben sie ein relevantes Resistenzgen für die Pflanzenzüchtung gefunden.
Wichtig ist auch, die genaue Funktion der Resistenzgene aufzuklären, z. B. in welche Stoffwechselwege der Pflanze das Gen eingreift. Dies erfolgt durch Expressionsanalysen der Kandidatengene, mikroskopische Untersuchungen der Wirt-Pathogen-Interaktion und eine Bestätigung der Kandidatengene durch das beschriebene Verfahren der Genomeditierung gepaart mit Infektionsversuchen.
Für nachgewiesene Resistenzgene erstellen die Beteiligten dann einfach handhabbare molekulare Selektionsmarker. Mit ihnen können Züchter bei Kreuzungen nun schnell verfolgen, ob das benötigte Resistenzgen in den Nachkommen vorhanden ist.
Bereits in der ersten Phase konnten einige Resistenz-relevante Regionen des Gerstengenoms identifiziert und molekulare Marker entwickelt werden. Diese Arbeiten werden nun in der zweiten Phase fortgeführt, um zusätzliche Marker und weitere Kandidatengene zu finden.
„Anfälligkeitsgene“ gezielt ausschalten
Am IPK in Gatersleben wurde die CRISPR/Cas9-Technologie auch eingesetzt, um ein „Anfälligkeitsgen“ gezielt zu verändern – dieses Gen ermöglicht es Gelbmosaikviren, ihren Wirt erfolgreich zu besiedeln. Die Idee dahinter: Verschließt man das „Einfallstor“, kann das Pathogen der Pflanze nicht mehr schaden.
Die im Labor zusammengesetzte Genschere wurde dabei mithilfe eines bakteriellen Vektors in die Gerstenzellen gebracht. Ziel war es, den an der Umsetzung von Erbinformation in ein Protein beteiligten Faktor EIF4E auszuschalten. Dieser wird vom Virus für die Vermehrung und Verbreitung seines Erbguts benötigt. Das Team konnte in der ersten Projektphase zeigen, dass sie die Methode bei Gerste anwenden können und die Pflanzen dadurch widerstandsfähig werden (Hoffie et al., 2021). Dieser Ansatz belegt das prinzipielle Potenzial, resistenzvermittelnde Allele von bestätigten Kandidatengenen ohne langwierige Kreuzung für die Züchtung nutzbar machen zu können.
Im zweiten Teil wird eine neue molekularbiologische Methode, das sogenannte Base Editing (z. Dt. Basen-Editierung), genutzt. Bei diesem Ansatz wird an einer bestimmten Stelle eine Base der DNA durch eine andere ersetzt und durch diese Punktmutation eine andere Genvariante (Allel) hervorgebracht. Das Team möchte damit gezielt neue Allele von Resistenzgenen erzeugen, um der Resistenzbildung bei den Krankheitserregern entgegenwirken zu können, ohne das Gen komplett auszuschalten.
Markerbasierte Züchtung
Der wirtschaftliche Partner des Projektes, die Saaten-Union Biotec GmbH, kreuzt die Resistenzgene in sein Sortenmaterial ein. Mithilfe der markergestützten Selektion können die Pflanzen nun bereits in frühen Generationen selektiert werden, die über die gewünschte molekulare Ausstattung verfügen. Die Resistenzeigenschaften dieser Elitepflanzen prüft das Team anschließend in Feldversuchen, also unter realen Anbaubedingungen.
Einige Resistenzgene, die unter kontrollierten Bedingungen mit nur einem Pathogen im Labor erfolgreich eine Infektion abwehren konnten, zeigten sich bei den Freilandversuchen als unwirksam. Das Team um Schondelmaier hofft, in der zweiten Phase weitere praxisrelevante Resistenzgene in das Zuchtmaterial einkreuzen und anhand der entwickelten Marker selektieren zu können.
Ausgewählte Publikationen aus dem Projekt:
- Wang, Y. et al. (2022): High-Resolution Mapping of Barley mild mosaic virus Resistance Gene rym15. In: Front. Plant Sci., 13:908170, (2. Juni 2022), doi: 10.3389/fpls.2022.908170.
- Hoffie, R.E. et al. (2021): Targeted Knockout of Eukaryotic Translation Initiation Factor 4E Confers Bymovirus Resistance in Winter Barley. In: Frontiers in Genome Editing, (29. November 2021), doi: 10.3389/fgeed.2021.784233.
- Wang, Y. et al. (2021): Delineating the elusive BaMMV resistance gene rym15 in barley by medium-resolution mapping. In: Molecular Breeding 41: 76, (2. Dezember 2021), doi: 10.1007/s11032-021-01270-9.
- Fazlikhani, L. et al. (2019): High Resolution Mapping of RphMBR1012 Conferring Resistance to Puccinia hordei in Barley (Hordeum vulgare L.). In: Front. Plant Sci., (22. Mai 2019), doi: 10.3389/fpls.2019.00640.
Zum Weiterlesen auf Pflanzenforschung.de:
- „Kontroversen sind gut, wenn sie konstruktiv sind“ - Interview mit Robert Hoffie
- Kampfstrategien gegen Viren - Neues Resistenzgen in Gerste gefunden
- Ros(t)ige Zeiten - Non-Host-Resistenz soll Weizen vor Blattrost schützen
- Eine grüne Zukunft gestalten - Förderprogramm „Pflanzenzüchtungsforschung für die Bioökonomie“ startet durch
Titelbild: Die Gerste steht im Fokus des BMBF-Forschungsprojektes IdeMoDeResBar. (Bildquelle: © Robert Hoffie)
PLANT 2030 vereint die vom Bundesministerium für Bildung und Forschung (BMBF) geförderten Forschungsaktivitäten im Bereich der angewandten Pflanzenforschung. Derzeit umfasst dies die nationalen Förderinitiativen: „Pflanzenzüchtungsforschung für die Bioökonomie“ und „Bioökonomie International“. Weitere Informationen finden Sie unter: PLANT 2030