TALEN übertrifft Cas9 im Heterochromatin
Das ideale Werkzeug zur Genom-Editierung hängt von der Umgebung ab
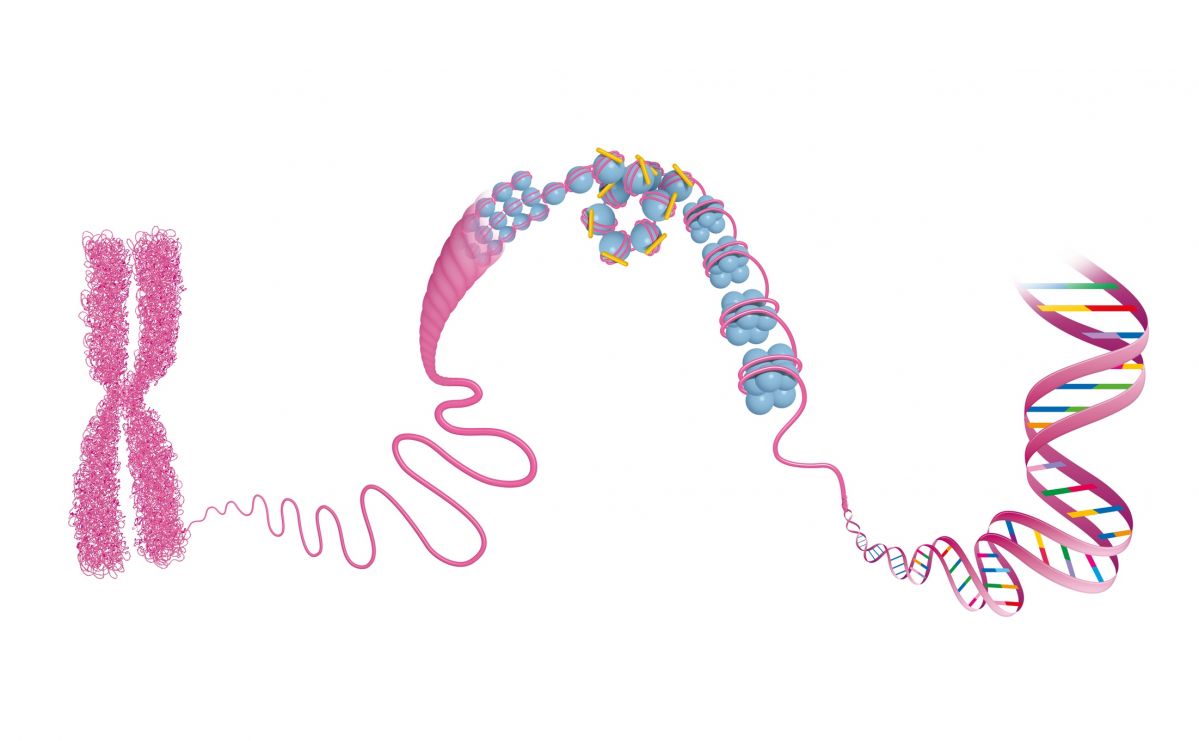
Als Chromatin bezeichnet man einen speziellen Komplex aus DNA und Proteinen, aus dem die Chromosomen bestehen. Je nachdem, wie verdichtet das Chromatin ist, sind die Werkzeuge der Genom-Editierung unterschiedlich effektiv. (Bildquelle: © iStock.com/Aldona
Die Genom-Editierung hat im vergangenen Jahrzehnt einen riesigen Techniksprung erlebt und damit auch rasante Entwicklungen in der Pflanzenzüchtung ermöglicht. Doch erst langsam wird klar, welche Werkzeuge unter welchen Bedingungen die besten sind.
Die Genom-Editierungsmethode CRISPR/Cas9 ist seit ein paar Jahren selbst Laien ein Begriff. Ihre Entdeckung wurde 2020 mit dem Chemie-Nobelpreis geehrt, so enorm ist der Einfluss auf die Genetik – von Medizin bis Pflanzenzüchtung. Entscheidend ist dabei die Spezifität, mit der die eingesetzten Enzyme im Erbgut die zu verändernde Stelle identifizieren.
Erstmals konnte ein Forschungsteam in den USA jetzt durch bildgebende Verfahren untersuchen, unter welchen Bedingungen und wie das Enzym Cas9 (CRISPR-assoziiertes Protein) besonderes spezifisch sein Ziel findet. Dabei zeigte sich, dass eine andere Methode der Genom-Editierung im hoch verdichteten Heterochromatin fünf Mal effektiver arbeitet. Sie nutzt das Enzym TALEN (transkriptions-aktivatorartige Effektornuklease).
Suchen, finden, editieren
Die Enzyme Cas9 und TALEN sind eine Art biochemische Genom-Suchmaschine, ähnlich wie Google im Internet: Sie wandern am Genom entlang, und wenn ein Abschnitt dem Suchmuster entspricht, binden sie dort. Lange genug, um dort ihre enzymatische Wirkung zu entfalten und die Sequenz zu verändern.

Die Enzyme Cas9 und TALEN sind eine Art biochemische Genom-Suchmaschine: Sie wandern am Genom entlang, und wenn ein Abschnitt dem Suchmuster entspricht, binden sie dort.
Bildquelle: © Arek Socha / Pixabay
Cas9 wird dazu mittels einer gezielt synthetisierten single guide RNA ausgestattet, die dem Molekül an der gewünschten Zielsequenz eine höchst spezifische RNA-DNA-Bindung ermöglicht. TALEN dagegen besitzt eine DNA-Bindungsdomäne aus 33 bis 34 Aminosäuren, die auch an die gewünschte Zielsequenz passgenau angepasst werden kann.
Mittels Fluoreszenzmikroskopie hat das Forschungsteam in lebenden Säugetierzellen untersucht, auf welche Weise sich die beiden Enzyme im Chromatin bewegen und welche „Suchmethode“ sie verwenden, um die passende Bindungsstelle zu finden. Lediglich die Fähigkeit, bei einem Treffer auch die DNA zu schneiden, entfernten die Forschenden für den Versuch. Die resultierenden Moleküle bezeichnete das Team als TALE und dCas9. Als Ziel wurden zwei Sequenz gewählt: eine, die im gesamten Genom nur vier Mal anzutreffen ist, und eine andere, die zu rund einer Million möglicher Treffer führt.
Wechsel zwischen lokaler und globaler Suche
Sowohl TALE als auch dCas9 zeigten jeweils zwei Formen von Suchverhalten. Die sogenannte globale Suche (Sprünge und 3-D-Diffusion) zeichnete sich durch eine hohe Diffusionsgeschwindigkeit aus, die lokale Suche (1-D-Diffusion, also ein Gleiten entlang des DNA-Moleküls) durch eine niedrige. Zwischen beiden Methoden können die Enzyme innerhalb von rund 20 Millisekunden wechseln. Die beobachteten kinetischen Parameter beider Suchproteine unterschieden sich wenig, obwohl bislang davon ausgegangen wird, dass Cas9 im wenig verdichteten Euchromatin effektiver arbeitet als TALEN.
In einem nächsten Schritt untersuchte das Team daher, für wie lange die Enzyme jeweils die Bindung mit den Zielsequenzen aufrechterhalten. Für das seltene Ziel ergab sich für TALE eine durchschnittliche Bindungsdauer von 1,8 Sekunden, für das häufige Ziel waren es 20,2 Sekunden. Außerdem kamen in beiden Fällen mehr als vier von fünf Treffern durch die globale Suche zustande. dCas9 hingegen verbrachte durchschnittlich 13,4 Sekunden an seltenen Zielsequenzen und 5,9 Sekunden an den häufigen. Auch hier hatte die globale Suche den deutlich größeren Anteil, immerhin noch drei von vier Treffer.
Unterschiede in der Bewegung
Darüber hinaus zeigte sich, dass dCas9 für einen einzelnen lokalen Suchzyklus rund 50 Prozent länger benötigt als TALEN. Ein weiterer Unterschied in der Suchdynamik besteht der Studie zufolge im Winkel, in dem sich die Enzyme innerhalb des Chromatins fortbewegen. TALE verhält sich dabei sehr unterschiedlich, was darauf hindeutet, dass das Protein durch „genomische Einschlüsse“ behindert wird. dCas9 hingegen weist ein sehr einheitliches Bewegungsmuster auf, was zu einer hohen Sucheffizienz führt. Das dürfte ursächlich für die bessere Leistung des Enzyms Cas9 im Euchromatin sein.

Das Forschungsteam um Huimin Zhao, Professor für Chemie- und Biomolekulartechnik an der University of Illinois at Urbana-Champaign (Dritter von links). Zusammengesetztes Bild aus separaten Fotos, in Übereinstimmung mit den COVID-19-Sicherheitsprotokollen.
Bildquelle: © L. Brian Stauffer
Anders verhält es sich im dicht gepackten Heterochromatin. Für beide Enzyme ist bekannt, dass ihre Leistung hier abfällt, doch die kinetischen Ursachen waren bislang unklar. Die neue Studie zeigt, dass beide Enzyme deutlich langsamer suchen, weil sie in ihrer Bewegung durch das gewundene Chromatin stärker blockiert werden. Trotzdem erwies sich TALE mit einer Suchgeschwindigkeit von 2,35 Quadratmikrometern pro Sekunde als deutlich schneller als dCas9 mit 1,93 Quadratmikrometern pro Sekunde. Offensichtlich kann TALE effizienter durch das dichte Heterochromatin manövrieren.
TALEN editiert besser im Heterochromatin, Cas9 im Euchromatin
Das spiegelte sich am Ende auch in der Editiereffizienz wider. Entwarfen die ForscherInnen TALEN- und Cas9-Moleküle, die ein Ziel im Heterochromatin schneiden sollten, zeigte TALEN an elf der möglichen zwölf Zielorte eine zumindest gleich hohe oder sogar höhere Editieraktivität als Cas9. An den vier passenden Stellen im Euchromatin hingegen war durchweg Cas9 gleichwertig oder überlegen.
Die Effizienz der Genom-Editierung ist damit bei beiden Verfahren maßgeblich vom Kontext abhängig, resümiert das Studienteam: „Wir vermuten, dass die schwerpunktmäßig lokale Suche von Cas9 im Heterochromatin keine vorteilhafte Eigenschaft ist und zu einer geringeren Schnitteffizienz führt. TALEN hingegen kann sich das Heterochromatin mit geringeren Zeitanteilen für die lokale Suche besser erschließen.“ Obwohl diese Beobachtungen bislang nur für Säugetierzellen angestellt wurden, dürften sie auf Pflanzenzellen übertragbar sein – und sind damit auch für Pflanzenforscher eine wichtige Orientierung bei der Wahl des geeigneten Werkzeugs zur Genom-Editierung.
Quelle:
Jain, S. et al. (2021): TALEN outperforms Cas9 in editing heterochromatin target sites. In: Nature Communications 12, Article number: 606, (27. Januar 2021), doi: 10.1038/s41467-020-20672-5.
Zum Weiterlesen auf Pflanzenforschung.de:
- CRISPR/Cas hat den Dreh raus - Inversionen machen Chromosomenabschnitte wieder für Rekombination zugänglich
- Effiziente Genomeditierung mit weniger Nebeneffekten - Vielversprechende Weiterentwicklungen der CRISPR/Cas9-Methode
- Genscheren-Tuning - Gewebespezifische Genomeditierung mit CRISPR
Titelbild: Als Chromatin bezeichnet man einen speziellen Komplex aus DNA und Proteinen, aus dem die Chromosomen bestehen. Je nachdem, wie verdichtet das Chromatin ist, sind die Werkzeuge der Genom-Editierung unterschiedlich effektiv. (Bildquelle: © iStock.com/Aldona)
