Rasanter technologischer Fortschritt
CRISPR/Cas9 kann Punktmutationen ohne zu schneiden korrigieren
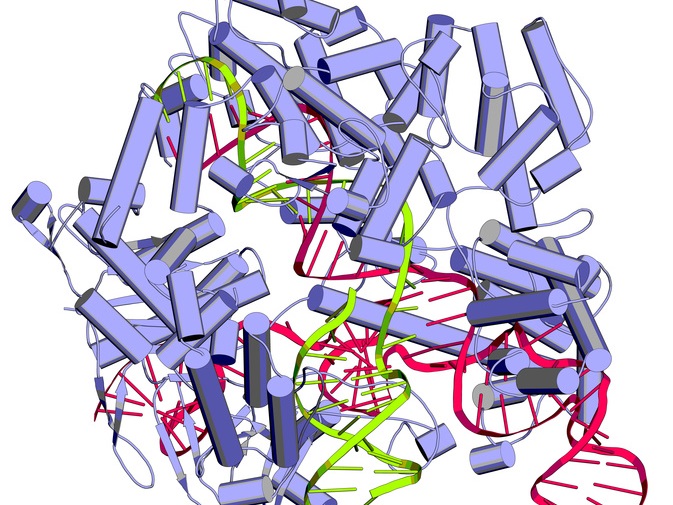
Schematische Darstellung des Proteins Cas9 (lila) zusammen mit guide RNA (pink) und DNA (grün). Forscher haben die CRISPR-Cas9-Technologie so weiterentwickelt, dass damit Punktmutationen korrigiert werden können. (Bildquelle: © molekuul.be/Fotolia.com)
Forscher konnten die noch junge Genom Editierungstechnologie CRISPR/Cas9 so anpassen, dass der Austausch einer einzigen Base möglich ist, ohne dabei den DNA-Strang zu zerschneiden. Großes Potential besteht für die Humanmedizin, denn fast alle Erbkrankheiten beruhen auf Punktmutationen. Aber auch für die Pflanzenforschung, denn auch bei Pflanzen sind es Punktmutationen, die für Resistenzen oder veränderte Synthesewege von Metaboliten verantwortlich sind. Etablierte Methoden wie das „TILLING“ erscheinen überholt. Auf Pflanzenforschung.de halten wir Sie über neue wissenschaftliche Erkenntnisse aber auch über aktuelle methodische Entwicklungen auf dem Laufenden. Im folgenden Beitrag über die Weiterentwicklung der Genom Editierung mittels CRISPR/Cas9.
Es ist noch nicht lange her, dass Forscher die neue Genom Editierungsmethode (engl.: Genome Editing) CRISPR/Cas9 vorstellten. Im Jahr 2012 wurde sie erstmals beschrieben. Seither nutzen Forscher weltweit die Technologie so fleißig, dass nicht nur unzählig Publikationen, sondern auch viele Weiterentwicklungen der Technologie veröffentlicht wurden.
Die Ereignisse überschlagen sich

Champignons sind nicht lange haltbar - sie verfärben sich schnell braun. Mithilfe der CRISPR/Cas9-Technologie gelang es Forschern eine Sorte zu entwickeln, die langsamer braun wird. Das US-amerikanische Landwirtschaftsministerium (USDA) kam nun zu dem Schluss, dass der Pilz ohne weitere Regularien zugelassen werden kann. Er gilt demnach nicht als gentechnisch veränderter Organismus.
Bildquelle: © Raysonho / wikimedia.org; CC0
Die Technologie wird bereits erfolgreich angewendet. So gelang es Forschern mithilfe von CRISPR-Cas9 einen Speisepilz - den Zucht-Champignon Agaricus bisporus - zu entwickeln, der langsamer braun wird und dadurch länger gelagert werden kann. Dabei hatten die Wissenschaftler lediglich eins von sechs Genen ausgeschaltet, die für das Enzym Polyphenoloxidase kodieren, welches für das braun werden verantwortlich ist.
Vor kurzem, genauer gesagt am 13. April 2016, hat das US-amerikanische Landwirtschaftsministerium (USDA) grünes Licht gegeben und den Champignon als Lebensmittel zugelassen, welches nicht unter die Regularien von gentechnisch veränderten Organismen fällt (USDA/APHIS, 2016). Damit ist der Pilz das erste mit CRISPR-Cas9 veränderte Lebensmittel und kann ab sofort gehandelt werden. Hintergrund für diese Einschätzung ist, dass sich der mittels CRISPR/Cas9 veränderte Pilz nicht von natürlichen Mutationen unterscheiden lässt.
Vor wenigen Tagen gab das USDA bekannt, dass eine auch mit CRISPR-Cas9 entwickelte Pflanzensorte, eine Wachsmaissorte der Firma DuPont Pioneer, ebenfalls keiner weiteren Regulation bedarf. Wachsmais unterscheidet sich von herkömmlichem Mais durch die Stärkezusammensetzung des Korns. Diese besteht ausschließlich aus Amylopektin und nicht wie üblich aus Amylopektin und Amylose (etwa 25%). Bei dieser speziellen Sorte wurde durch Genom Editierung ein Gen (Wx1) gezielt entfernt (Deletion), das die Produktion von Amylose unterstützt und somit im Wachsmais unerwünscht ist.
Bisherige Wachsmaissorten wurden über aufwendige Rückkreuzungsschritte mit natürlich vorkommenden „Mutanten“ - wie Pflanzenlinien, die ein defektes Gen enthalten genannt werden - erzeugt. Die Körner bestehen zu fast 100 Prozent aus Amylopektin, was für sehr unterschiedliche Anwendungen interessant ist. Diese verzweigten Stärkemoleküle verändern die physikalisch-chemischen Eigenschaften hinsichtlich Viskosität, Verkleisterung oder Bindung. Bei der Herstellung von Papier, Lebensmitteln oder Textilien aber auch in der Bauindustrie sind diese Eigenschaften gefragt. Bisherige energieaufwendige Trennungsschritte der beiden Stärkefraktionen entfallen beim Waxy-Mais.
Immer neue Anwendungen erprobt
Nicht nur bei Lebensmitteln und Nutzpflanzen für die industrielle Anwendung tut sich demnach einiges, vor allem in der Medizin hat man die Potentiale der Technologien erkannt. Um für die unterschiedlichen Anwendungen bestmögliche Ergebnisse zu erzielen, muss jedoch die vielversprechende Technologie weiter justiert und präzisiert werden.
Übersicht: Nukleobasen, auch Nukleinbasen
| Base: | Bestandteil von: |
| Adenin (A) | DNA und RNA |
| Cytosin (C) | DNA und RNA |
| Guanin (G) | DNA und RNA |
| Thymin (T) | nur DNA |
| Uracil (U) | nur RNA |
Forscher haben nun eine Studie veröffentlicht, in der sie CRISPR-Cas9 so anpassten, dass das molekulare Werkzeug eine einzelne Nukleobase, den Grundbausteinen der DNA und RNA, austauschen kann ohne dabei das zu tun, für das das System bisher bekannt war: den DNA-Strang in der Zelle punktgenau zu zerschneiden und anschließend mit der gewünschten Veränderung auf beiden DNA-Strängen gleichzeitig reparieren zu lassen.
Kleine Veränderung, große Wirkung
Derzeitige Genom Editierungsmethoden beruhen darauf, dass sie einen DNA-Doppelstrangbruch hervorrufen, um Gene gezielt zu verändern. Viele Merkmalsunterschiede zwischen Organismen aber auch Erbkrankheiten beim Menschen beruhen allerdings auf Punktmutationen. Das heißt, Veränderungen nur einer einzigen Base sind für diese verantwortlich. Für die gezielte Korrektur solcher Punktmutationen waren die bisherigen Methoden zu ungenau und ineffizient. Darüber hinaus schleichen sich bei den nachfolgenden zelleigenen Reparaturmechanismen solcher gezielt gesetzten Strang-Brüche Fehler ein, sogenannte Indels (Deletionen und/oder Insertionen), die nicht gewollt sind und ein unerwünschtes Ergebnis hervorrufen. Dieses Manko motivierte das Forscherteam, die bisherige CRISPR-Cas9-Technologie weiter zu verfeinern.
Direkter Austausch geglückt
Die Wissenschaftler nutzten dafür ein Cas9-Enzym, das zwar durch guide RNA (gRNA) spezifisch an die DNA binden kann, aber katalytisch inaktiv ist. Das sogenannte dCas9 (für „deactivated“) kann also keinen Doppelstrangbruch mehr verursachen, verfügt aber sonst über alle Eigenschaften. Dann kombinierten sie dies mit einem Enzym, das den Basenaustausch einer Nukleobase hervorrufen kann. Sie nutzten eine Cytidindeaminase, um die Base Cytosin (C) durch Uracil (U) zu ersetzen, was letztlich in der DNA-Sequenz zu Thymin (T) wurde. Der neue Ansatz führte zu einem direkten und irreversiblen Austausch einer DNA-Base durch eine andere.
Die Forscher verfeinerten die Methode nachfolgend noch weiter, indem sie intrazelluläre DNA-Reparaturmechanismen entweder verhinderten oder ausgetricksten, um letztlich vermehrt das angestrebte Paar T-A (als Ersatz für C-G) zu erhalten und die Rate an Indels möglichst gering zu halten.

Die Wissenschaftler testeten ihre Technologie im Labor, dafür setzten sie (in vitro, d. h. außerhalb des lebenden Organismus) in Zellen von Mäusen gezielt Punktmutationen.
Bildquelle: © JacobST / Fotolia.com
In der Zelle führt der Austausch von C zu T zunächst dazu, dass statt dem Basenpaar Adenin (A) - Thymin (T) und Cytosin (C) - Guanin (G) nun das ungleiche Paar T-G entsteht. Dies ruft einen DNA-Reparaturmechanismus auf den Plan (die Mismatch-Reparatur), die die Basenfehlpaarung korrigiert und eine der fehlerhaften Basen durch die korrekte ersetzt. Um sicherzustellen, dass der Reparaturmechanismus die „richtige“ Base – also in diesem Fall Guanin (G) – austauscht, modifizierten sie das Cas9-Protein derart, dass das Enzym am DNA-Strang, der das Guanin (G) enthält, für eine kleine Kerbe (engl.: „nick“), einen Einzelstrangbruch sorgt. Diese Kerbe garantierte, dass der Reparaturmechanismus bevorzugt Guanin (G) zu Adenin (A) austauscht und damit die gewünschte Punktmutation stabil in der Erbsubstanz einbaut wurde.
Risikofaktor für Alzheimer korrigiert
Die Wissenschaftler testeten ihre Technologie daraufhin in vitro in Zelllinien von Mäusen und zwar bei einem Allel des Gens APOE (APOE4), das zwei Punktmutationen enthält und als der größte genetische Risikofaktor für Alzheimer gilt.
Es gelang ihnen, eine der beiden Punktmutationen in bis zu 75 Prozent aller Zellen zu korrigieren, so dass sich das Krankheitsrisiko senkte. Darüber hinaus beobachteten die Forscher, dass lediglich 4 bis 6 Prozent Indels auftraten. Ein deutlich geringerer Wert als beim Einsatz der klassischen CRISPR-Cas9-Methode in einem Kontrollexperiment: In diesem traten 26-40 Prozent Indels auf. Die Weiterentwicklung der Genom Editierung war deutlich effizienter und verursachte weniger Fehler als der normale CRISPR-Cas9-Ansatz. Damit eröffnet sich die neue Möglichkeit, Punktmutationen, die Erbkrankheiten verursachen gezielt zu korrigieren.
Das Werkzeug könnte nach den vielversprechenden Ergebnissen im Mausmodell auch für andere Krankheiten geeignet sein, wie z. B. Krebs. Allerdings steht man erst am Anfang. Es ist noch ein weiter Weg von Säugetierzellen zum Menschen. Doch das Tempo, mit dem Wissenschaftler derzeit Anpassungen und Verbesserungen präsentieren ist enorm und könnte dazu führen, Technologien wie diese in der Therapie von Krankheiten einsetzen zu können. Aber auch andere Anwendungsfelder, wie die Pflanzenforschung oder die Editierung von Genomen von Mikroorganismen profitieren von diesen Weiterentwicklungen.
Quelle:
Komor, A.C. et al. (2016): Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. In: Nature, (20. April 2016), doi: 10.1038/nature17946.
Zum Weiterlesen:
- Neuer, schneller, präziser - Mit CRISPR/Cas9 zu herbizidresistenten Kulturpflanzen
- RNA Live-Tracking mit CRISPR/Cas - Forscher nutzen CRISPR/Cas-Komplex als „GPS-Sender“ für RNA
- CRISPR/Cas wird auch unsere Nahrungspflanzen verändern
- Passgenaues Immunsystem in Pflanzen - Forschern gelingt Virusabwehr mit CRISPR
- Was CRISPR/Cas leisten kann
Titelbild: Schematische Darstellung des Proteins Cas9 (lila) zusammen mit guide RNA (pink) und DNA (grün). Forscher haben die CRISPR-Cas9-Technologie so weiterentwickelt, dass damit Punktmutationen korrigiert werden können ohne die DNA zu zerschneiden. (Bildquelle: © molekuul.be/Fotolia.com)
