Methodenforschung
Rasante Entwicklung der Genom-Editierung

Genom-Editierungen ermöglichen Veränderungen an der DNA, die in der Regel nicht von natürlichen Mutationen zu unterscheiden sind. (Bildquelle: © Gerd Altmann / Pixabay)
Die EU-Kommission will den Marktzugang für bestimmte Genom-editierte Pflanzen vereinfachen. Zugleich wird die Technik immer besser und vielseitiger – ein Überblick über den Stand der Technik und noch verbleibende Hürden.
Die EU-Kommission hat im Juli 2023 einen neuen Gesetzentwurf zur Regulation gentechnisch veränderter Pflanzen vorgelegt, der erstmals auf die Unterschiede zwischen klassischer Gentechnik und moderner Genom-Editierung eingeht. Einfache genom-editierte Pflanzen könnten demnach von der Gentechnik-Zulassungspflicht ausgenommen werden und die Genschere CRISPR/Cas somit zur Züchtung widerstandsfähiger und klimaresilienter Pflanzensorten eingesetzt werden.
In den rund zehn Jahren seit der Entwicklung der Methode hat sich viel getan. Für zahlreiche Organismen sind Standardverfahren für die Genom-Editierung entwickelt worden, die Spezifität wurde verbessert, und es ist ein ganzes Spektrum verschiedener Editierungen möglich geworden – von der Punktmutation bis zur Inversion großer Chromosomenabschnitte. In rund 120 Pflanzenarten haben Forscher:innen die Methode bereits erfolgreich genutzt. Dennoch bestehen weiterhin zahlreiche Herausforderungen für Forschung und Entwicklung mittels Genom-Editierung.
Die Genom-Editierung kann heute schon ohne rekombinante DNA ablaufen und ohne verbleibende DNA-Spuren des CRISPR/Cas-Systems im Zielorganismus. Aber Verbesserungen sind vor allem im Bereich Gewebekultur und -regeneration sowie beim Mutantenscreening nötig. Auch der hohe Polyploidiegrad einiger Kulturarten macht noch zu schaffen. Schließlich haben noch Verfahren zur simultanen Editierung verschiedener Zielgene (Multiplex-Editierung) Optimierungsbedarf.
Gewebekultur, Transformation und Regeneration

Für die Pflanzenforschung haben Techniken zur Genom-Editierung ein enorm großes Potenzial. Vor allem pathogenresistente Pflanzen lassen sich schnell herstellen – dadurch kann der Einsatz von Pflanzenschutzmittel gesenkt werden.
Bildquelle: © Kenneth Rodriguez / Pixabay
Inzwischen gibt es Genotyp-unabhängige Protokolle zur Transformation und Regeneration, ebenso Tests für die Funktionalität und Effizienz der Editierungskonstrukte. Große Fortschritte bei der Gewebekultur und beim Transformationserfolg wurden durch Entwicklungsregulatoren und deren kontrollierte Expression durch induzierbare Promotoren möglich. Andere Methoden wie der chimärische Proteinkomplex GRF4-GIF1 ermöglichen die Regeneration fruchtbarer Pflanzen, ohne das spezialisierte Promotoren erforderlich sind und das Transgen ausgeschnitten werden muss.
Generell kann es bei heterozygoten, polyploiden, zweihäusigen und selbstinkompatiblen Arten schwierig sein, mittels Rückkreuzung ein Transgen wieder zu entfernen, das infolge der Transformation durch Rhizobien stabil ins Genom integriert wurde. Aktuelle Entwicklungen zielen daher daraus ab, die Cas-Nuklease nur vorübergehend zu exprimieren. Das führt zu transgenfreien genom-editieren Pflanzenlinien und verringert Nicht-Ziel-Effekte. Erreicht werden kann das beispielsweise durch Ribonukleoproteinkomplexe, die in der Zelle schnell degradieren und nicht ins Genom integriert werden. Aber auch mit DNA- und RNA-Molekülen ist diese vorübergehende Expression von CRISPR/Cas bei Mais und Weizen bereits gelungen.
Erste in vivo-Erfolge
Herausfordernd bleiben insbesondere mehrjährige Gehölze. Sie sind schwierig in vitro zu transformieren und regenerieren und wachsen nur langsam. Auch ist das Risiko hoch, Chimären zu erzeugen, in denen nur ein Teil der Pflanze von der editierten Zelle abstammt. Ähnlich ist das Problem bei hoch regenerativem Gewebe polyploider Pflanzen, in die der Transfer von CRISPR/Cas oft ineffektiv ist. Allerdings können hier erfolgreich transformierte Zellen mittels Fluoreszenz-aktivierter Zellsortierung selektiert werden.
Generell entwickelt die Forschung deshalb zur Transformation und Regeneration in planta-Methoden, die unabhängig vom Genotyp funktionieren. Bislang sind diese Ansätze jedoch nur für wenige Pflanzenarten in einem fortgeschrittenen Entwicklungsstadium. In ein paar wenigen Arten ist es außerdem gelungen, das Editier-System in vivo ins Meristem zu verabreichen, sodass die Editierung an die Zellnachkommen übertragen werden kann.
Erprobt werden derzeit neue virale Vektoren, die auch größere Sequenzinformationen wie für Cas9 beherbergen und die Keimbahn infizieren können. Diskutiert werden außerdem Nanopartikel als Transporter in der gewebekulturfreien Genom-Editierung. Doch bislang verhindert die Größe der Nukleasen einen Transport durch die Zellwand hindurch und damit die Editierung der Keimbahn.
Erfolgskontrolle der Genom-Editierung
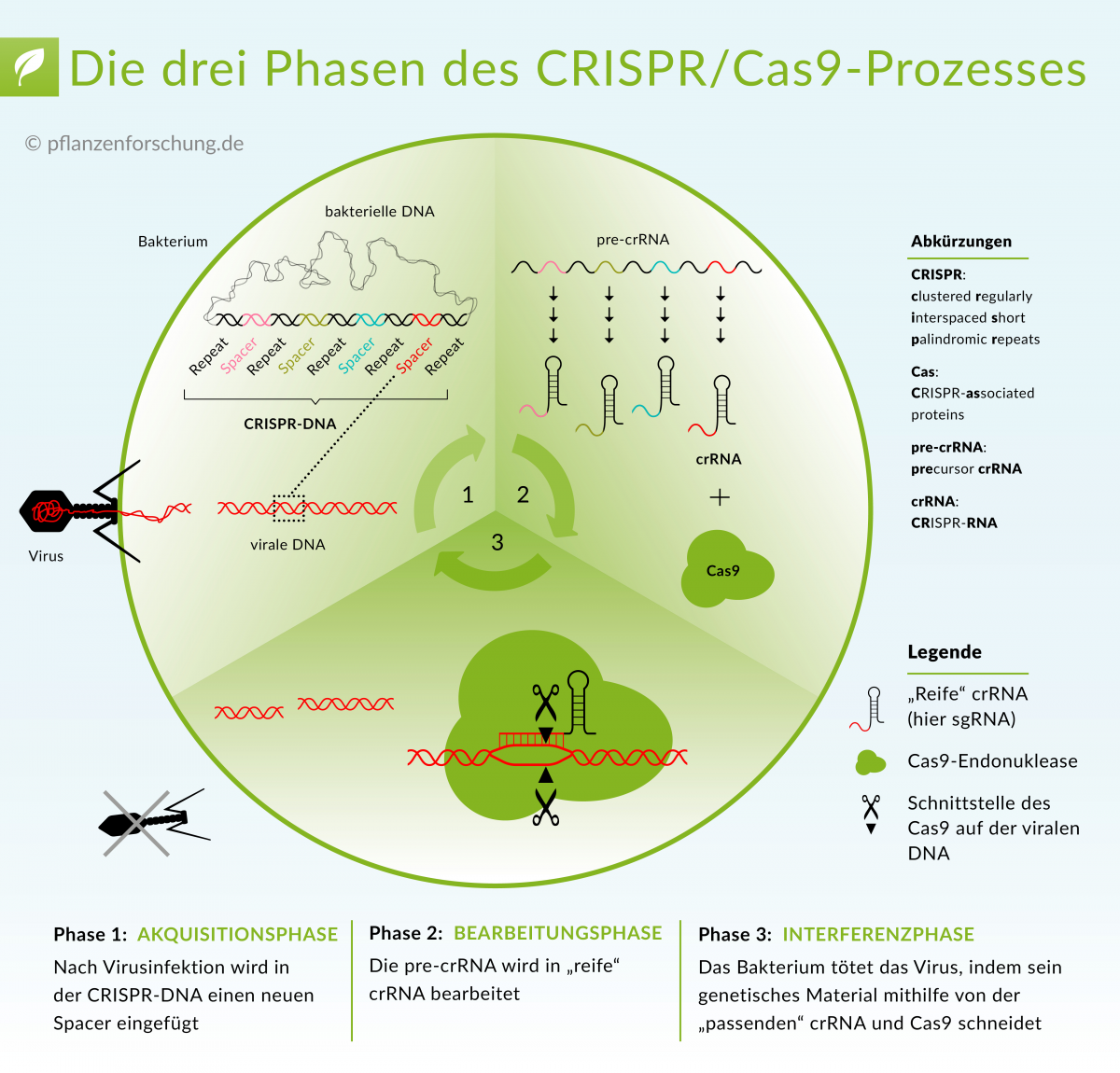
Die Funktionsweise des Typ II CRISPR/Cas-Systems. Der Ablauf des Prozesses erfolgt in drei Phasen: Akquisition eines neuen Distanzstückes, Bearbeitung der pre-crRNA in cr-RNA, Abbau der viralen DNA.
Bildquelle: © Pflanzenforschung.de
Um erfolgreiche Editierungen nachzuweisen, gibt es mehrere Methoden. Die Echtzeit-PCR ist weit verbreitet, ist jedoch begrenzt verlässlich, um einzelne Nukleotid-Variationen zu erkennen. Vielversprechend ist die digitale Tröpfchen-PCR, die solche Veränderungen ebenso gut erkennt wie auch Deletionen einzelner Nukleotide. Wie alle PCR-Methoden ist auch dieser Nachweis jedoch indirekt und zeigt nicht die exakte Veränderung auf Nukleotidebene.
Das gelingt mittels Sequenzierungen. Die Sanger-Sequenzierung der Zielorte weist die Häufigkeit und Art einer Mutation nach. Sie ist jedoch begrenzt tauglich für polyploide Organismen mit heterozygoten oder multiallelischen Mutationen. Aufgrund ihrer schlechten Sensitivität übersieht die Methode auch leicht Editierungen mit niedriger Effizienz. Sequenzierungsmethoden der zweiten und dritten Generation haben diese Limitierungen jedoch überwunden. Sie produzieren zwar enorme Datenmengen, doch diese lassen sich bioinformatisch handhaben und auswerten. Vollgenomsequenzierungen haben bereits bewiesen, dass sich auch seltene Off-target-Mutationen zuverlässig entdecken lassen. Unmöglich ist es aber selbst mit den neuesten Sequenzierungsmethoden, künstliche Editierungen von natürlichen Mutationen zu unterscheiden.
Neue Anwendungen für CRISPR/Cas
CRISPR/Cas kann auch in die Genregulation eingreifen, indem es die Häufigkeit von miRNAs reguliert, die ihrerseits mRNAs regulieren. Ebenso lässt sich Cas9 mit Transkriptionsfaktoren oder epigentischen Modulatoren fusionieren, um transkriptionelle Aktivatoren zu rekrutieren.
Auch das CRISPR/Cas-System selbst wird weiterentwickelt. Neu entdeckte Nukleasen zur sequenzspezifischen Mutation (SDN), modifizierte Cas-Proteine und die Kombination mit DNA-modifizierenden Proteinen sollen die Zielgenauigkeit verbessern. Einige wurden bereits erfolgreich getestet und haben sich selbst bei Pflanzengenotypen mit geringer Regenerationskapazität bewährt.
Chancen und Herausforderungen
Auf neue Pathogene könnte die Pflanzenzüchtung mittels CRISPR/Cas schnell reagieren und in vergleichsweise kurzer Zeit multiresistente Sorten entwickeln. Schwieriger ist es bei abiotischen Stressfaktoren, da diese meist durch komplexe Mechanismen reguliert sind, an denen eine Vielzahl von Genen beteiligt ist.
Hoffnung setzt die Pflanzenforschung auch auf de novo-Domestizierungen: Viele Wildverwandte unserer Kulturpflanzen und Landrassen besitzen noch breite Resistenzen oder sind an schwierige Klimata angepasst. Mittels Genom-Editierung könnten deren Genome so verändert werden, dass sie die wichtigsten Merkmale enthalten, auf denen die Produktivität der heutigen Hochertragssorten beruht.
Quelle:
Cardi, T., et al. (2023): CRISPR/Cas-mediated plant genome editing: outstanding challenges a decade after implementation. In: Trends in Plant Science, 2472 (16. Juni 2023). dio: j.tplants.2023.05.012.
Zum Weiterlesen auf Pflanzenforschung.de:
- Genom-Editierung wird präziser - Künstliche Intelligenz verbessert die Erfolgsrate beim Prime Editing
- TALEN übertrifft Cas9 im Heterochromatin - Das ideale Werkzeug zur Genom-Editierung hängt von der Umgebung ab
Titelbild: Genom-Editierungen ermöglichen Veränderungen an der DNA, die in der Regel nicht von natürlichen Mutationen zu unterscheiden sind. (Bildquelle: © Gerd Altmann / Pixabay)
