„Polyploidization First“
Polyploidisierung ging der Domestikation voraus

Emmer (Triticum dicoccum) ist eine der ältesten kultivierten Nutzpflanzen. Vermutlich gingen die ersten Kultursorten aus polyploiden Wildtypen hervor. (Bildquelle: © Rasbak/ wikimedia.org/ CC BY-SA 3.0)
Die Frage, ob der Domestizierung einst eine Phase mehrerer Polyploidisierungsereignisse vorausging oder umgekehrt, beschäftigt Experten seit Jahrzehnten. Anhand der Sequenzierungsdaten von über 3.000 Wild- und Nutzpflanzen steht die Antwort nun vorläufig fest. Bei ihrer Suche stießen die Forscher darüber hinaus auf ein interessantes Grundmuster in Bezug auf das Auftreten von Polyploidie.
Die Parallelen zur altbekannten Frage, was zuerst da war, die Henne oder das Ei, sind unübersehbar. Doch geht es hier weder um Hühner noch um Eier, sondern um eine Frage, die die Forschung seit Jahrzehnten beschäftigt und bis zu den Anfängen der Domestikationsgeschichte reicht: Ging der Domestizierung einstiger Wildpflanzen eine Reihe von Polyploidisierungsereignissen voraus oder geschah dies in umgekehrter Reihenfolge? Neueste Zahlen sprechen dafür, dass die ersten Landwirte und Pflanzenzüchter vor rund 13.000 Jahren überwiegend mit Pflanzen hantierten, die ihren Chromosomensatz zuvor vervielfacht hatten (Genomduplikation), also bereits polyploid waren.
Wie hängt beides zusammen?
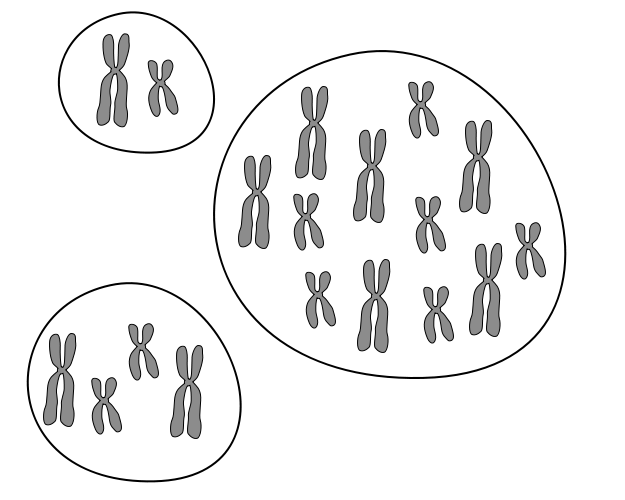
Vereinfacht dargestellt sind hier ein haploider (oben links), ein diploider (unten links) und ein polyploider bzw. hexaploider Zellkern am Beispiel eines Chromosomensatzes mit zwei Chromosomen zu sehen.
Bildquelle: © Talos/ wikimedia.org/ CC BY-SA 3.0
Dass Domestikation und Polyploidie in einem Atemzug genannt werden, liegt daran, dass viele höhere Pflanzen (Kormophyten), insbesondere wichtige Kulturarten bzw. -sorten polyploid sind. Sie besitzen mehr als zwei Chromosomensätze. Zu ihnen zählen u. a. Weizen (Triticum aestivum) und Hafer (Avena), Baumwolle (Gossypium), Zuckerrohr (Saccharum officinarum) und Kartoffel (Solanum tuberosum), verschiedene Obst- und Gemüsearten und -sorten, bis hin zu Farnen und Orchideen (Orchidaceae).
Größerer Genpool zahlt sich aus
Dass dem so ist, verwundert nicht. Schließlich sind die Unterschiede mitunter selbst für Laien mit bloßem Auge erkennbar, weshalb ihnen vermutlich auch unsere Vorfahren bei der Selektion den Vorzug gaben. So sind polyploide Pflanzen häufig größer, kräftiger und vitaler als ihre diploiden Verwandten, tragen größere Samen, kommen besser mit verschiedenen, teils widrigen Umweltbedingungen zurecht und halten auch einem höheren Schädlingsdruck stand. Es ist dabei nicht nur das Mehr eines Genprodukts durch die Vervielfachung der Genome, sondern auch der größere und über die Zeit diversere Genpool, aus dem sie schöpfen, der sie anpassungsfähiger macht. Dass oben stets auf Art und Sorte Bezug genommen wurde, liegt daran, dass innerhalb einer Art, z. B. bei der Kartoffel, durchaus diploide wie auch polyploide Sorten existieren.
Bis zu den Anfängen
Obwohl Experten seit langem nahezu sicher sind, dass von Anfang an ein enger Zusammenhang zwischen der Pflanzendomestizierung und der Polyploidie bestanden haben musste, fehlten für ein vollständiges Bild wichtige Details. Diese zu finden, war das Anliegen der Molekularbiologin Ayelet Salman Minkov. Gemeinsam mit ihren Mitstreitern verglich sie deshalb die Sequenzierungsdaten von 297 Kulturlinien mit denen von 2.836 wilden Verwandten. Eine Einschränkung, die die Aussagekraft in Bezug auf die Pflanzenevolution im Allgemeinen zwar reduzierte, dennoch zu neuen Erkenntnissen in einem wichtigen Teilbereich führte. Minkovs Ziel war es, die stammesgeschichtliche Entwicklung (Phylogenese) zurückzuverfolgen, um herauszufinden, ob die Mehrzahl der Polyploidisierungsereignisse vor den entscheidenden Domestikationsschritten stattgefunden hatte oder erst im Zuge dessen.
Der Silber-Brandschopf (Celosia argentea), auch Hahnenkamm genannt, gehört zu den Rekordhalten in Bezug auf die Zahl der Chromosomensätze im Zellkern. Sie liegt bei 12, weshalb er auch als dodecaploid bezeichnet wird.
Bildquelle: © Stefan Dressler/ wikimedia.org / CC BY-SA 2.5
Stammbaum mit Zusatzinfos
Vereinfacht gesagt basierte ihr Ansatz darauf, die Zahl der Chromosomen zu vergleichen, um signifikante Veränderungen im Laufe der Evolution bzw. Domestizierung aufzuspüren und den wesentlichen Domestikationsschritten gegenüberzustellen. Am Ende galt es, insgesamt 474 Polyploidisierungsereignisse mit 197 Domestikationsschritten in eine chronologische Reihenfolge zu bringen. Man kann sich das Ergebnis wie einen Stammbaum vorstellen, dessen Linien nicht mehr nur die Verwandtschaftsverhältnisse zeigten, sondern auch, ob und wann sich der Chromosomensatz vervielfacht hatte.
Wer hatte recht?
Im Großen und Ganzen bestätigten die Zahlen zunächst den oben skizzierten Zusammenhang. Während insgesamt 30 % der untersuchten Nutzpflanzen polyploid waren, waren es bei den wilden Verwandten 24 %. In Bezug auf die drängende Frage, welcher der beiden Prozesse nun dem anderen vorausgegangen war, steht nun fest, dass die Mehrheit der 474 Polyploidisierungsereignisse vor der Domestizierung stattgefunden hatte. Genau genommen in 448 Fällen, also 95 %. Die lang ersehnte Bestätigung für die Befürworter der „Polyploidization First“-Hypothese.
Auffällige Häufigkeitsverteilung
Bemerkenswert war darüber hinaus aber, dass die Polyploidiehäufigkeit keineswegs gleichmäßig verteilt war. So waren Nutzpflanzen, deren Wurzeln oder Wurzelknollen wir nutzen, häufiger betroffen, ebenso wie Textilpflanzen und Getreide. Weniger hingegen Obst, Gemüse und Hülsenfrüchte. Es sollte sich dabei um ein Muster handeln, wie die Forscher herausfanden.
Mit einem Anteil von 41 % waren in der Regel weitaus mehr Einkeimblättrige (Monokotyledonen) polyploid als Zweikeimblättrige (Dikotylen) mit 18 %. Auch in puncto Lebenszyklus und Wuchsform zeigten sich Unterschiede. So waren unter den Mehrjährigen mit 26 % mehr Polyploide zu finden als unter den Einjährigen mit 21 %, unter den krautigen Pflanzen mit 29 % mehr als unter den Gehölzen mit 16 %.
Das Geheimnis hinter dem Muster
Mit Verweis darauf, dass die meisten Wurzelgemüse mehrjährig seien, Obst- und Nussbäume per definitionem zu den Gehölzen zählten, Hülsenfrüchte hingegen meist einjährig seien, die Getreide wiederum aus den Süßgräsern (Poaceae) hervorgingen, bei denen Polyploidie keine Seltenheit sei, konnten die Forscher das Geheimnis jedoch schnell lüften. Sie waren damit zwar nicht die Ersten, die auf dieses Muster gestoßen sind, jedoch datiert der einzige Verweis, der in der Studie zitiert wird, auf das Jahr 1938.
Somit liegt auf der Hand, dass die im Nature-Magazin veröffentlichte Studie zur Klärung einer Frage beiträgt, die die Fachcommunity lange beschäftigte, ebenso zum besseren Verständnis der Bedeutung der Polyploidie in einem Teilbereich der Pflanzenevolution. Doch wie sieht es darüber hinaus aus? Die Antwort findet sich im parallel erschienenen Kommentar von Zhou Fang und Peter L. Morrell.
Aus dem Schatten der Diploiden

Herbstzeitlose (Colchicum automnale) im Frühjahr. Alle ihre Teile enthalten das Alkaloid Colchicin, das zur künstlichen Erzeugung von Polyploidie eingesetzt wird. Immer wieder kommt es zu Vergiftungsfällen, da ihre Blätter mit denen des beliebten Bärlauchs verwechselt werden.
Bildquelle: © Dr. Nachtigaller/ wikimedia.org/ CC0
Beide sind überzeugt, dass die Studie ein wichtiges und überfälliges Schlaglicht auf die Gruppe der polyploiden Nutzpflanzen werfe, denen im Vergleich zu ihren diploiden Verwandten nach wie vor zu wenig Aufmerksamkeit vonseiten der Forschung geschenkt werde. Zwar räumen sie ein, dass der Umgang mit polyploiden Genomen in der Tat kniffliger sei als mit diploiden, jedoch sehen sie die Technologie so weit fortgeschritten, dass auch dieses Problem bald Vergangenheit sein dürfte.
Schwierigkeiten im Umgang mit polyploiden Genomen
Eine besondere Hürde sei nach wie vor die Sequenzierung polyploider Genome. Problematisch sei dabei die Kürze der sequenzierten Fragmente, auch „Reads“ genannt, so die Forscher. Sie erschwere es, diese bei der Resequenzierung anhand eines Referenzgenoms wieder seinem Ursprungsort zuzuordnen, da polyploide Genome aufgrund der vervielfachten Chromosomen zum einen größer seien als diploide und sich die besagten Chromosomen darüber hinaus ähnelten.
Hoffnung setzen Fang und Morrell daher in die Weiterentwicklung der sogenannten „Long-Read-Sequenzierungstechnologien“. Hintergrund ist, dass durch die Nutzung von längeren „Reads“auch die Wahrscheinlichkeit steige, diese ihrem richtigen Ursprungsort zuzuordnen und Verwechslungen vorzubeugen. So wie einst auch unsere Vorfahren auf polyploide Pflanzenarten setzten, ließe sich die Polyploidie auf diese Weise noch besser und zielsicherer bei der Entwicklung neuer und verbesserter Nutzpflanzen einsetzen als bisher.
Nicht erst seit gestern wird die künstliche Polyploidieerzeugung, u. a. mithilfe einer giftigen Verbindung (Colchicin) aus der Herbstzeitlosen (Colchicum autumnale), eingesetzt. Eine Weiterentwicklung der Technologie und des Wissens in diese Richtung würde diesen Ansatz einen deutlichen Schritt nach vorn bringen.
Quellen:
- Salman-Minkov, A. et al. (2016): Whole-genome duplication as a key factor in crop domestication. In: Nature Plants, Vol. 2 (16115), (1. August 2016), doi:10.1038/nplants.2016.115
- Fang, Z., Morell, P. (2016): Polyploidy boosts domestication. In: Nature Plants, Vol. 2 (16116), doi:10.1038/nplants.2016.116
Weiterlesen auf Pflanzenforschung.de:
Titelbild: Emmer (Triticum dicoccum) ist eine der ältesten kultivierten Nutzpflanzen. Vermutlich gingen die ersten Kultursorten aus polyploiden Wildtypen hervor. (Bildquelle: © Rasbak/ wikimedia.org/ CC BY-SA 3.0)
